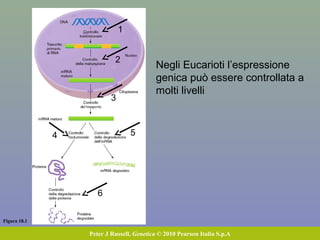
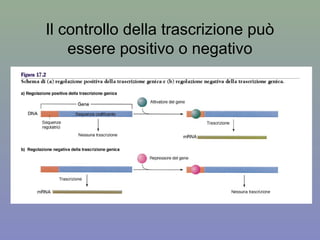
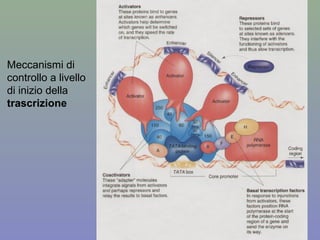
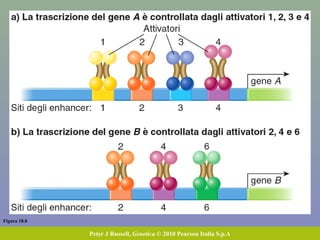
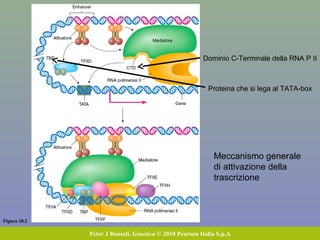
Il documento discute la regolazione dell'espressione genica negli eucarioti a vari livelli, inclusi i meccanismi di trascrizione, regolazione post-trascrizionale tramite microRNA e si occupa delle modifiche epigenetiche come la metilazione del DNA. Esamina inoltre il ruolo dei fattori di trascrizione, attivatori, repressori e l'importanza delle sequenze regolatrici nel controllo dell'attivazione genica. Infine, si analizzano le differenze nell'espressione genica tra le popolazioni umane e quelle di altri primati in relazione a tratti come il linguaggio.