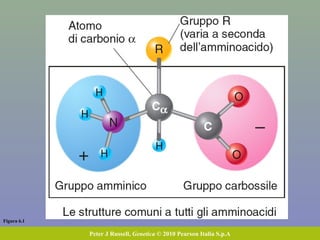
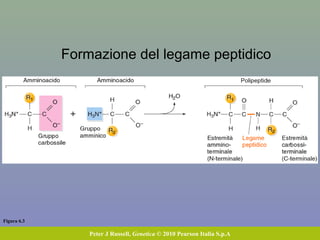
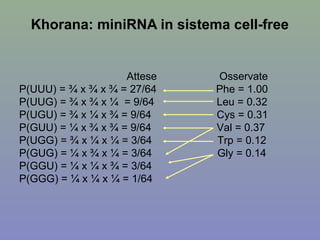
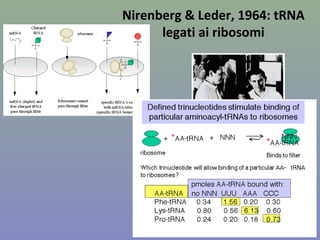
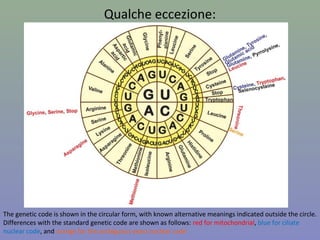
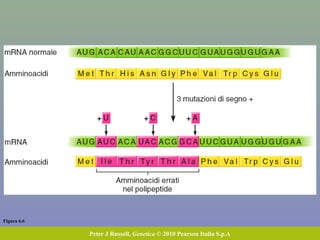
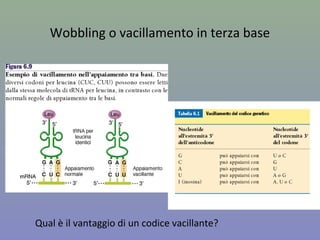
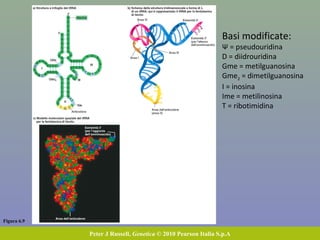
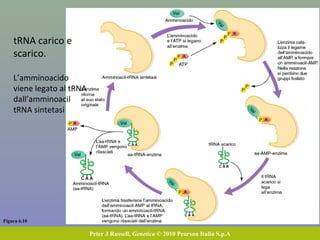
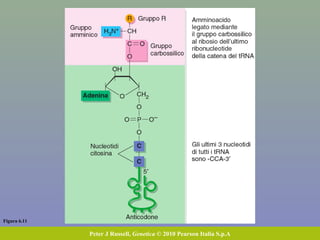
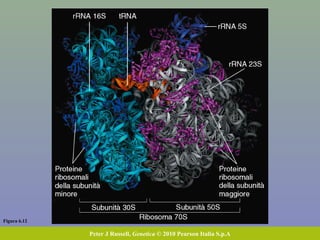
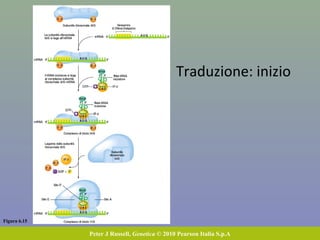
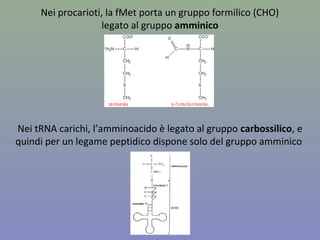
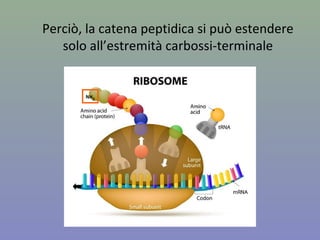
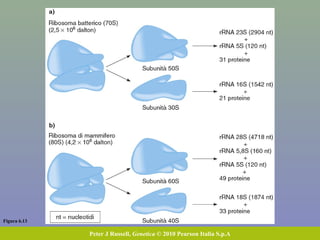
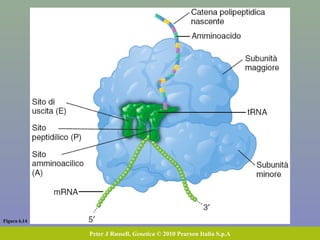
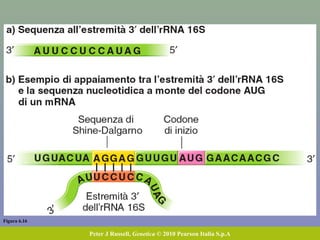
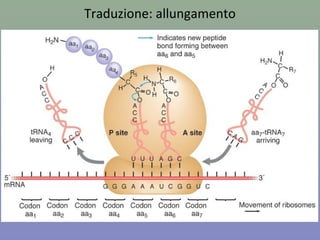
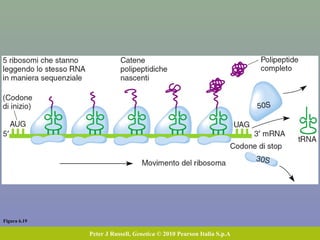
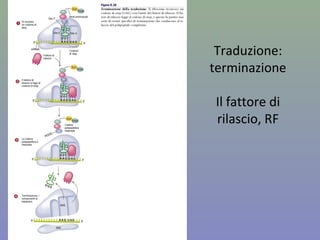
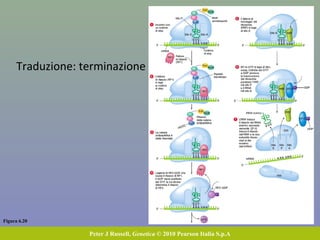
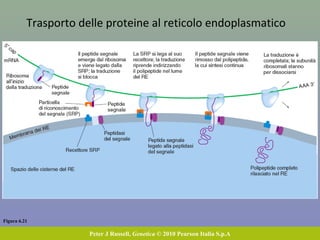
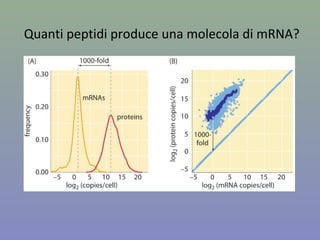
Il capitolo tratta della traduzione e dell'espressione genica, spiegando come le proteine sono sintetizzate attraverso il legame tra il DNA e gli amminoacidi tramite l'RNA. Viene discusso il codice genetico, le sue caratteristiche e le differenze tra procarioti ed eucarioti nel processo di traduzione. Infine, si evidenziano i ruoli dei vari RNA e le sequenze che segnano l'inizio e la fine della traduzione.