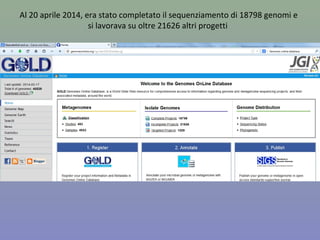
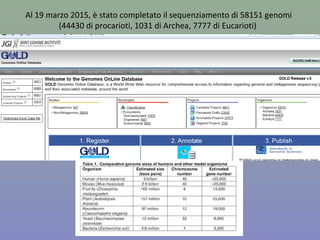
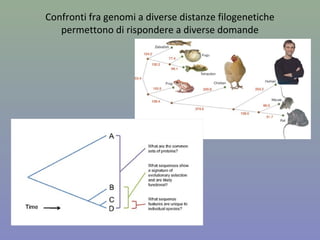
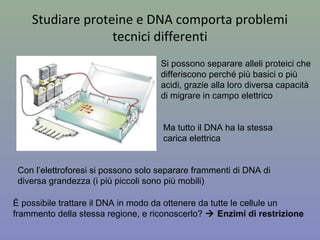
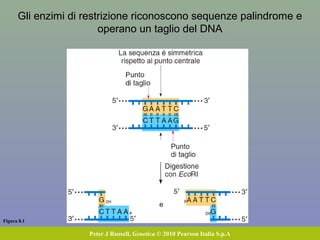
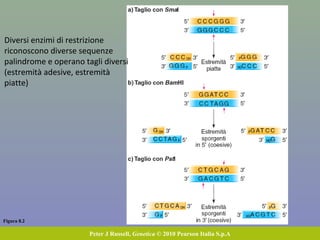
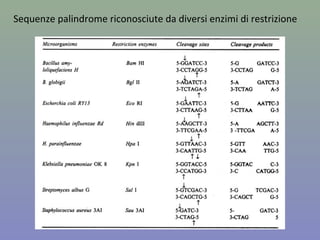
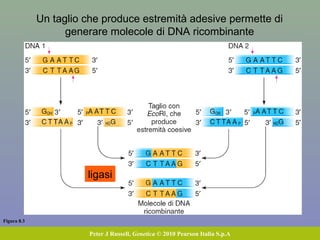
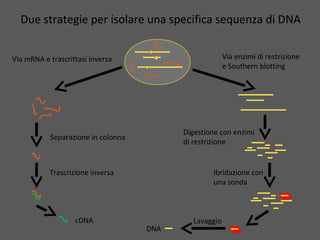
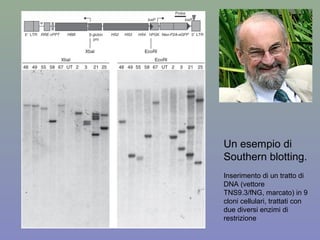
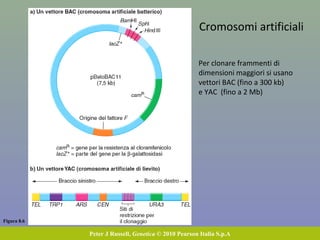
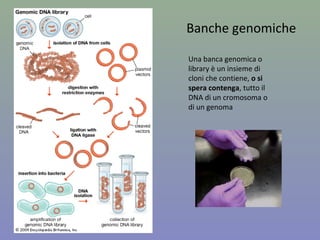
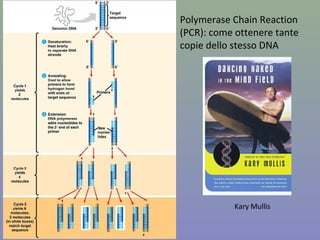
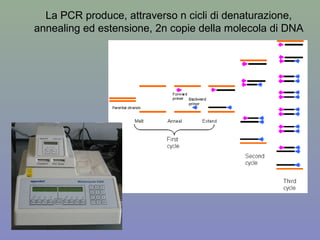
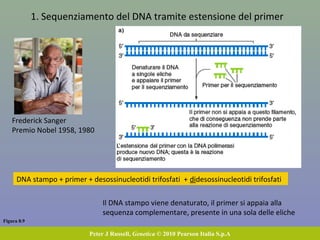
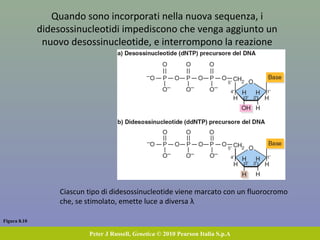
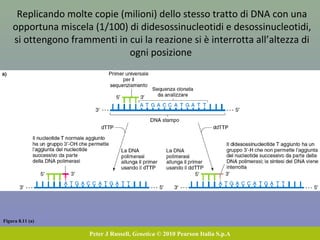
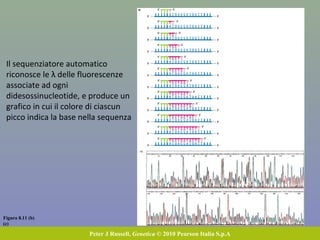
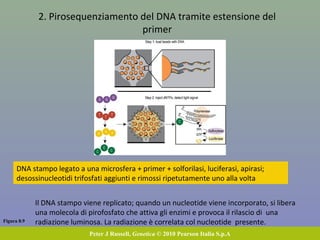
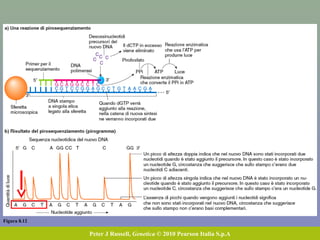
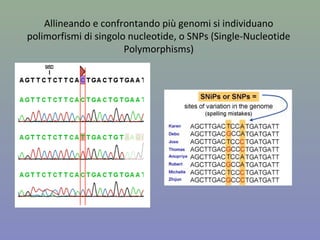
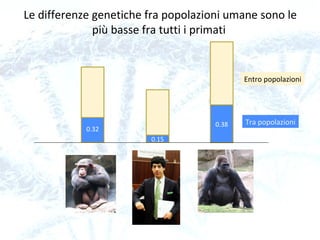
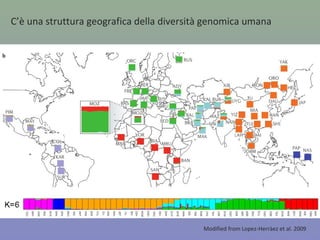
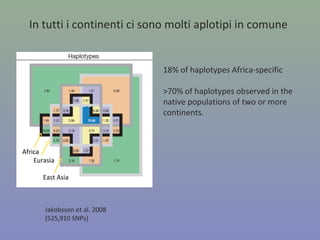
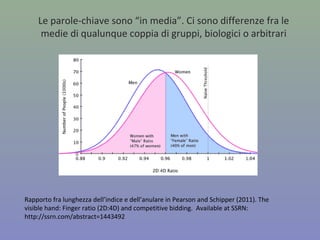
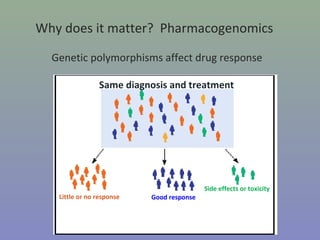
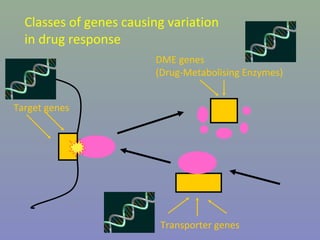
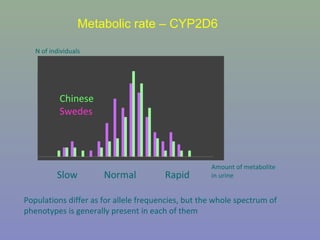
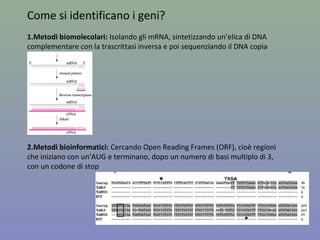
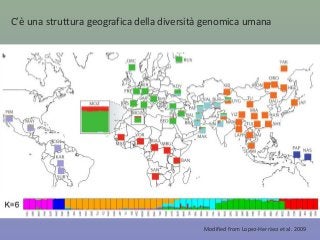
Il capitolo 8 tratta la genomica, inclusa la mappatura e il sequenziamento del DNA. Si esplorano le tecniche di isolamento, clonaggio e sequenziamento del DNA, come l'uso di enzimi di restrizione e la PCR, fino al sequenziamento di Sanger e al next-generation sequencing. Inoltre, si discute la variabilità genetica umana, evidenziando che la differenza media tra individui è dello 0,1%, la più bassa tra i primati.