More Related Content
PDF
PDF
PDF
PDF
Disentanglement Survey:Can You Explain How Much Are Generative models Disenta... PDF
SSII2021 [OS2-02] 深層学習におけるデータ拡張の原理と最新動向 PPTX
勾配ブースティングの基礎と最新の動向 (MIRU2020 Tutorial) PPTX
【論文紹介】How Powerful are Graph Neural Networks? PPTX
【DL輪読会】ViT + Self Supervised Learningまとめ What's hot
PPTX
【DL輪読会】Efficiently Modeling Long Sequences with Structured State Spaces PDF
【基調講演】『深層学習の原理の理解に向けた理論の試み』 今泉 允聡(東大) PDF
最近のKaggleに学ぶテーブルデータの特徴量エンジニアリング PDF
PDF
cvpaper.challenge 研究効率化 Tips PPTX
PPTX
猫でも分かるVariational AutoEncoder PDF
PDF
PDF
PDF
BlackBox モデルの説明性・解釈性技術の実装 PDF
実践多クラス分類 Kaggle Ottoから学んだこと PDF
ブレインパッドにおける機械学習プロジェクトの進め方 PDF
PDF
[DL輪読会]Wasserstein GAN/Towards Principled Methods for Training Generative Adv... PPTX
【DL輪読会】時系列予測 Transfomers の精度向上手法 PDF
PDF
PDF
PDF
トピックモデルの評価指標 Perplexity とは何なのか? Viewers also liked
PDF
「深層学習」勉強会LT資料 "Chainer使ってみた" PPTX
PPTX
PDF
PDF
科学と機械学習のあいだ:変量の設計・変換・選択・交互作用・線形性 PPTX
PPTX
面倒くさいこと考えたくないあなたへ〜Tpotと機械学習〜 PDF
GBDTを使ったfeature transformationの適用例 PDF
Randomforestで高次元の変数重要度を見る #japanr LT PPTX
PPTX
PDF
PDF
PDF
ZIP
PDF
「はじめてでもわかる RandomForest 入門-集団学習による分類・予測 -」 -第7回データマイニング+WEB勉強会@東京 Similar to 不均衡データのクラス分類
PPTX
SMOTE resampling method slides 02-19-2018 PDF
A systematic study of the class imbalance problem in convolutional neural net... PDF
PPTX
PPTX
PDF
PPTX
Long-Tailed Classificationの最新動向について PDF
PDF
PDF
PDF
PDF
PDF
Introduction to ensemble methods for beginners PDF
PDF
Jubatusにおける大規模分散オンライン機械学習 PPTX
0610 TECH & BRIDGE MEETING PPTX
Feature Selection with R / in JP PPTX
DLLab 異常検知ナイト 資料 20180214 PDF
レコメンドアルゴリズムの基本と周辺知識と実装方法 PDF
More from Shintaro Fukushima
PDF
20230216_Python機械学習プログラミング.pdf PDF
PDF
Materials Informatics and Python PDF
PDF
最近のRのランダムフォレストパッケージ -ranger/Rborist- PDF
Why dont you_create_new_spark_jl PDF
PDF
PDF
PDF
PDF
PDF
data.tableパッケージで大規模データをサクッと処理する PDF
アクションマイニングを用いた最適なアクションの導出 PDF
PDF
PDF
PDF
統計解析言語Rにおける大規模データ管理のためのboost.interprocessの活用 PDF
PDF
PDF
不均衡データのクラス分類
- 1.
- 2.
アジェンダ
自己紹介
クラス分類
不均衡データ
不均衡データへの対処方法
- 3.
- 4.
- 5.
- 6.
- 7.
クラス分類を行うための手法は
数多く提案されている.
決定木
ナイーブベイズ
サポートベクタマシン
ブースティング
ランダムフォレスト etc.
- 8.
- 9.
- 10.
- 11.
- 12.
- 13.
> library(kernlab)
> #データの読み込み(データは"../data/"ディレクトリに置いておく)
> abalone <- read.csv("../data/abalone.data", header=FALSE)
> # 19番目のクラスを正例に,それ以外のクラスを負例とする
> label <- abalone[, 9]
> label[label==19] <- "positive"
> label[label!="positive"] <- "negative"
> label <- factor(label)
> table(label)
label
negative positive
4145 32
正例32サンプル,
負例4145サンプルのデータ
- 14.
> set.seed(123)
> #クロスバリデーションの実行(多項式カーネルを用い,次数は2とする)
> idx <- sample(1:10, nrow(abalone), replace=TRUE)
> for (i in 1:10) {
+ is.test <- idx == i
+ abalone.train <- abalone[!is.test, ]
+ abalone.test <- abalone[is.test, -9]
+ fit.ksvm <- ksvm(label ~., data=abalone.train, kernel="polydot",
kpar=list(degree=2))
+ pred[is.test] <- as.character(predict(fit.ksvm, abalone.test))
+}
> # 予測結果の集計
> table(pred)
pred
negative 全てを負例と判別!!
4177
- 15.
- 16.
- 17.
①正例を誤答したときの
ペナルティを重くする
(cost-sensitive learning)
②正例と負例のサンプル数を
調整する
- 18.
①正例を誤答したときの
ペナルティを重くする
(cost-sensitive learning)
SVMでは,
ksvm関数(kernlabパッケージ)の
class.weights引数に指定
- 19.
> label.table <- table(label)
> # 正例の重み(負例と正例のサンプル数の比とする)
> weight.positive <- as.numeric(label.table[1]/label.table[2])
> # 10-fold クロスバリデーションの実行
> for (i in 1:10) { 正例と負例の
+ is.test <- idx == i
+ abalone.train <- abalone[!is.test, ]
サンプル数に反比例した
+ abalone.test <- abalone[is.test, -9] ペナルティの重みを指定
+ fit.ksvm <- ksvm(label ~., data=abalone.train,
+ class.weights=c("positive"=weight.positive,
+ "negative"=1),
+ kernel="polydot", kapr=list(degree=2))
+ pred[is.test] <- as.character(predict(fit.ksvm, abalone.test))
+ }
> table(label, pred) 何も工夫しないよりは
pred
label negative positive 良くなったが,まだまだ
negative 3118 1027 (モデルパラメータの
positive 19 13 チューニングの余地もまだまだあり)
- 20.
②正例と負例のサンプル数を
調整する
オーバーサンプリング
→正例を増やす
アンダーサンプリング
→負例を減らす
両方
- 21.
- 22.
- 23.
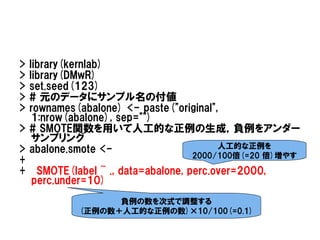
> library(kernlab)
> library(DMwR)
>set.seed(123)
> # 元のデータにサンプル名の付値
> rownames(abalone) <- paste("original",
1:nrow(abalone), sep="")
> # SMOTE関数を用いて人工的な正例の生成,負例をアンダー
サンプリング
> abalone.smote <- 人工的な正例を
2000/100倍(=20 倍)増やす
+
+ SMOTE(label ~ ., data=abalone, perc.over=2000,
perc.under=10)
負例の数を次式で調整する
(正例の数+人工的な正例の数)×10/100(=0.1)
- 24.
>idx <- sample(1:10,nrow(abalone.smote), replace=T)
>pred <- rep(NA, nrow(abalone.smote))
># 10-fold クロスバリデーションの実行
>for (i in 1:10) {
+ is.test <- idx == i
+ abalone.train <- abalone.smote[!is.test, ]
+ abalone.test <- abalone.smote[is.test, -9]
+ fit.ksvm <- ksvm(label ~., data=abalone.train,
kernel="polydot", kpar=list(degree=2))
+ pred[is.test] <- predict(fit.ksvm, abalone.test)
+}
- 25.
> # SMOTEでの補間点も含むデータに対する分割表
>table(abalone.smote$label, pred)
negative positive
negative 19 13
positive 1 351
> # 元々のデータに対する分割表
> is.original <- rownames(abalone.smote) %in%
+ rownames(abalone)
> table(abalone.smote[is.original, "label"],
+ pred[is.original])
negative positive
negative 19 13 まだまだチューニングの
positive 0 32 余地はあるが,
それでも精度はかなり
向上
- 26.
- 27.
- 28.
- 29.
- 30.
Weighted RandomForest
正例,負例をそれぞれ誤って分類する際のペ
ナルティを以下の2箇所で考慮する.
Gini係数を評価基準として決定木の枝を作成する
とき
予測ラベルを決定する際に重み付き多数決を取
るとき
Balanced Random Forest
各ツリーを構築する際, 正例のデータ数と同じ
だけ負例のデータをサンプリングして学習する.
- 31.
- 32.
引数classwtを調整しても
あまり効果はない?
randomForestパッケージの管理者によると・・・
(http://bit.ly/xJ2mUJ)
現在のclasswtオプションはパッケージが開発された当初から存在しているが,
公式のfortranのコード(バージョン4以降)の実装とは異なる.
クラスの重みを考慮するのは,ノード分割時にGini係数を算出する際のみである.
我々は,クラスの重みをGini係数の算出においてのみ用いても,極端に不均衡
なデータ(例えば1:100やそれ以上)に対してはあまり役に立たないことが分かっ
た.そこで,Breiman教授はクラスの重みを考慮する新しい方法を考案した.こ
の方法は新しいfortranコードに実装されている.
現在のパッケージのclasswtにクラスの重みを指定しても,我々が望んでいた結
果が過去に得られなかったことだけは付記しておく.
- 33.
まとめ
不均衡データ="各クラスに属するサンプ
ル数に偏りがあるデータ"
不均衡データに対するクラス分類におい
てはいろいろと工夫が必要な場合がある
対応方法としては,正例の誤判別ペナル
ティを調整する方法とサンプリングを工夫
する方法が代表的
個々の問題に応じていろいろと試すべし
- 34.












![> library(kernlab)
> # データの読み込み(データは"../data/"ディレクトリに置いておく)
> abalone <- read.csv("../data/abalone.data", header=FALSE)
> # 19番目のクラスを正例に,それ以外のクラスを負例とする
> label <- abalone[, 9]
> label[label==19] <- "positive"
> label[label!="positive"] <- "negative"
> label <- factor(label)
> table(label)
label
negative positive
4145 32
正例32サンプル,
負例4145サンプルのデータ](https://image.slidesharecdn.com/random-120128040703-phpapp02/85/slide-13-320.jpg)
![> set.seed(123)
> # クロスバリデーションの実行(多項式カーネルを用い,次数は2とする)
> idx <- sample(1:10, nrow(abalone), replace=TRUE)
> for (i in 1:10) {
+ is.test <- idx == i
+ abalone.train <- abalone[!is.test, ]
+ abalone.test <- abalone[is.test, -9]
+ fit.ksvm <- ksvm(label ~., data=abalone.train, kernel="polydot",
kpar=list(degree=2))
+ pred[is.test] <- as.character(predict(fit.ksvm, abalone.test))
+}
> # 予測結果の集計
> table(pred)
pred
negative 全てを負例と判別!!
4177](https://image.slidesharecdn.com/random-120128040703-phpapp02/85/slide-14-320.jpg)




![> label.table <- table(label)
> # 正例の重み(負例と正例のサンプル数の比とする)
> weight.positive <- as.numeric(label.table[1]/label.table[2])
> # 10-fold クロスバリデーションの実行
> for (i in 1:10) { 正例と負例の
+ is.test <- idx == i
+ abalone.train <- abalone[!is.test, ]
サンプル数に反比例した
+ abalone.test <- abalone[is.test, -9] ペナルティの重みを指定
+ fit.ksvm <- ksvm(label ~., data=abalone.train,
+ class.weights=c("positive"=weight.positive,
+ "negative"=1),
+ kernel="polydot", kapr=list(degree=2))
+ pred[is.test] <- as.character(predict(fit.ksvm, abalone.test))
+ }
> table(label, pred) 何も工夫しないよりは
pred
label negative positive 良くなったが,まだまだ
negative 3118 1027 (モデルパラメータの
positive 19 13 チューニングの余地もまだまだあり)](https://image.slidesharecdn.com/random-120128040703-phpapp02/85/slide-19-320.jpg)



![>idx <- sample(1:10, nrow(abalone.smote), replace=T)
>pred <- rep(NA, nrow(abalone.smote))
># 10-fold クロスバリデーションの実行
>for (i in 1:10) {
+ is.test <- idx == i
+ abalone.train <- abalone.smote[!is.test, ]
+ abalone.test <- abalone.smote[is.test, -9]
+ fit.ksvm <- ksvm(label ~., data=abalone.train,
kernel="polydot", kpar=list(degree=2))
+ pred[is.test] <- predict(fit.ksvm, abalone.test)
+}](https://image.slidesharecdn.com/random-120128040703-phpapp02/85/slide-24-320.jpg)
![> # SMOTEでの補間点も含むデータに対する分割表
> table(abalone.smote$label, pred)
negative positive
negative 19 13
positive 1 351
> # 元々のデータに対する分割表
> is.original <- rownames(abalone.smote) %in%
+ rownames(abalone)
> table(abalone.smote[is.original, "label"],
+ pred[is.original])
negative positive
negative 19 13 まだまだチューニングの
positive 0 32 余地はあるが,
それでも精度はかなり
向上](https://image.slidesharecdn.com/random-120128040703-phpapp02/85/slide-25-320.jpg)








