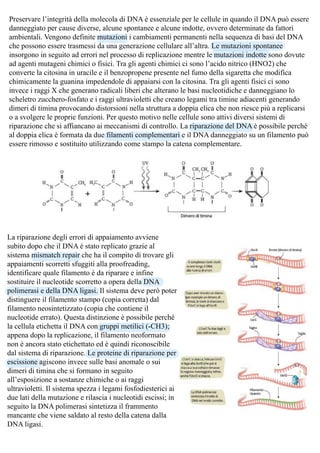
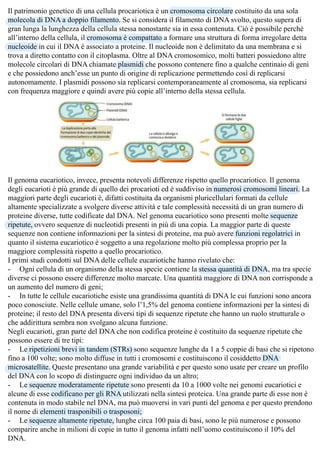
La replicazione del DNA è un processo semiconservativo che genera due copie identiche di DNA utilizzando filamenti parentali come stampo, richiedendo enzimi e nucleotidi. Il processo si svolge in due fasi: la desalinizzazione della doppia elica tramite elicasi e la sintesi dei nuovi filamenti da parte della DNA polimerasi, con meccanismi di controllo per garantire precisione. Differenze tra DNA procariotico ed eucariotico includono la struttura dei cromosomi e la complessità del regolamento genetico, con sequenze di DNA ripetute e cromatina altamente impacchettata per mantenere l'integrità genetica.