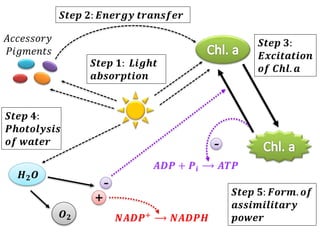
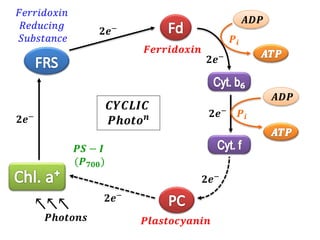
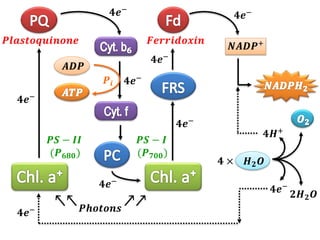
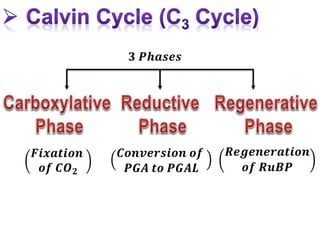
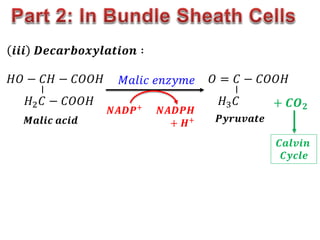
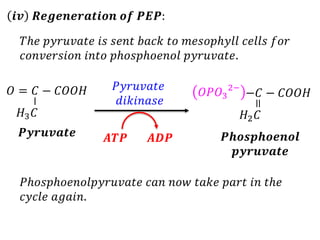
The document provides a comprehensive overview of photosynthesis, detailing the structures involved such as chloroplasts, thylakoids, and various pigments including chlorophyll and carotenoids. It describes the light reactions and dark reactions in photosynthesis, specifically the processes occurring in Photosystems I and II, and how energy from light is captured and converted into chemical energy. Additionally, it covers the Calvin cycle and the C4 photosynthetic pathway, highlighting the differences between mesophyll and bundle sheath cells in C4 plants.
















![𝐶 = 𝑂
𝐻𝐶 − 𝑂𝐻
𝐻𝐶 − 𝑂𝐻
𝐻2𝐶 − 𝑶𝑷𝑶𝟑
𝟐−
𝟏
𝟐
𝟑
𝟒
𝟓
𝐻2𝐶 − 𝑶𝑷𝑶𝟑
𝟐−
𝑹𝒊𝒃𝒖𝒍𝒐𝒔𝒆 𝟏, 𝟓 −
𝒃𝒊𝒔𝒑𝒉𝒐𝒔𝒑𝒉𝒂𝒕𝒆
(𝑹𝒖𝑩𝑷)
6 ×
𝑹𝒖𝑩𝒊𝒔𝑪𝑶
𝐻𝐶 − 𝑂𝐻
𝐶𝑂𝑂𝐻
𝐻2𝐶 − 𝑶𝑷𝑶𝟑
𝟐−
𝟏
𝟐
𝟑
12 ×
𝟑 − 𝑷𝒉𝒐𝒔𝒑𝒉𝒐 −
𝒈𝒍𝒚𝒄𝒆𝒓𝒊𝒄 𝒂𝒄𝒊𝒅
(𝟑 − 𝑷𝑮𝑨)
[𝟔 × 𝟓 𝑪 = 𝟑𝟎 𝑪] [𝟏𝟐 × 𝟑 𝑪 = 𝟑𝟔 𝑪]
+ 𝟔 𝑪𝑶𝟐
+ 𝟔 𝑯𝟐𝑶
𝑅𝑖𝑏𝑢𝑙𝑜𝑠𝑒
𝐵𝑖𝑠𝑝ℎ𝑜𝑠𝑝ℎ𝑎𝑡𝑒
𝐶𝑎𝑟𝑏𝑜𝑥𝑦𝑙𝑎𝑠𝑒
𝑂𝑥𝑖𝑑𝑎𝑠𝑒](https://image.slidesharecdn.com/photosynthesis-copy-211007115149/85/Photosynthesis-25-320.jpg)
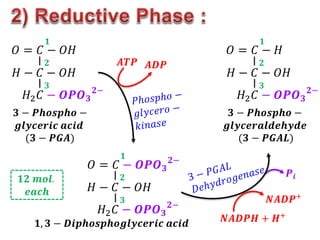
![𝑇ℎ𝑒 12 𝑃𝐺𝐴𝐿 𝑚𝑜𝑙𝑒𝑐𝑢𝑙𝑒𝑠 𝑐𝑎𝑛 𝑏𝑒 𝑢𝑡𝑖𝑙𝑖𝑠𝑒𝑑 𝑖𝑛 4 𝑤𝑎𝑦𝑠:
5 × [𝑷𝑮𝑨𝑳] 5 × [𝑫𝑯𝑨𝑷]
𝑇𝑟𝑖𝑜𝑠𝑒
𝑃ℎ𝑜𝑠𝑝ℎ𝑎𝑡𝑒
𝐼𝑠𝑜𝑚𝑒𝑟𝑎𝑠𝑒 ②
③
[𝟓 × 𝟑 𝑪 = 𝟏𝟓 𝑪] [𝟓 × 𝟑 𝑪 = 𝟏𝟓 𝑪]
[𝟓 × 𝑷] [𝟓 × 𝑷]
3 [𝑷𝑮𝑨𝑳] 3 [𝑫𝑯𝑨𝑷]
[𝟑 × 𝟑 𝑪 = 𝟗 𝑪] [𝟑 × 𝟑 𝑪 = 𝟗 𝑪]
+
[𝟑 × 𝑷] [𝟑 × 𝑷]
𝐴𝑙𝑑𝑜𝑙𝑎𝑠𝑒
3
𝑭𝒓𝒖𝒄𝒕𝒐𝒔𝒆
𝟏, 𝟔 − 𝒅𝒊𝒑𝒉𝒐𝒔.
[𝟑 × 𝟔 𝑪 = 𝟏𝟖 𝑪]
[𝟑 × 𝟐 𝑷]
𝑺𝒕𝒆𝒑 𝟏:
𝑺𝒕𝒆𝒑 𝟐:](https://image.slidesharecdn.com/photosynthesis-copy-211007115149/85/Photosynthesis-27-320.jpg)
![3
𝑭𝒓𝒖𝒄𝒕𝒐𝒔𝒆
𝟏, 𝟔 − 𝒅𝒊𝒑𝒉𝒐𝒔.
3
𝑭𝒓𝒖𝒄𝒕𝒐𝒔𝒆 𝟔 −
𝒑𝒉𝒐𝒔𝒑𝒉𝒂𝒕𝒆
𝑃ℎ𝑜𝑠𝑝ℎ𝑎𝑡𝑎𝑠𝑒
𝟑 𝑷𝒊
① ②
𝑺𝒕𝒆𝒑 𝟑:
𝑺𝒕𝒆𝒑 𝟒:
𝑭𝒓𝒖𝒄𝒕𝒐𝒔𝒆
𝟔 − 𝒑𝒉𝒐𝒔𝒑𝒉𝒂𝒕𝒆
𝑮𝒍𝒖𝒄𝒐𝒔𝒆
𝟔 − 𝒑𝒉𝒐𝒔𝒑𝒉𝒂𝒕𝒆
𝑮𝑳𝑼𝑪𝑶𝑺𝑬
𝐼𝑠𝑜𝑚𝑒𝑟𝑎𝑠𝑒
𝑷𝒊
[𝟔 𝑪] [𝟔 𝑪]
[𝟔 𝑪]](https://image.slidesharecdn.com/photosynthesis-copy-211007115149/85/Photosynthesis-28-320.jpg)
![2 [𝑷𝑮𝑨𝑳] 2 [𝑭𝒓𝒖𝒄𝒕𝒐𝒔𝒆 𝟔 − 𝒑𝒉𝒐𝒔𝒑𝒉𝒂𝒕𝒆]
+
[𝟐 × 𝟑 𝑪] [𝟐 × 𝟔 𝑪]
𝑇𝑟𝑎𝑛𝑠 − 𝑘𝑒𝑡𝑜𝑙𝑎𝑠𝑒
2
𝑿𝒚𝒍𝒖𝒍𝒐𝒔𝒆
𝟓 − 𝒑𝒉𝒐𝒔𝒑𝒉𝒂𝒕𝒆
2
𝑬𝒓𝒚𝒕𝒉𝒓𝒐𝒔𝒆
𝟒 − 𝒑𝒉𝒐𝒔𝒑𝒉𝒂𝒕𝒆
+
[𝟐 × 𝟓 𝑪] [𝟐 × 𝟒 𝑪]
𝑺𝒕𝒆𝒑 𝟓:
𝑺𝒕𝒆𝒑 𝟑](https://image.slidesharecdn.com/photosynthesis-copy-211007115149/85/Photosynthesis-29-320.jpg)
![+
[𝟐 × 𝟒 𝑪] [𝟐 × 𝟑 𝑪]
𝑇𝑟𝑎𝑛𝑠 − 𝑎𝑙𝑑𝑜𝑙𝑎𝑠𝑒
2
𝑺𝒆𝒅𝒐𝒉𝒆𝒑𝒕𝒖𝒍𝒐𝒔𝒆
𝟏, 𝟕 − 𝒅𝒊𝒑𝒉𝒐𝒔𝒑𝒉𝒂𝒕𝒆
2
𝑬𝒓𝒚𝒕𝒉𝒓𝒐𝒔𝒆
𝟒 − 𝒑𝒉𝒐𝒔𝒑𝒉𝒂𝒕𝒆
[𝟐 × 𝟕 𝑪]
𝑺𝒕𝒆𝒑 𝟔: 𝑺𝒕𝒆𝒑 𝟏
2 [𝑫𝑯𝑨𝑷]
[𝟐 × 𝑷] [𝟐 × 𝑷]
[𝟐 × 𝟐 𝑷]
2
𝑺𝒆𝒅𝒐𝒉𝒆𝒑𝒕𝒖𝒍𝒐𝒔𝒆
𝟕 − 𝒑𝒉𝒐𝒔𝒑𝒉𝒂𝒕𝒆
𝑃ℎ𝑜𝑠𝑝ℎ𝑎𝑡𝑎𝑠𝑒
[𝟐 × 𝟕 𝑪]
[𝟐 × 𝑷]
𝑺𝒕𝒆𝒑 𝟕:
𝟐 𝑷𝒊](https://image.slidesharecdn.com/photosynthesis-copy-211007115149/85/Photosynthesis-30-320.jpg)
![2 [𝑷𝑮𝑨𝑳] 2 [𝑺𝒆𝒅𝒐𝒉𝒆𝒑𝒕𝒖𝒍𝒐𝒔𝒆 𝟕 − 𝒑𝒉𝒐𝒔𝒑𝒉𝒂𝒕𝒆]
+
[𝟐 × 𝟑 𝑪] [𝟐 × 𝟕 𝑪]
𝑇𝑟𝑎𝑛𝑠 − 𝑘𝑒𝑡𝑜𝑙𝑎𝑠𝑒
2
𝑿𝒚𝒍𝒖𝒍𝒐𝒔𝒆
𝟓 − 𝒑𝒉𝒐𝒔𝒑𝒉𝒂𝒕𝒆
2
𝑹𝒊𝒃𝒐𝒔𝒆
𝟓 − 𝒑𝒉𝒐𝒔𝒑𝒉𝒂𝒕𝒆
+
[𝟐 × 𝟓 𝑪] [𝟐 × 𝟓 𝑪]
𝑺𝒕𝒆𝒑 𝟖:](https://image.slidesharecdn.com/photosynthesis-copy-211007115149/85/Photosynthesis-31-320.jpg)
![𝑺𝒕𝒆𝒑 𝟗:
2 𝑿𝒚𝒍𝒖. 𝟓 − 𝑷
[𝟒 × 𝟓 𝑪]
2 𝑿𝒚𝒍𝒖. 𝟓 − 𝑷
𝑺𝒕𝒆𝒑 𝟓 𝑺𝒕𝒆𝒑 𝟖
4
𝑿𝒚𝒍𝒖𝒍𝒐𝒔𝒆
𝟓 − 𝒑𝒉𝒐𝒔𝒑𝒉𝒂𝒕𝒆
4
𝑹𝒊𝒃𝒖𝒍𝒐𝒔𝒆
𝟓 − 𝒑𝒉𝒐𝒔𝒑𝒉𝒂𝒕𝒆
𝐸𝑝𝑖𝑚𝑒𝑟𝑎𝑠𝑒
[𝟒 × 𝟓 𝑪]
𝑺𝒕𝒆𝒑 𝟏𝟎:
2
𝑹𝒊𝒃𝒐𝒔𝒆
𝟓 − 𝒑𝒉𝒐𝒔𝒑𝒉𝒂𝒕𝒆
[𝟐 × 𝟓 𝑪]
𝐼𝑠𝑜𝑚𝑒𝑟𝑎𝑠𝑒
2
𝑹𝒊𝒃𝒖𝒍𝒐𝒔𝒆
𝟓 − 𝒑𝒉𝒐𝒔𝒑𝒉𝒂𝒕𝒆
[𝟐 × 𝟓 𝑪]
𝑺𝒕𝒆𝒑 𝟖](https://image.slidesharecdn.com/photosynthesis-copy-211007115149/85/Photosynthesis-32-320.jpg)
![𝑺𝒕𝒆𝒑 𝟏𝟏:
6
𝑹𝒊𝒃𝒖𝒍𝒐𝒔𝒆
𝟓 − 𝒑𝒉𝒐𝒔𝒑𝒉𝒂𝒕𝒆
[𝟔 × 𝟓 𝑪]
[𝟔 × 𝑷]
6
𝑹𝒊𝒃𝒖𝒍𝒐𝒔𝒆
𝟏, 𝟓 − 𝒃𝒊𝒔𝒑𝒉𝒐𝒔𝒑𝒉𝒂𝒕𝒆
[𝟔 × 𝟓 𝑪]
[𝟔 × 𝟐 𝑷]
𝑃ℎ𝑜𝑠𝑝ℎ𝑜𝑟𝑖𝑏𝑢𝑙𝑜𝑠𝑒
𝑘𝑖𝑛𝑎𝑠𝑒
𝟔 𝑨𝑫𝑷
𝟔 𝑨𝑻𝑷](https://image.slidesharecdn.com/photosynthesis-copy-211007115149/85/Photosynthesis-33-320.jpg)
![𝟔 𝑹𝒖𝑩𝑷 + 6 𝐶𝑂2 + 6 𝐻2𝑂
𝟏𝟐 𝑷𝑮𝑨 + 12 𝐴𝑇𝑃 + 12 𝑁𝐴𝐷𝑃𝐻 + 12 𝐻+
𝟏𝟐 [𝑷𝑮𝑨]
𝟏𝟐 𝑷𝑮𝑨𝑳 + 12 𝐴𝐷𝑃 + 12 𝑃𝑖 + 12 𝑁𝐴𝐷𝑃+
𝟏𝟐 𝑷𝑮𝑨𝑳 + 6 𝐴𝑇𝑃
𝑯𝑬𝑿𝑶𝑺𝑬 (𝑭𝒓𝒖𝒄. 𝟔 − 𝑷) + 𝟔 𝑹𝒖𝑩𝑷 + 6 𝐴𝐷𝑃 + 6 𝑃𝑖](https://image.slidesharecdn.com/photosynthesis-copy-211007115149/85/Photosynthesis-34-320.jpg)
![𝐶𝑜𝑚𝑏𝑖𝑛𝑖𝑛𝑔 𝑎𝑙𝑙 3 𝑟𝑒𝑎𝑐𝑡𝑖𝑜𝑛𝑠:
𝟔 𝑪𝑶𝟐 + 𝟔 𝑯𝟐𝑶 + 18 𝐴𝑇𝑃 + 12 [𝑁𝐴𝐷𝑃𝐻 + 𝐻+]
𝑯𝑬𝑿𝑶𝑺𝑬 + 18 [𝐴𝐷𝑃 + 𝑃𝑖] + 12 [𝑁𝐴𝐷𝑃+]](https://image.slidesharecdn.com/photosynthesis-copy-211007115149/85/Photosynthesis-35-320.jpg)
![𝟑 − 𝑷𝑮𝑨𝑳 (𝟏𝟐)
𝟐 𝟐 𝟓 𝟑
𝑫𝑯𝑨𝑷
𝟐 𝟑
𝑭𝒓𝒖𝒄. 𝟏, 𝟔
−𝒅𝒊𝒑𝒉𝒐𝒔.
𝑭𝒓𝒖𝒄. 𝟔 − 𝑷
(𝟑)
(𝟑)
𝑯𝑬𝑿𝑶𝑺𝑬
𝟏
𝟐
𝑿𝒚𝒍. 𝟓 − 𝑷
𝑬𝒓𝒚. 𝟒 − 𝑷
(𝟐)
𝑺𝒆𝒅. 𝟏, 𝟕 − 𝒅𝒊𝒑.
(𝟐)
𝑺𝒆𝒅. 𝟕 − 𝑷
(𝟐)
𝑹𝒊𝒃. 𝟓 − 𝑷
(𝟐) (𝟒)
𝑹𝒊𝒃𝒖. 𝟓 − 𝑷
(𝟔)
𝑹𝒖𝑩𝑷
𝟑 − 𝑷𝑮𝑨
(𝟔)
𝟏, 𝟑 − 𝒅𝒊𝑷𝑮𝑨
(𝟏𝟐) (𝟏𝟐)
⟵ 𝟔 𝑪𝑶𝟐
⟶ 𝟑𝑷𝒊
⟶ 𝟐𝑷𝒊
⟶ 𝟔 𝑨𝑫𝑷
⟵ 𝟔 𝑨𝑻𝑷
𝟏𝟐 𝑨𝑻𝑷 𝟏𝟐 𝑨𝑫𝑷
↓ ↑
𝟏𝟐 [𝑵𝑨𝑫𝑷𝑯 + 𝑯+] 𝟏𝟐 𝑵𝑨𝑫𝑷+
↓ ↑
𝟏𝟐 𝑷𝒊 ↘](https://image.slidesharecdn.com/photosynthesis-copy-211007115149/85/Photosynthesis-36-320.jpg)

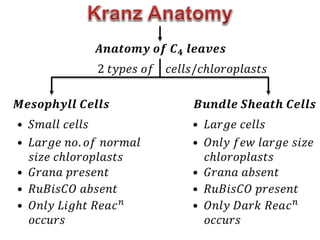
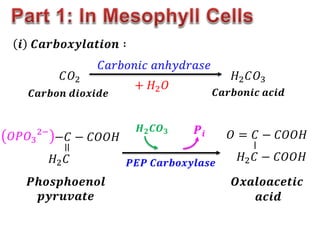
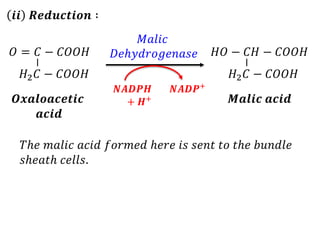
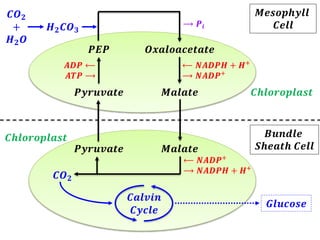

![𝐴𝑡 𝑛𝑖𝑔ℎ𝑡, 𝑡ℎ𝑒 𝑠𝑡𝑜𝑚𝑎𝑡𝑎 𝑜𝑓 𝑝𝑙𝑎𝑛𝑡 𝑜𝑝𝑒𝑛 𝑎𝑙𝑙𝑜𝑤𝑖𝑛𝑔 𝐶𝑂2 𝑡𝑜
𝑑𝑖𝑓𝑓𝑢𝑠𝑒 𝑖𝑛𝑡𝑜 𝑡ℎ𝑒 𝑚𝑒𝑠𝑜𝑝ℎ𝑦𝑙𝑙 𝑐𝑒𝑙𝑙𝑠.
𝑪𝑶𝟐 𝑯𝑪𝑶𝟑
−
+ 𝑶𝑯 −
[𝑩𝒊𝒄𝒂𝒓𝒃𝒐𝒏𝒂𝒕𝒆]
𝑷𝑬𝑷
𝑷𝒉𝒐𝒔𝒑𝒉𝒐𝒆𝒏𝒐𝒍
𝒑𝒚𝒓𝒖𝒗𝒂𝒕𝒆
𝑯𝑪𝑶𝟑
−
𝑯𝟑𝑷𝑶𝟒
𝑶𝑨𝑨
𝑶𝒙𝒂𝒍𝒐𝒂𝒄𝒆𝒕𝒂𝒕𝒆
𝑶𝑨𝑨 𝑴𝒂𝒍𝒂𝒕𝒆
𝑀𝑎𝑙𝑎𝑡𝑒 𝐷𝑒ℎ𝑦𝑑𝑟𝑜𝑔𝑒𝑛𝑎𝑠𝑒
𝑵𝑨𝑫𝑷+
𝑵𝑨𝑫𝑷𝑯 + 𝑯+
𝑃𝐸𝑃 𝐶𝑎𝑟𝑏𝑜𝑥𝑦𝑙𝑎𝑠𝑒](https://image.slidesharecdn.com/photosynthesis-copy-211007115149/85/Photosynthesis-45-320.jpg)

