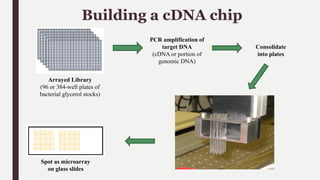
DNA Mikroarray 'DNA çip', 'gen çip', 'genom çip', veya gen-dizi olarak da bilinen, genellikle her biri bir geni temsil eden, ayrı ayrı küçük katı yüzeye kovalent bağlarla sabitlenmiş binlerce DNA parçacıkları toplusudur.
Her DNA spotlarda 10-12 mol spesifik DNA sekansları bulunur ki “probe” yada oligo olarak bilinirler.
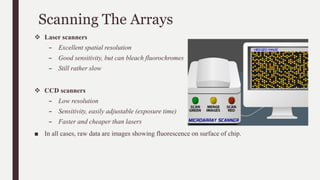
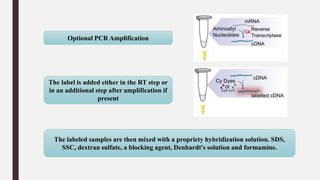
Bilinen her gen veya probe çip üzerinde belirli bir noktada oturup ve değişen seviyelerde floresan aktivitesi, dahil edilen genetik materyalde değişen seviyelerde gen aktivitesi gösterir.
Prob sekanslara bağlanan floresan etiketli hedef diziler bir sinyal üretir.