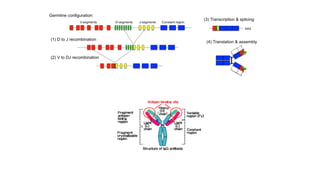
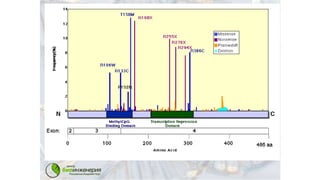
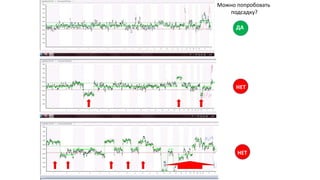
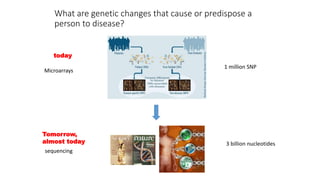
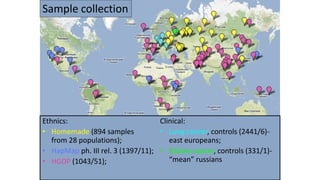
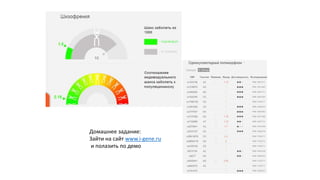
Документ описывает методологию и результаты анализа геномов древних микробиомов, включая щетинокамин, мамонта и носорога, основанные на более чем 10,000 последовательностях 16s rRNA. Также упоминаются технологии сборки геномов и новые исследования микроматриц ДНК для многопараметрического анализа. Результаты указывают на важность геномной информации для понимания заболеваний и развития новых диагностических решений.