How to manually align an XCT reconstructed volume with ImageJ
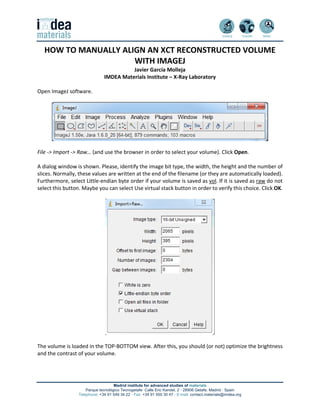
- 1. Madrid institute for advanced studies of materials Parque tecnológico Tecnogetafe· Calle Eric Kandel, 2 · 28906 Getafe, Madrid · Spain Telephone: +34 91 549 34 22 · Fax: +34 91 550 30 47 · E-mail: contact.materials@imdea.org HOW TO MANUALLY ALIGN AN XCT RECONSTRUCTED VOLUME WITH IMAGEJ Javier García Molleja IMDEA Materials Institute – X‐Ray Laboratory Open ImageJ software. File ‐> Import ‐> Raw… (and use the browser in order to select your volume). Click Open. A dialog window is shown. Please, identify the image bit type, the width, the height and the number of slices. Normally, these values are written at the end of the filename (or they are automatically loaded). Furthermore, select Little‐endian byte order if your volume is saved as vol. If it is saved as raw do not select this button. Maybe you can select Use virtual stack button in order to verify this choice. Click OK. The volume is loaded in the TOP‐BOTTOM view. After this, you should (or not) optimize the brightness and the contrast of your volume.
- 2. Madrid institute for advanced studies of materials Parque tecnológico Tecnogetafe· Calle Eric Kandel, 2 · 28906 Getafe, Madrid · Spain Telephone: +34 91 549 34 22 · Fax: +34 91 550 30 47 · E-mail: contact.materials@imdea.org Image ‐> Adjust ‐> Brightness/Contrast… (previously, seek a slice without shadows or artefacts). The fast way is by clicking Auto and then Apply, but it is important you can confirm these automatic choices. Yes to apply to all slices. Now the volume is visually improved by brightness and contrast modification of the LUT table. Click the button Magnifying glass to see the upper part of the volume and then click Angle tool to see if it is properly aligned. Scrolling tool can be useful. These buttons are in the ImageJ main window.
- 3. Madrid institute for advanced studies of materials Parque tecnológico Tecnogetafe· Calle Eric Kandel, 2 · 28906 Getafe, Madrid · Spain Telephone: +34 91 549 34 22 · Fax: +34 91 550 30 47 · E-mail: contact.materials@imdea.org In ImageJ main window the measured angle is 90.04 degrees (two angle points in the sample upper part and the third point is located in a vertical manner), so in order to correctly align the volume you need to rotate 0.04 degrees counter clockwise. Image ‐> Transform ‐> Rotate... Angle (degrees): ‐0.04, Grid Lines: 1: Interpolation: Bilinear. Click OK. Yes to apply to all slices. Note that clockwise rotations are positive and counter clockwise rotations are negative! Please, write this value in a notepad. Next, you need to change to the RIGHT‐LEFT view. Image ‐> Stacks ‐> Reslice... Output spacing (pixels): 1.000, Start at: Left, Flip vertically: uncheck, Rotate 90 degrees: uncheck, Avoid interpolation: check. Click OK and wait to memory allocation and volume reordering. You will see a yellow region during the process. A new window appears. Go to a slice from the middle (where the shadows are minimal) and click the Angle tool button. However, you may work with a bigger window. That is done by: Image ‐> Transform ‐> Rotate 90 Degrees Right. Indeed, you can save this step checking a button in the previous step.
- 4. Madrid institute for advanced studies of materials Parque tecnológico Tecnogetafe· Calle Eric Kandel, 2 · 28906 Getafe, Madrid · Spain Telephone: +34 91 549 34 22 · Fax: +34 91 550 30 47 · E-mail: contact.materials@imdea.org Now you can use the Angle tool button and make a good measurement in the upper region of the sample. The ImageJ main window depicts 90.33 degrees, so you need to rotate 0.33 degrees counter clockwise. Image ‐> Transform ‐> Rotate… Angle (degrees): ‐0.33, Grid Lines: 1: Interpolation: Bilinear. Then click OK and click Yes to the question if apply or not to all slices. Write this value, please. Now, you should go to the TOP‐BOTTOM view again, so you need to revert all changes:
- 5. Madrid institute for advanced studies of materials Parque tecnológico Tecnogetafe· Calle Eric Kandel, 2 · 28906 Getafe, Madrid · Spain Telephone: +34 91 549 34 22 · Fax: +34 91 550 30 47 · E-mail: contact.materials@imdea.org Image ‐> Transform ‐> Rotate 90 Degrees Left Image ‐> Stacks ‐> Reslice… Output spacing (pixels): 1.000, Start at: Top, Flip vertically: uncheck, Rotate 90 degrees: uncheck, Avoid interpolation: check. Wait to memory allocation and volume reordering. A new window appears. Image ‐> Transform ‐> Rotate 90 Degress Right (of couse, you can save some steps if you check or uncheck some buttons in the previous steps, go ahead!)
- 6. Madrid institute for advanced studies of materials Parque tecnológico Tecnogetafe· Calle Eric Kandel, 2 · 28906 Getafe, Madrid · Spain Telephone: +34 91 549 34 22 · Fax: +34 91 550 30 47 · E-mail: contact.materials@imdea.org Watch out! If you compare the starting volume you will see that this new volume is flipped. There is some operation with a weird ordering sequence. Furthermore, you need to verify with the starting volume that slices are ordered at the same manner, i.e., the beginning of one is the beginning of other. ORIGINAL LAST STEP Image ‐> Transform ‐> Flip Horizontally in order to reach the good outcome. Click Yes to apply to all slices. Now it is time to see the FRONT‐BACK view. Image ‐> Stacks ‐> Reslice… Output spacing (pixels): 1.000, Start at: Top, Flip vertically: uncheck, Rotate 90 degrees: uncheck, Avoid interpolation: check. Click OK. Memory will be allocated and the yellow region shows the reordering process. At these stages you should check the available memory of your computer (Ctrl+Shift+Esc is a good short key).
- 7. Madrid institute for advanced studies of materials Parque tecnológico Tecnogetafe· Calle Eric Kandel, 2 · 28906 Getafe, Madrid · Spain Telephone: +34 91 549 34 22 · Fax: +34 91 550 30 47 · E-mail: contact.materials@imdea.org Use the Angle tool button in the upper region of the sample. ImageJ main window will show 89.06 degrees, so you need to rotate 0.94 degrees clockwise. Image ‐> Transform ‐> Rotate… Angle (degrees): 0.94, Grid Lines: 1, Interpolation: Bilinear. Click OK and then click Yes. Please, write this value.
- 8. Madrid institute for advanced studies of materials Parque tecnológico Tecnogetafe· Calle Eric Kandel, 2 · 28906 Getafe, Madrid · Spain Telephone: +34 91 549 34 22 · Fax: +34 91 550 30 47 · E-mail: contact.materials@imdea.org Now it is time to obtain, again, the TOP‐BOTTOM view. Image ‐> Stacks ‐> Reslice… Output spacing (pixels): 1.000, Start at: Top, Flip vertically: uncheck, Rotate 90 degrees: uncheck, Avoid interpolation: check. Click OK. Wait to the memory allocation and volume reordering. A new window will appear. Now you can click the Rectangular tool button in the ImageJ main window and make a ROI (Region Of Interest). You can skip the air (outer darkest regions) and save memory for the new volume. Try to include all sample in the new ROI.
- 9. Madrid institute for advanced studies of materials Parque tecnológico Tecnogetafe· Calle Eric Kandel, 2 · 28906 Getafe, Madrid · Spain Telephone: +34 91 549 34 22 · Fax: +34 91 550 30 47 · E-mail: contact.materials@imdea.org Image ‐> Crop. Be careful, there is no undo option after this process. Now, use the slider to go to the first slices and the final ones. Write the range in which the sample is free of zero‐grey regions (created after rotations) and shadows (created by the reconstruction process). In this case slices from 164 to 2133 include the sample without artefacts and avoid the column stage. These slices are not important for sample processing, so you can delete them in order to save more memory. Leave some air around your sample. Image ‐> Duplicate… Title: the proposed one, Duplicate stack: check, Range: 164‐2133 and click OK. Finally, it is time to save your work (you can save at intermediate points if you are short of memory, for example). You write the name of the sample, the duplicated stack, the bit image type, the angles for alignment, the crop process and the volume size. File ‐> Save As ‐> Raw Data… (use the browser to find a good location, it is advisable to make a new folder in order to separate reconstructed volumes from the worked ones, typically its name is worked vols). RTM6_NCF_P1550_2.2_2000_F_164‐2133_16b_AngleCorr‐0p04‐0p33+0p94_crop_1980x260x1970.raw (Thanks to Iker Lizarralde for providing me the volume and to Federico Sket for the idea.)
