More Related Content
PPTX
Curriculum Learning (関東CV勉強会) PDF
PPTX
【DL輪読会】ViT + Self Supervised Learningまとめ PDF
【DL輪読会】Unbiased Gradient Estimation for Marginal Log-likelihood PDF
PDF
PPTX
PyTorch, PixyzによるGenerative Query Networkの実装 PDF
What's hot
PPTX
DLゼミ: Ego-Body Pose Estimation via Ego-Head Pose Estimation PDF
PDF
PPTX
音響メディア信号処理における独立成分分析の発展と応用, History of independent component analysis for sou... PDF
PDF
PPTX
ブラックボックスからXAI (説明可能なAI) へ - LIME (Local Interpretable Model-agnostic Explanat... PDF
PPTX
PDF
PDF
PDF
Deep State Space Models for Time Series Forecasting の紹介 PDF
サポートベクターマシン(SVM)の数学をみんなに説明したいだけの会 PDF
DOCX
PPTX
【DL輪読会】言語以外でのTransformerのまとめ (ViT, Perceiver, Frozen Pretrained Transformer etc) PDF
SSII2021 [OS2-02] 深層学習におけるデータ拡張の原理と最新動向 PDF
PPTX
[研究室論文紹介用スライド] Adversarial Contrastive Estimation PDF
Similar to 独立成分分析とPerfume
PPTX
Feature Selection with R / in JP PDF
PPTX
for関数を使った繰り返し処理によるヒストグラムの一括出力 PDF
「plyrパッケージで君も前処理スタ☆」改め「plyrパッケージ徹底入門」 PDF
10分で分かるr言語入門ver2.10 14 1101 PDF
PDF
10分で分かるr言語入門ver2.15 15 1010 PPTX
PDF
10分で分かるr言語入門ver2.8 14 0712 PDF
2016年12月18日AITC女子会 データ分析勉強会第7回「Rを使ったデータ分析の基礎・分析手法について知る」 PPTX
Protein-Protein Interaction Prediction PPTX
30分でわかる『R』によるデータ分析|データアーティスト PDF
XCMSを用いた質量分析データ処理_BioCAsia2021_yamamoto.pdf PDF
10分で分かるr言語入門ver2.9 14 0920 PDF
PDF
PPT
PPT
K030 appstat201203 2variable PDF
10分で分かるr言語入門ver2 upload用 PDF
10分で分かるr言語入門ver2.14 15 0905 独立成分分析とPerfume
- 1.
- 2.
- 3.
- 4.
- 6.
- 7.
- 8.
- 9.
- 11.
- 12.
- 13.
- 14.
- 15.
- 16.
- 17.
- 18.
- 19.
- 20.
- 21.
- 22.
- 23.
- 24.
- 25.
- 26.
- 27.
- 28.
- 29.
- 30.
- 31.
- 32.
- 33.
- 34.
- 35.
- 36.
- 37.
- 38.
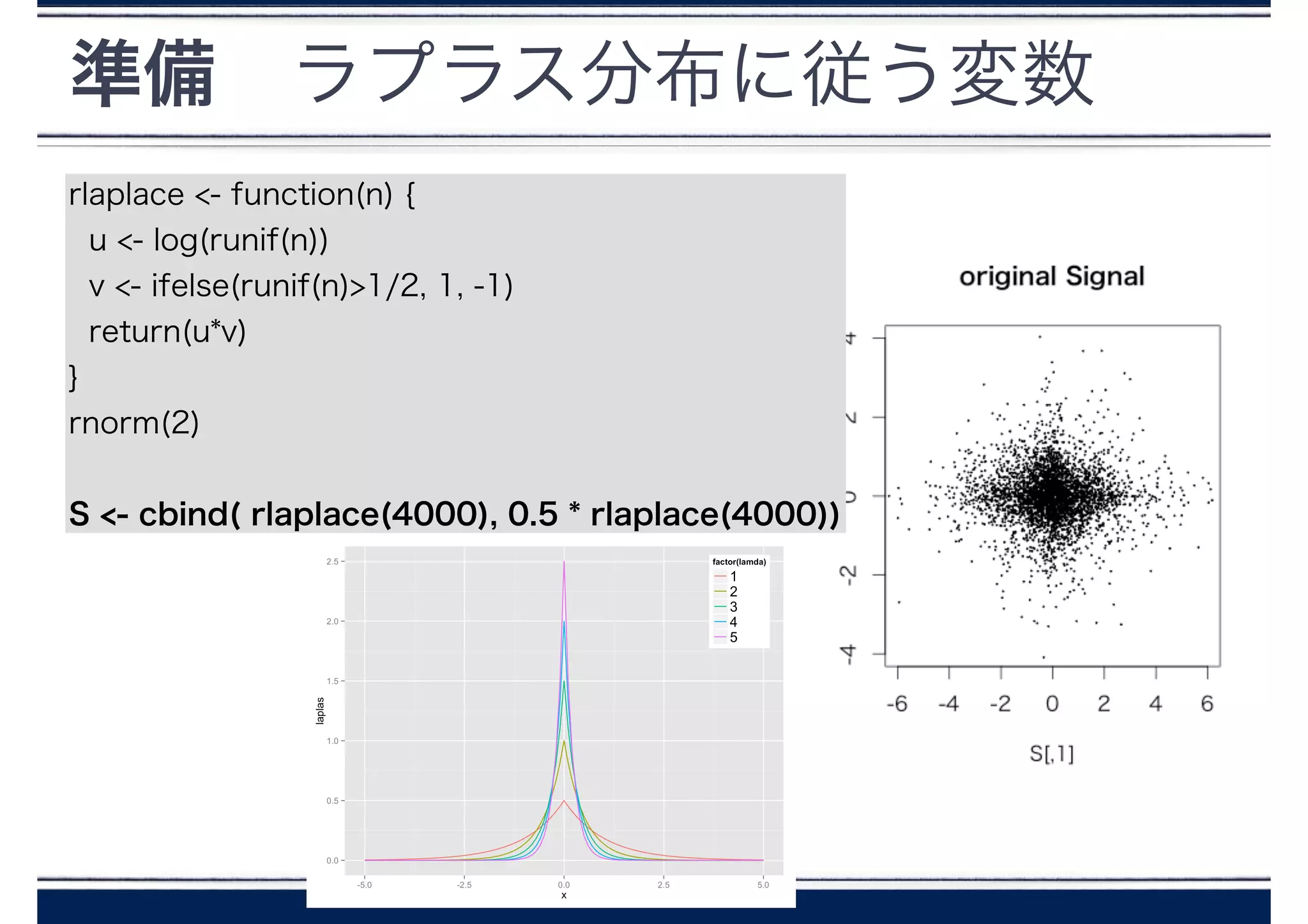
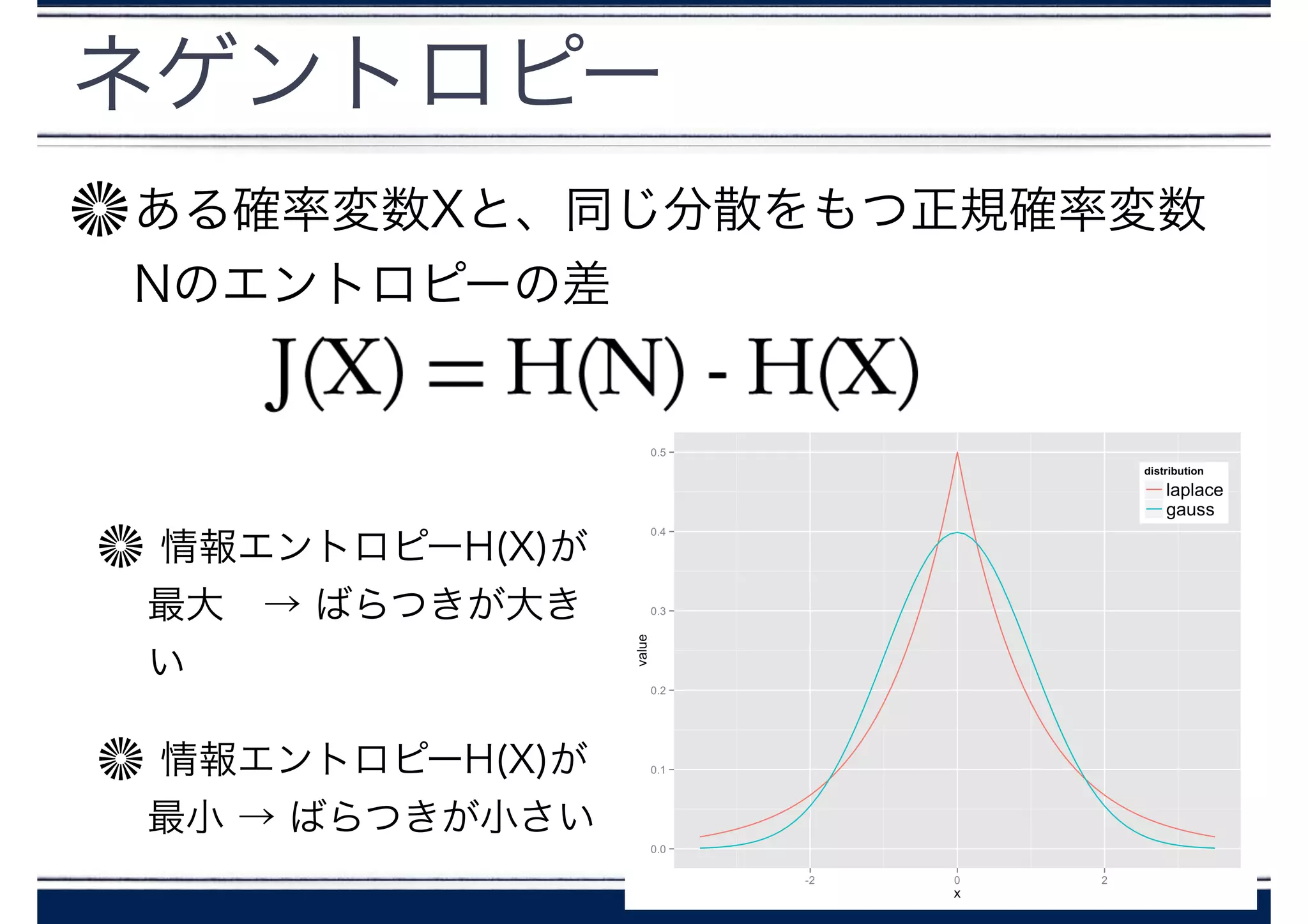
準備 ラプラス分布に従う変数
rlaplace <- function(n){
u <- log(runif(n))
v <- ifelse(runif(n)>1/2, 1, -1)
return(u*v)
}
rnorm(2)
!
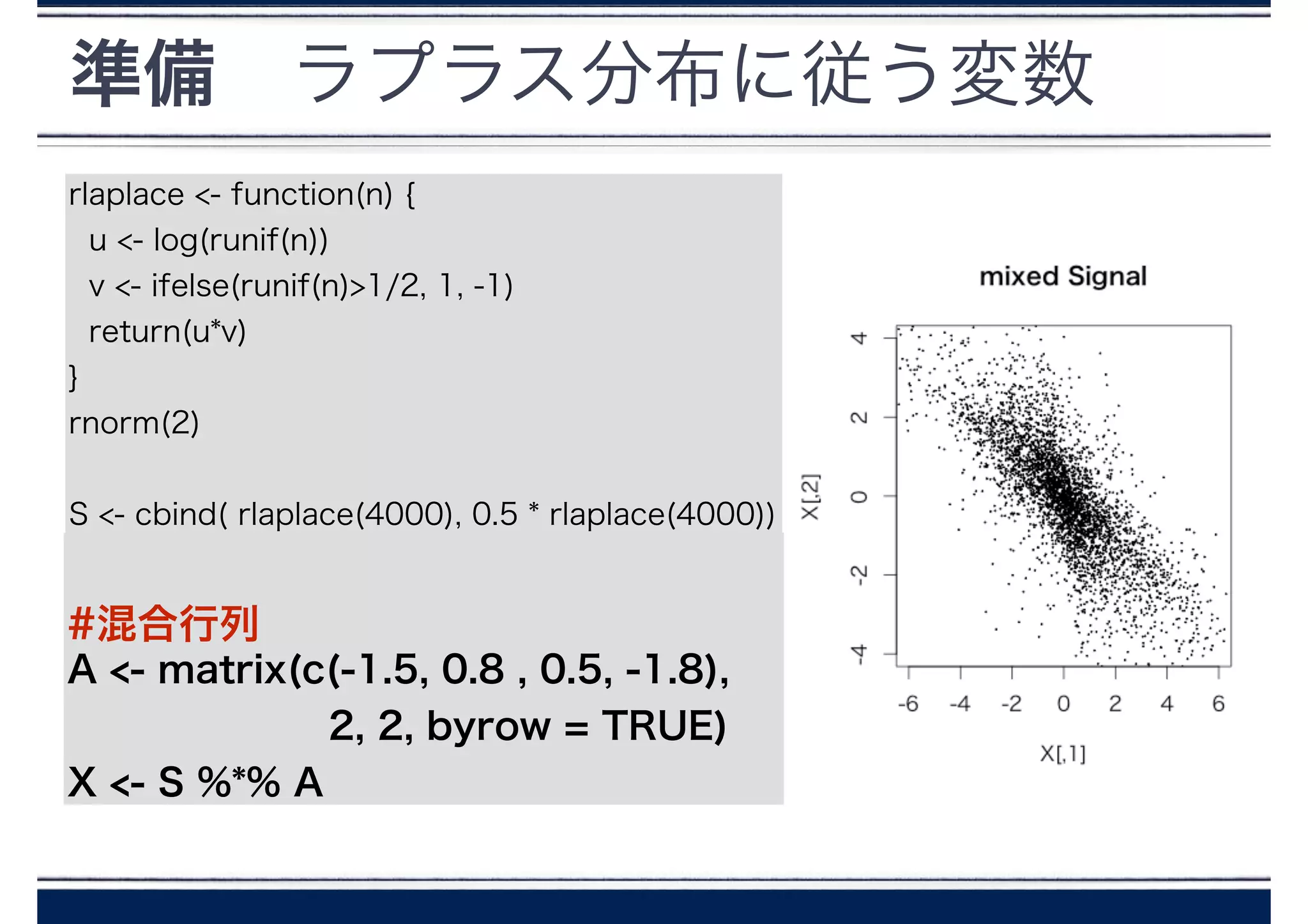
S <- cbind( rlaplace(4000), 0.5 * rlaplace(4000))
!
!
A <- matrix(c(-1.5, 0.8 , 0.5, -1.8),
2, 2, byrow = TRUE)
X <- S %*% A
#混合行列
- 39.
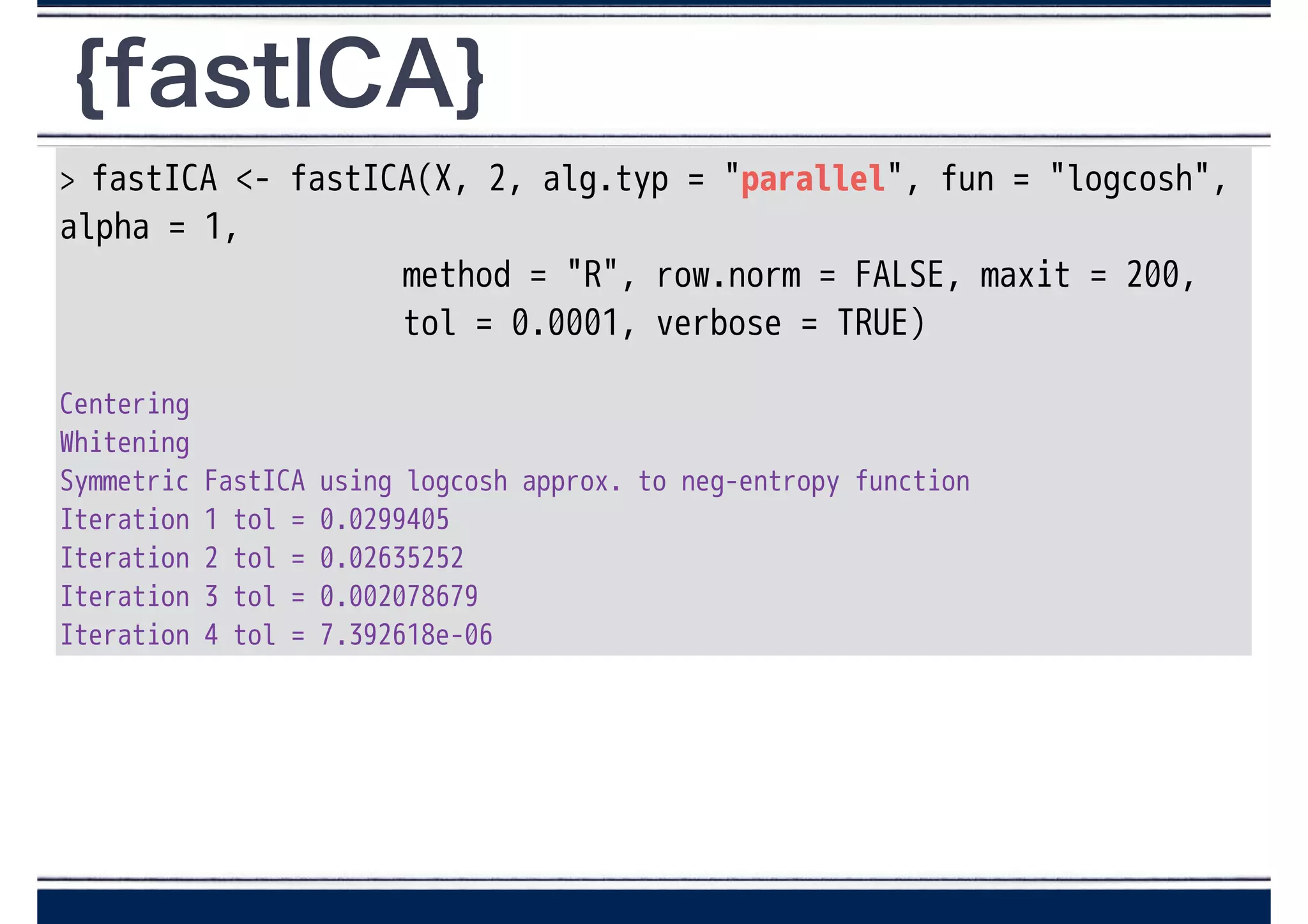
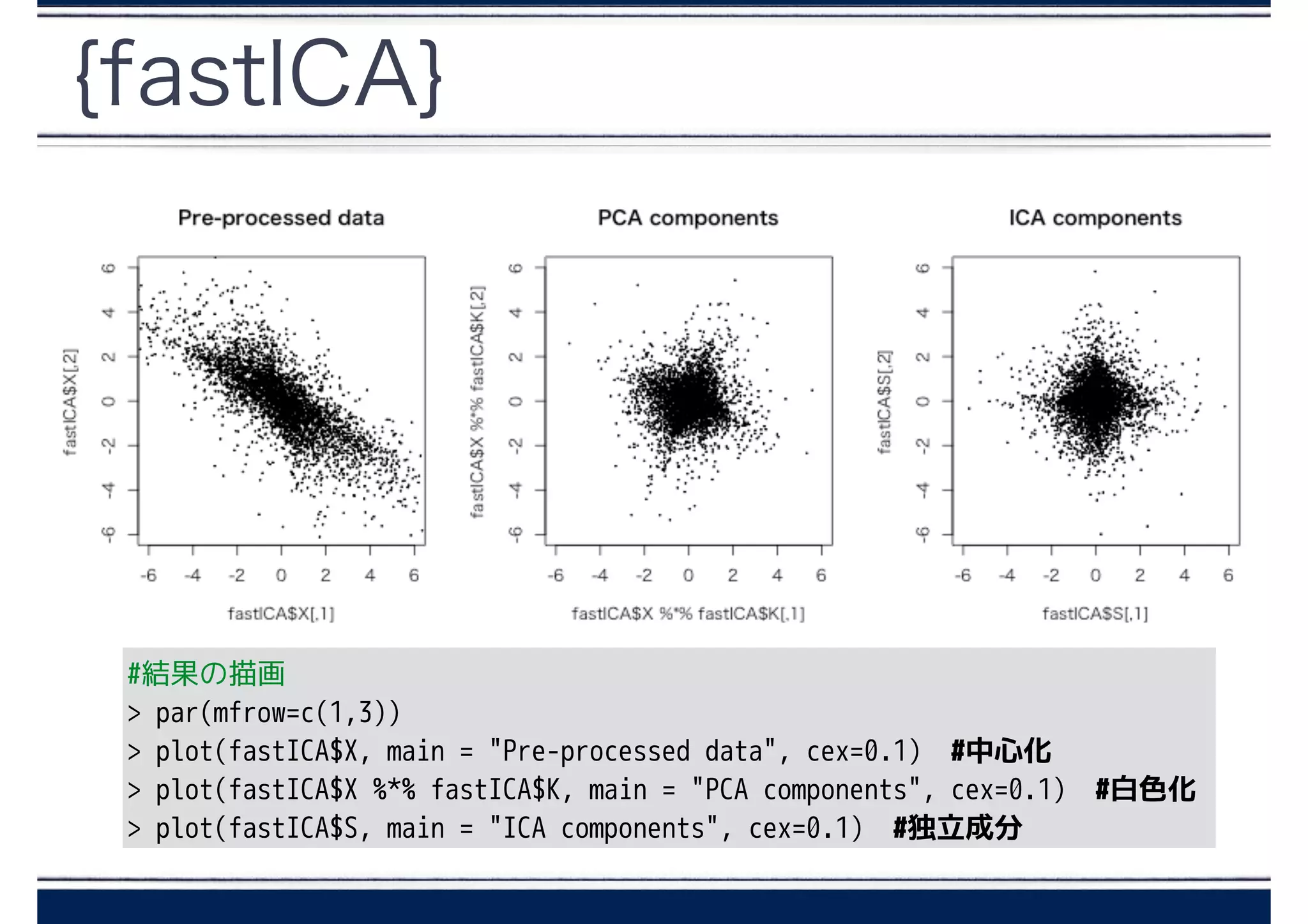
{fastICA}
> fastICA <-fastICA(X, 2, alg.typ = "parallel", fun = "logcosh",
alpha = 1,
method = "R", row.norm = FALSE, maxit = 200,
tol = 0.0001, verbose = TRUE)
!
Centering
Whitening
Symmetric FastICA using logcosh approx. to neg-entropy function
Iteration 1 tol = 0.0299405
Iteration 2 tol = 0.02635252
Iteration 3 tol = 0.002078679
Iteration 4 tol = 7.392618e-06
- 40.
- 41.
{fastICA} - fastICA()
fastICA(X,#[サンプル] x [観測値] の行列
n.comp, #分解するICの数
alg.typ = c(“parallel","deflation"), #ICを同時に推定するか
fun = c("logcosh","exp"), #G関数の形を決める
alpha = 1.0, #neg-entropyの形が"logcosh"のときに設定
#1~2 の間の定数
#αの値によって収束するまでの早さが変わる
method = R, #R→R言語で計算、C→C言語で計算
row.norm = FALSE, #Xが規格化されているかどうか
maxit = 200, #イテレーションの回数のリミット値
tol = 1e-04, #un-mixing matrixが収束したか決めるときのしきい値
verbose = FALSE, #結果をコンソールに表示するかどうか
w.init = NULL #非混合行列の初期値
)
- 42.
{fastICA} - fastICA()
fastICA(X,#[サンプル] x [観測値] の行列
n.comp, #分解するICの数
alg.typ = c(“parallel","deflation"), #ICを同時に推定するか
fun = c("logcosh","exp"), #G関数の形を決める
alpha = 1.0, #neg-entropyの形が"logcosh"のときに設定
#1~2 の間の定数
#αの値によって収束するまでの早さが変わる
method = R, #R→R言語で計算、C→C言語で計算
row.norm = FALSE, #Xが規格化されているかどうか
maxit = 200, #イテレーションの回数のリミット値
tol = 1e-04, #un-mixing matrixが収束したか決めるときのしきい値
verbose = FALSE, #結果をコンソールに表示するかどうか
w.init = NULL #非混合行列の初期値
)
- 43.
- 44.
- 45.
- 46.
- 47.
- 48.
- 49.
- 50.
- 51.
- 52.
- 53.
{fastICA} - fastICA()
fastICA(X,#[サンプル] x [観測値] の行列
n.comp, #分解するICの数
alg.typ = c(“parallel","deflation"), #ICを同時に推定するか
fun = c("logcosh","exp"), #G関数の形を決める
alpha = 1.0, #neg-entropyの形が"logcosh"のときに設定
#1~2 の間の定数
#αの値によって収束するまでの早さが変わる
method = R, #R→R言語で計算、C→C言語で計算
row.norm = FALSE, #Xが規格化されているかどうか
maxit = 200, #イテレーションの回数のリミット値
tol = 1e-04, #un-mixing matrixが収束したか決めるときのしきい値
verbose = FALSE, #結果をコンソールに表示するかどうか
w.init = NULL #非混合行列の初期値
)
- 54.
- 55.
- 56.
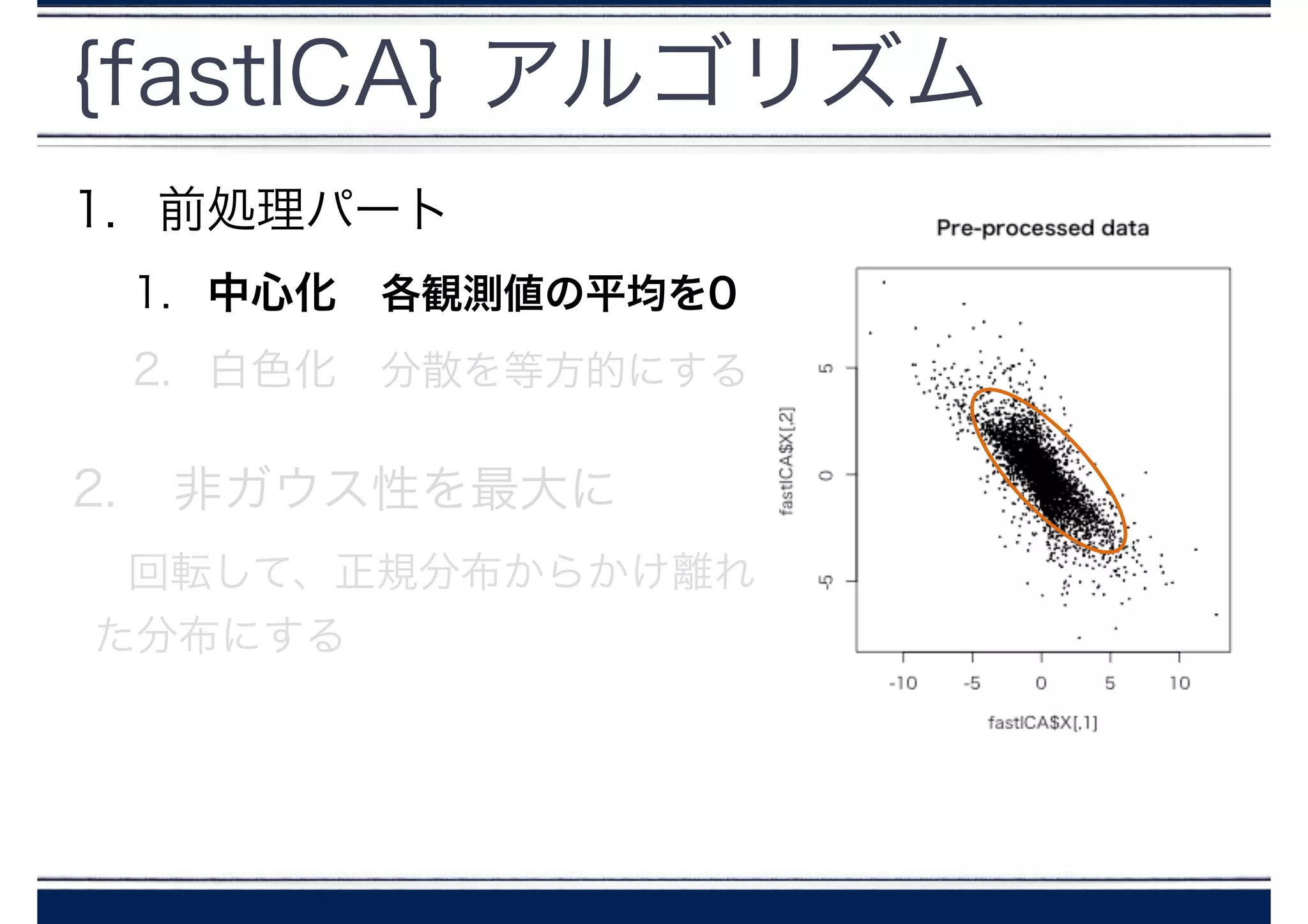
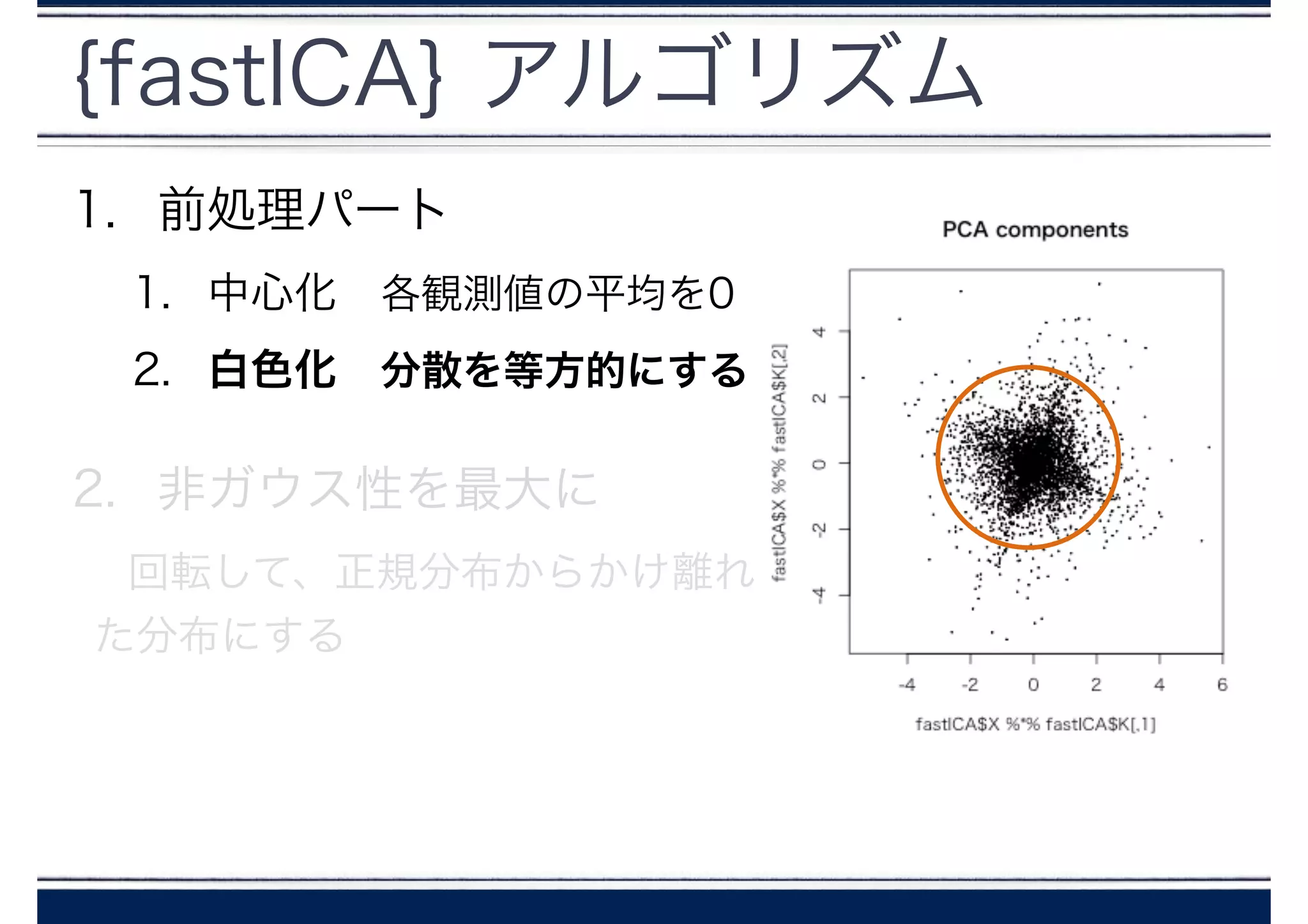
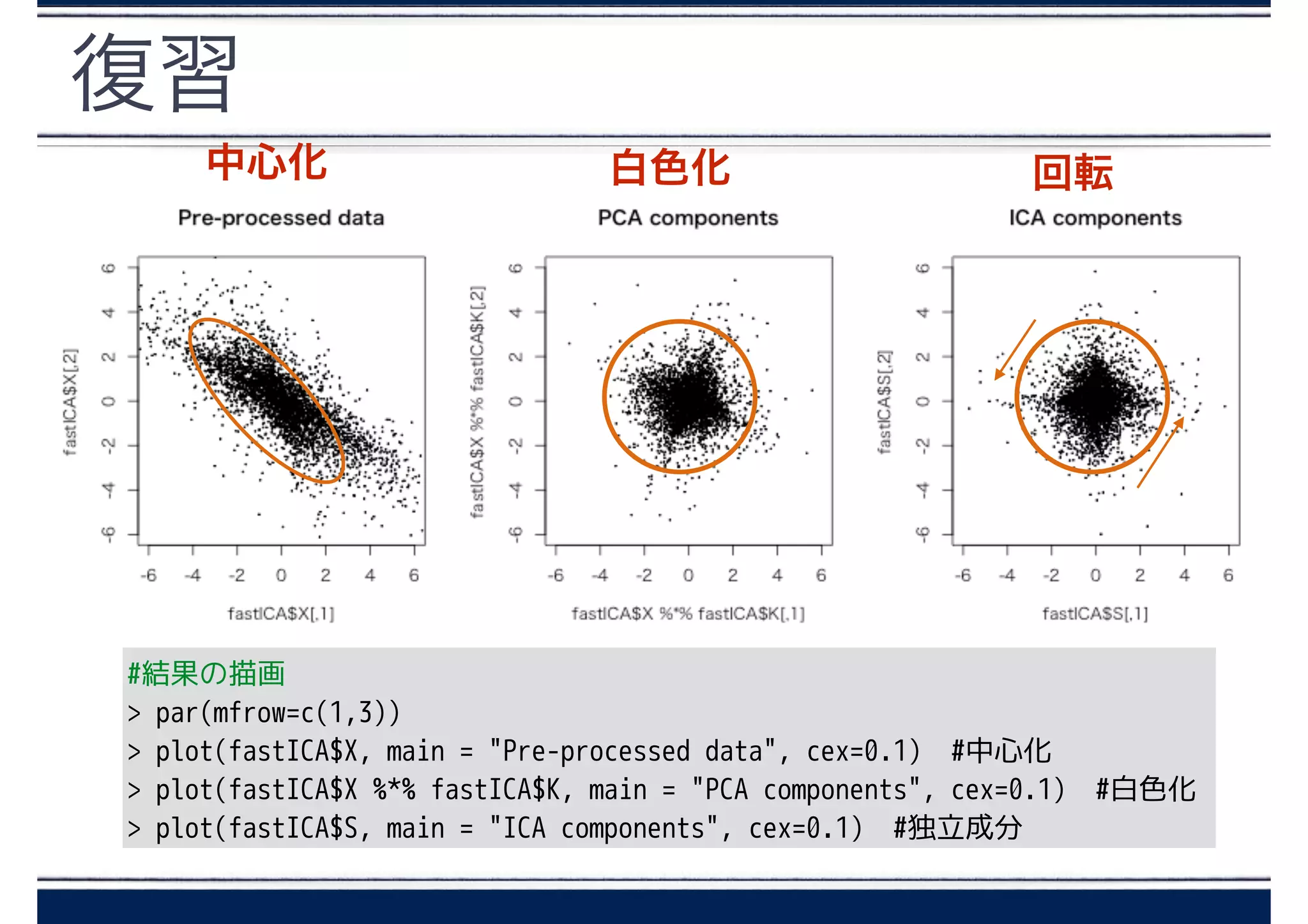
主成分分析 overview
定式化 &ポイント
!
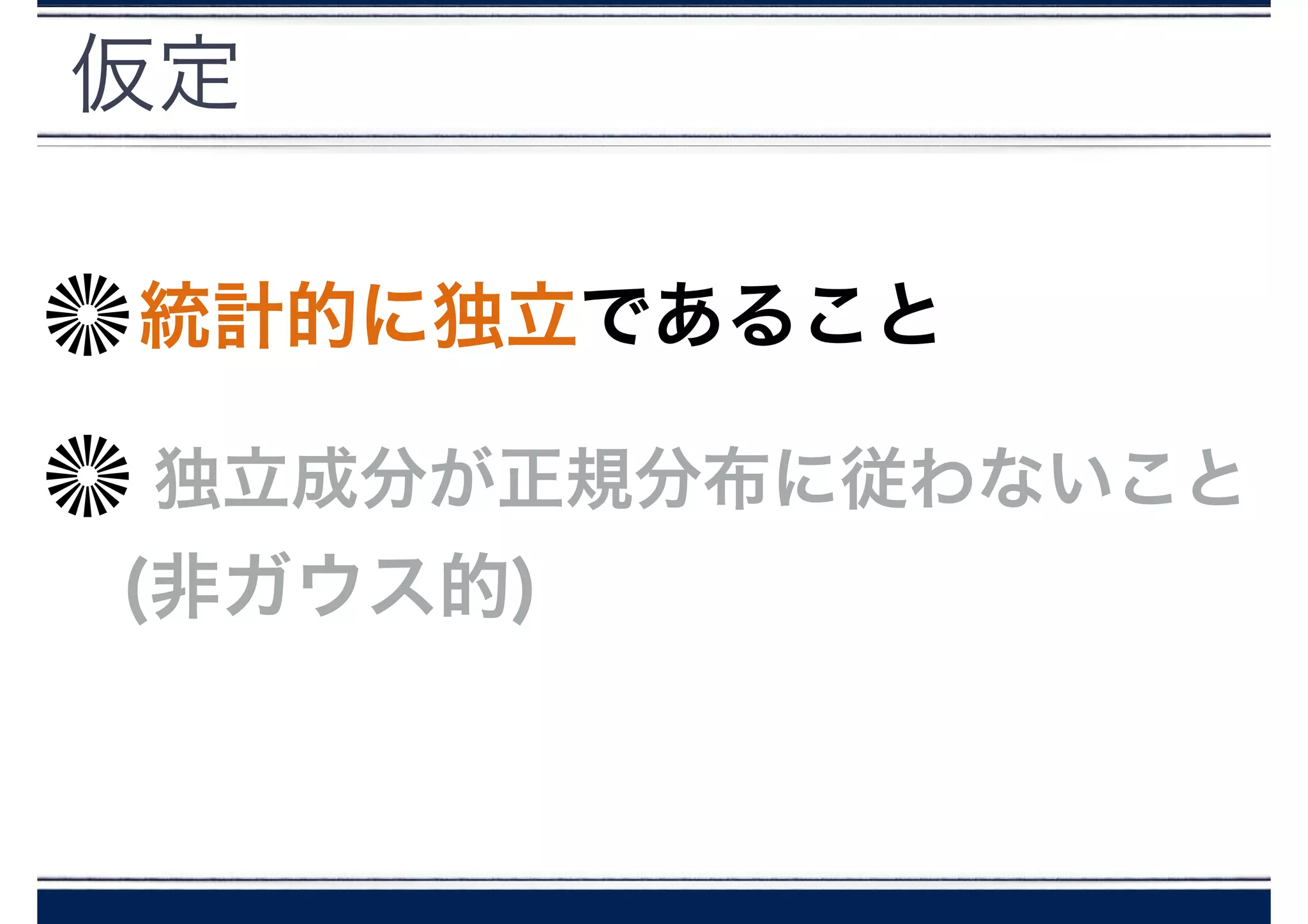
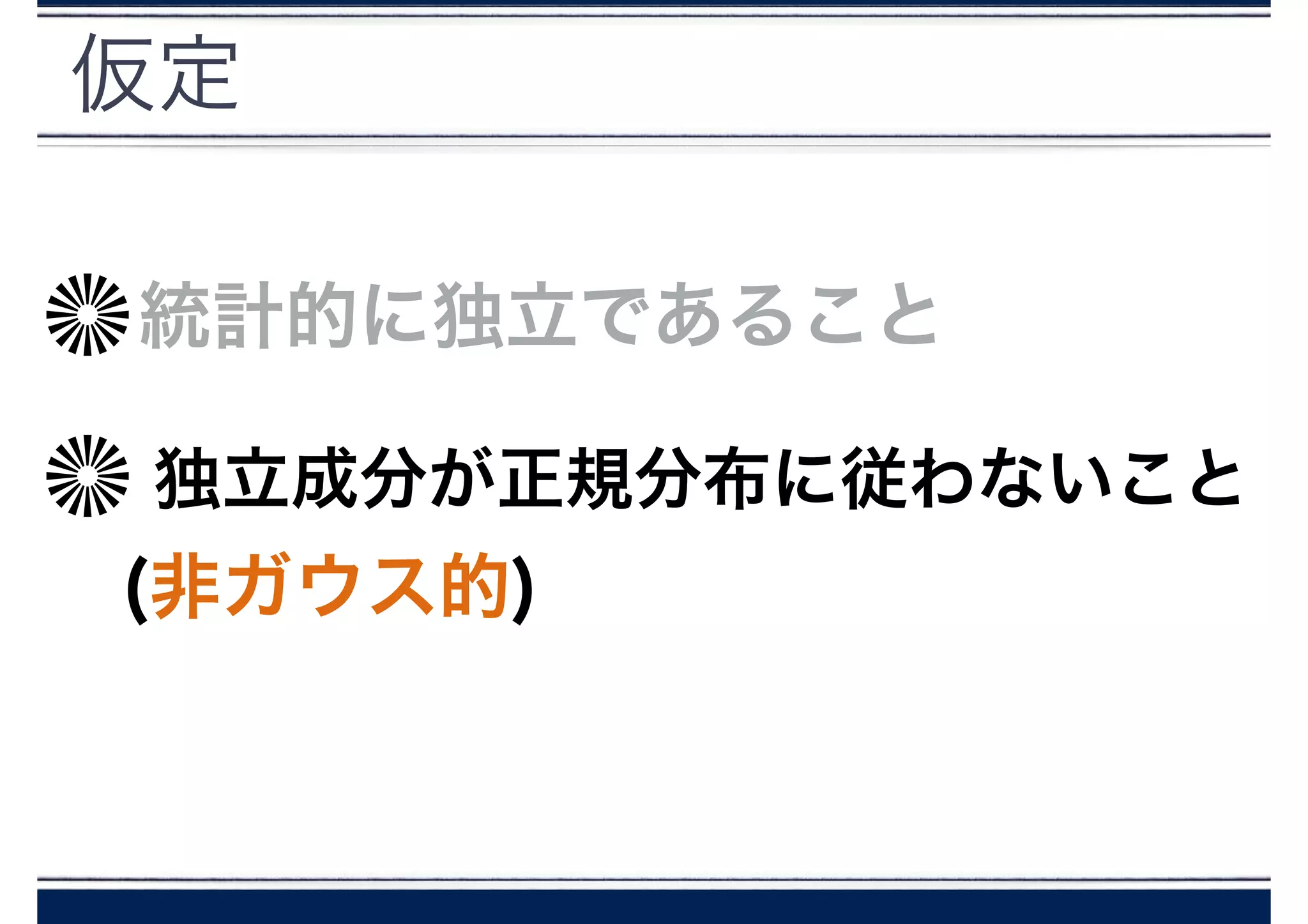
- 主成分は互いに相関はない(でも独立ではない)
- 主成分は、すべて正規分布に沿っている
正規分布の場合、相関が0なら独立性が保証される
使われ方
- 次元を削減する方法として用いられる
- ICA では、PCAを白色化のときに用いている。PCAの次
元が独立成分の要素の数に一致する
- 57.
- 58.
- 59.
- 60.
- 61.
- 62.
- 63.
- 64.
- 65.
- 66.
- 67.
- 68.
- 69.
- 70.
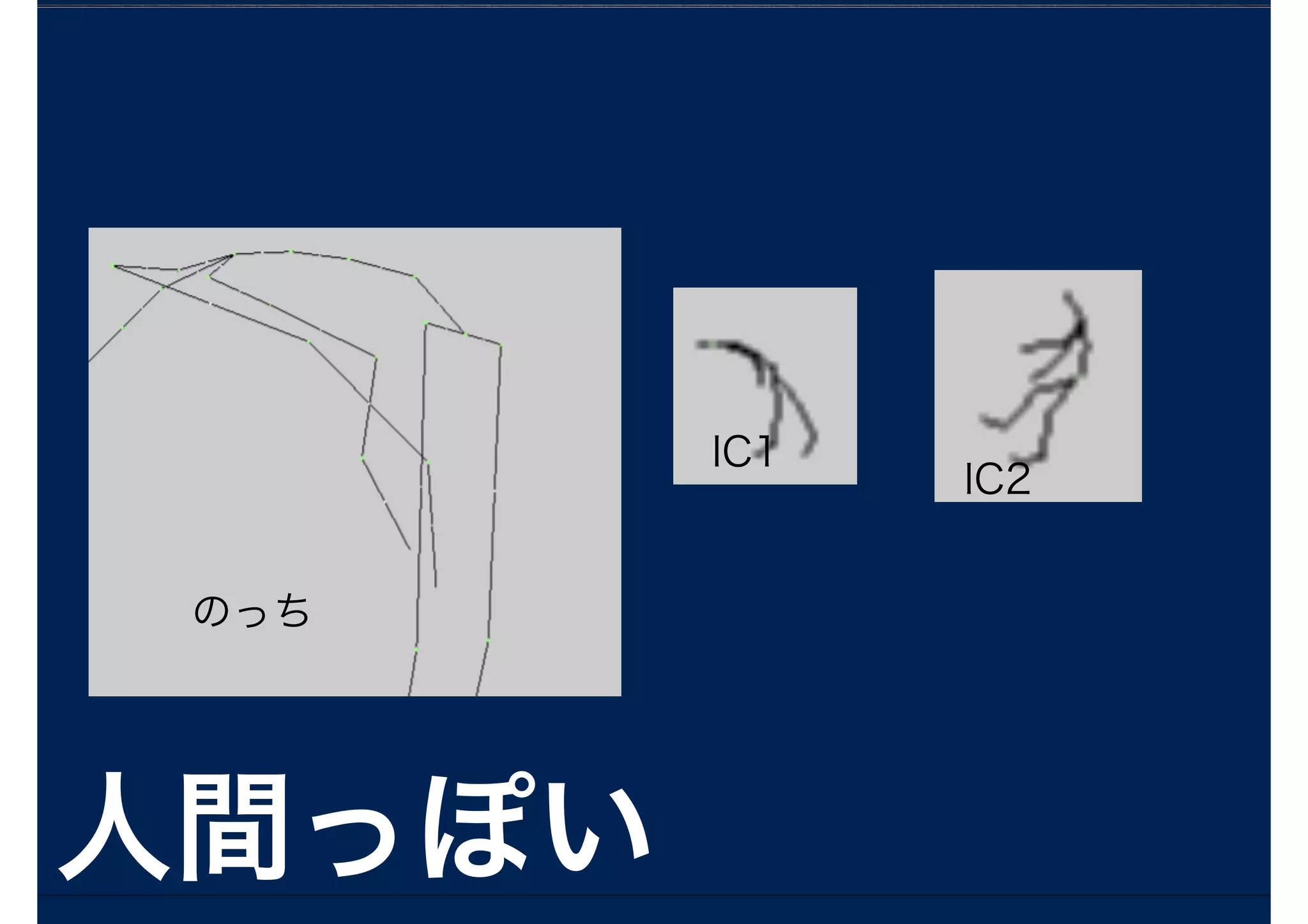
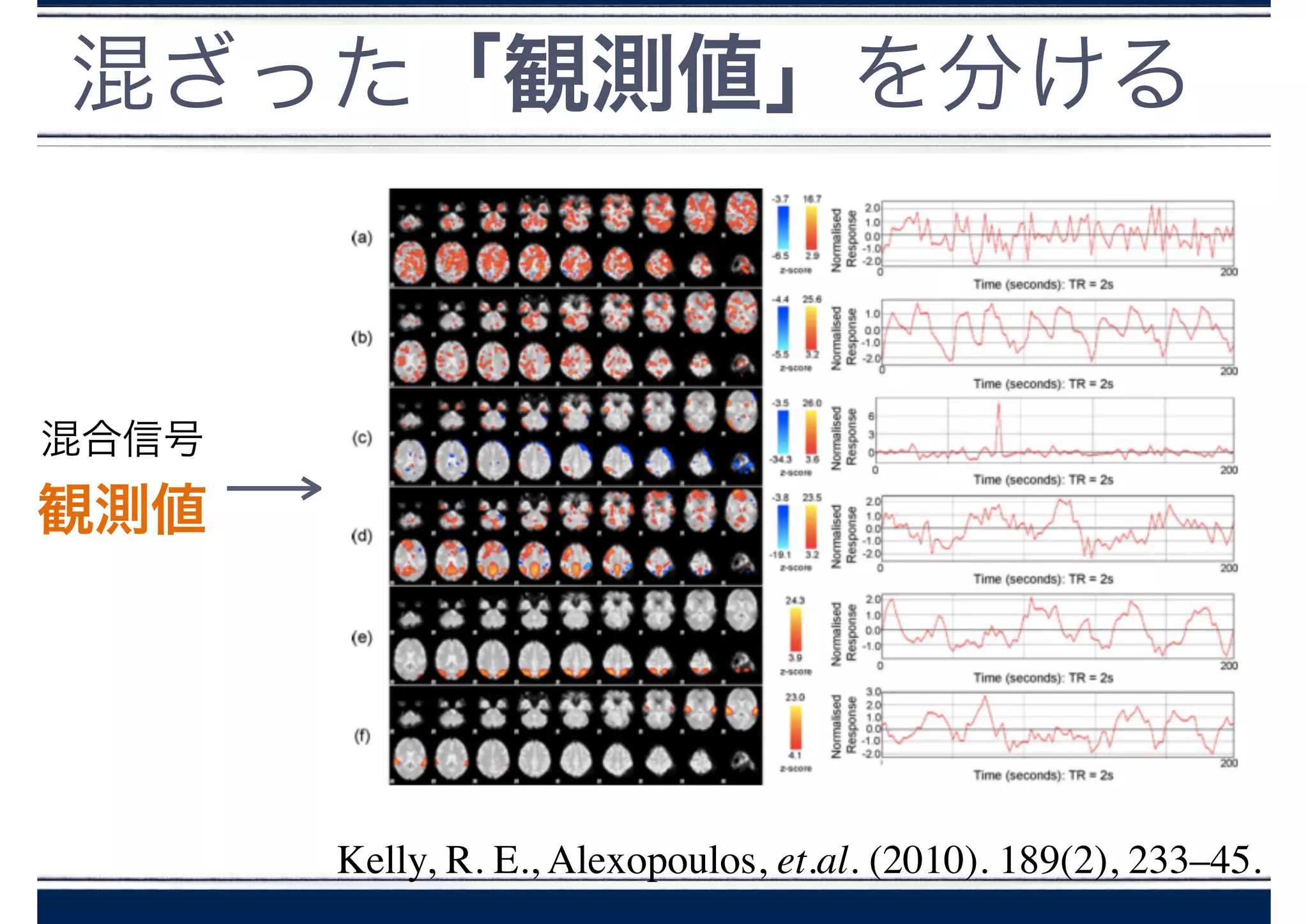
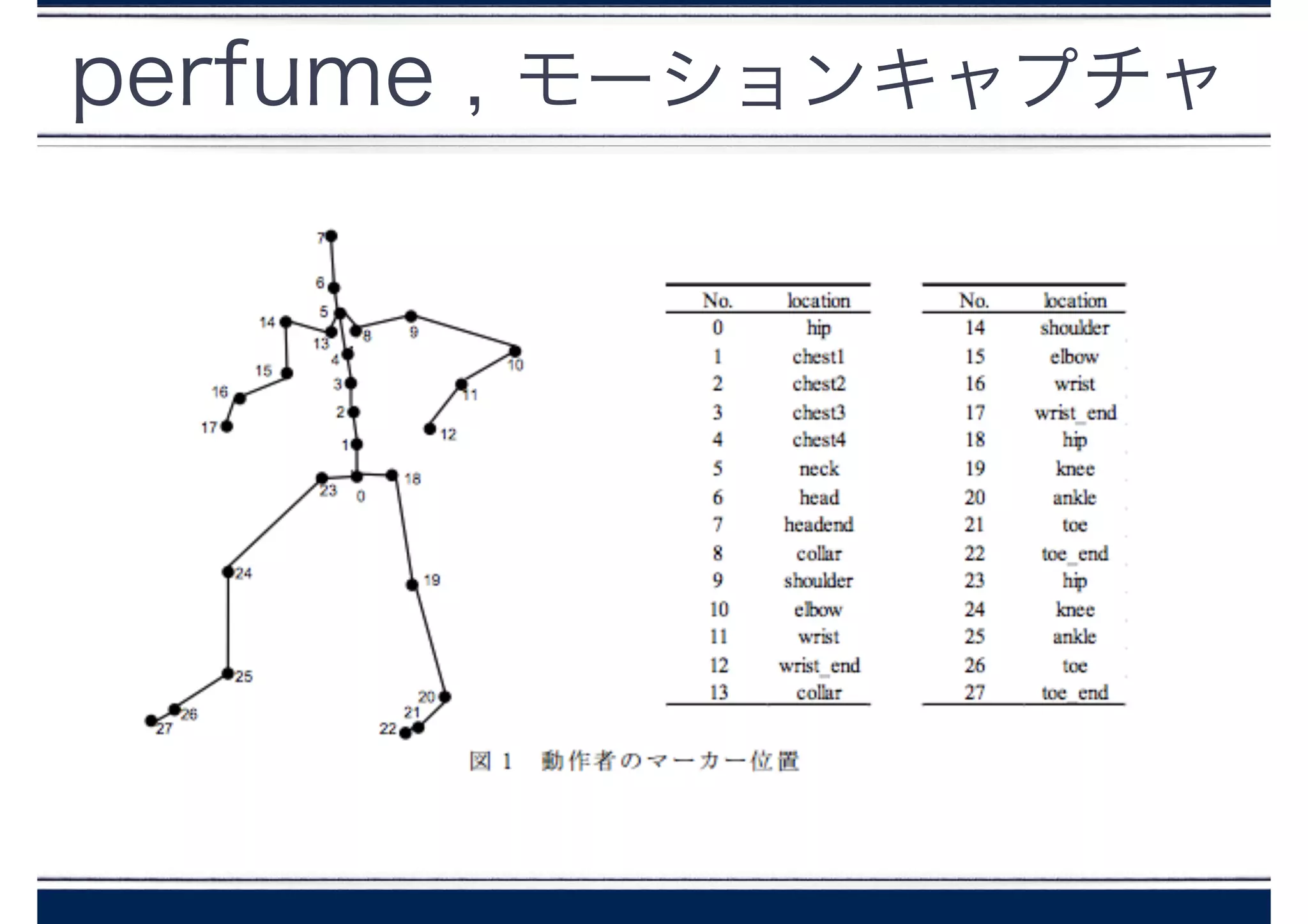
perfume , モーションキャプチャ
HIERARCHY
ROOTHips
{
OFFSET 0.000000 0.000000 0.000000
CHANNELS 6 Xposition Yposition Zposition Yrotation Xrotation
Zrotation
JOINT Chest
{
OFFSET 0.000000 10.678932 0.006280
CHANNELS 3 Yrotation Xrotation Zrotation
…,
}
MOTION
Frames: 2820
Frame Time: 0.025000
20.126024 83.31402 -18.424651 -9.129106 -6.517021 -0.118878
0.085519 9.753561 -0.537233 -0.010346 -5.417462 -0.070241 0.024747
1.596753 -0.097771 0.076825 0.255045 0.028866 -2.676143 13.137455
-0.003003 -4.902355 -11.317583 0.237067 0.227179 0.185755 0.125412
-0.119016 -11.836501 1.407777 2.313914 -7.359847 2.184012 7.121591
-4.059673 0.32346 0.027243 -2.476878 -0.074028 -0.097043 -7.619834
0.032649 -60.980338 -2.129588 -0.11772 0.245169 0.021119 9.869842
0.042872 8.453526 -4.572724 -3.248217 1.906954 -0.671854 2.029124
-4.900362 6.1163 0.030989 -0.279999 -0.068954 -11.233479
-12.051593 0.062998 1.140721 1.950329 1.686051 -0.00394 -4.658935
-8.5305 0.029141 -0.060092 0.0284
- 71.
- 72.
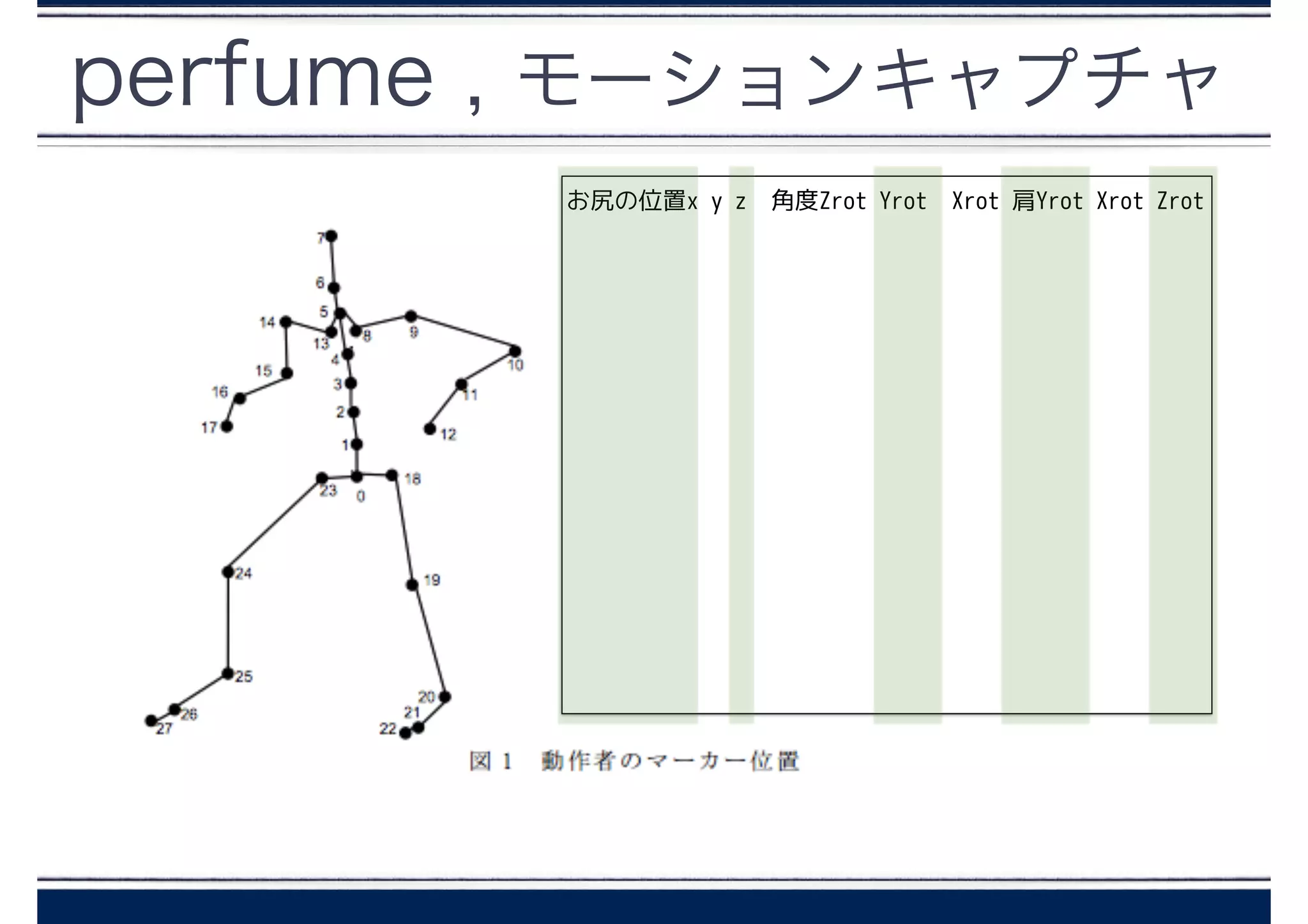
perfume データ準備
notch <-read.table("notch.dat", header = F)
aachan <- read.table("aachan.dat", header = F)
kashiyuka <- read.table("kashiyuka.dat", header = F)
X <- cbind(notch[,1], aachan[,1], kashiyuka[,1])
notch Y rot aachan Y rot kashiyuka Y rot
- 73.
主成分分析
Newperfume1 = {}
Newperfume2= {}
!
for( i in c(1:ncol(notch))){
X <- cbind(notch[,i], aachan[,i], kashiyuka[,i])
PCA <- prcomp(X)
Newperfume1 <- cbind(Newperfume1,PCA$x[,1])
Newperfume2 <- cbind(Newperfume2,PCA$x[,2])
}
write.table(x=round(Newperfume1,6), file="Newperfume1_PCA.bvh",row.names=F,
col.names=F)
write.table(x=round(Newperfume2,6), file="Newperfume2_PCA.bvh",row.names=F,
col.names=F)
!
- 74.
- 75.
- 76.
- 77.
- 78.
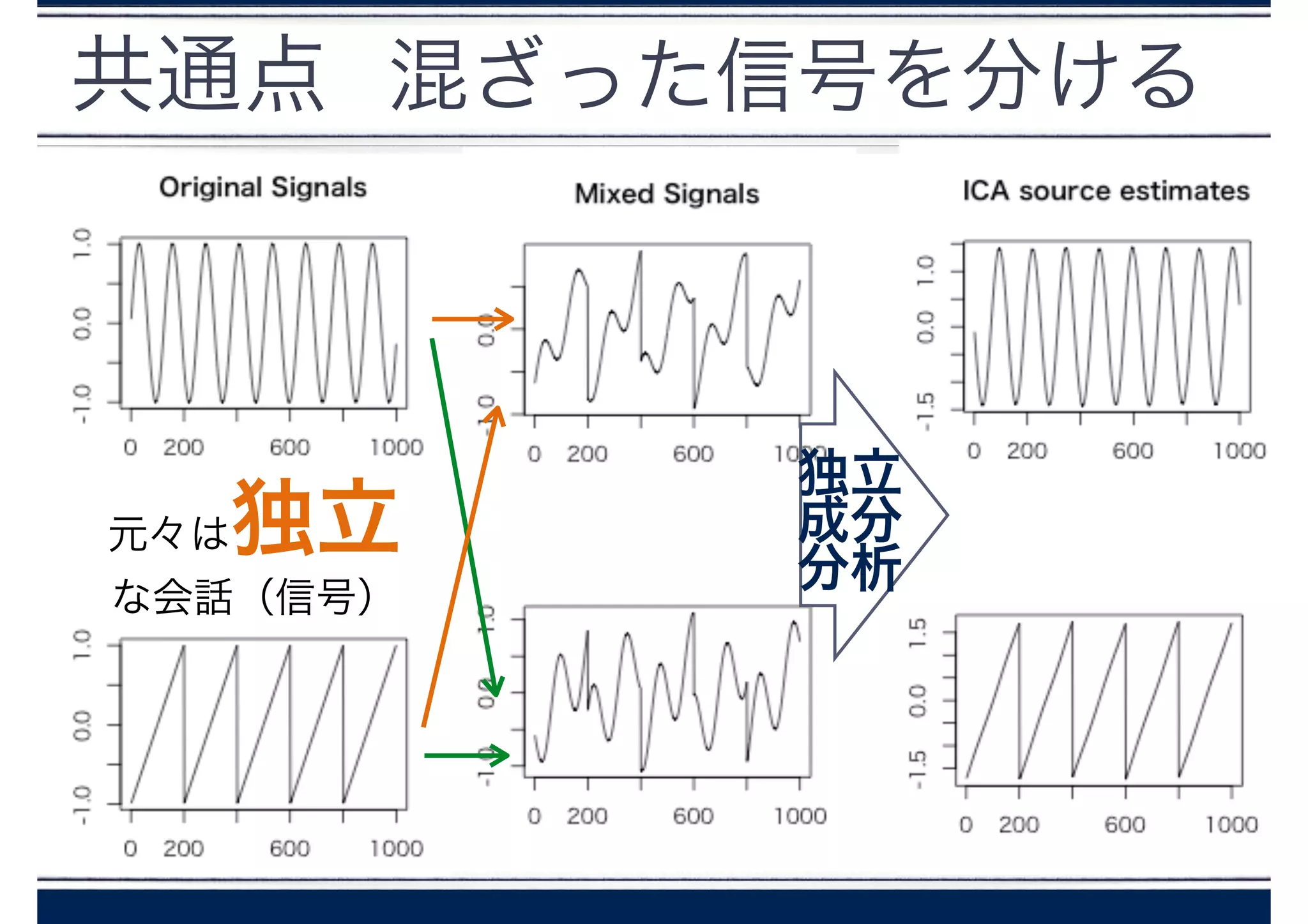
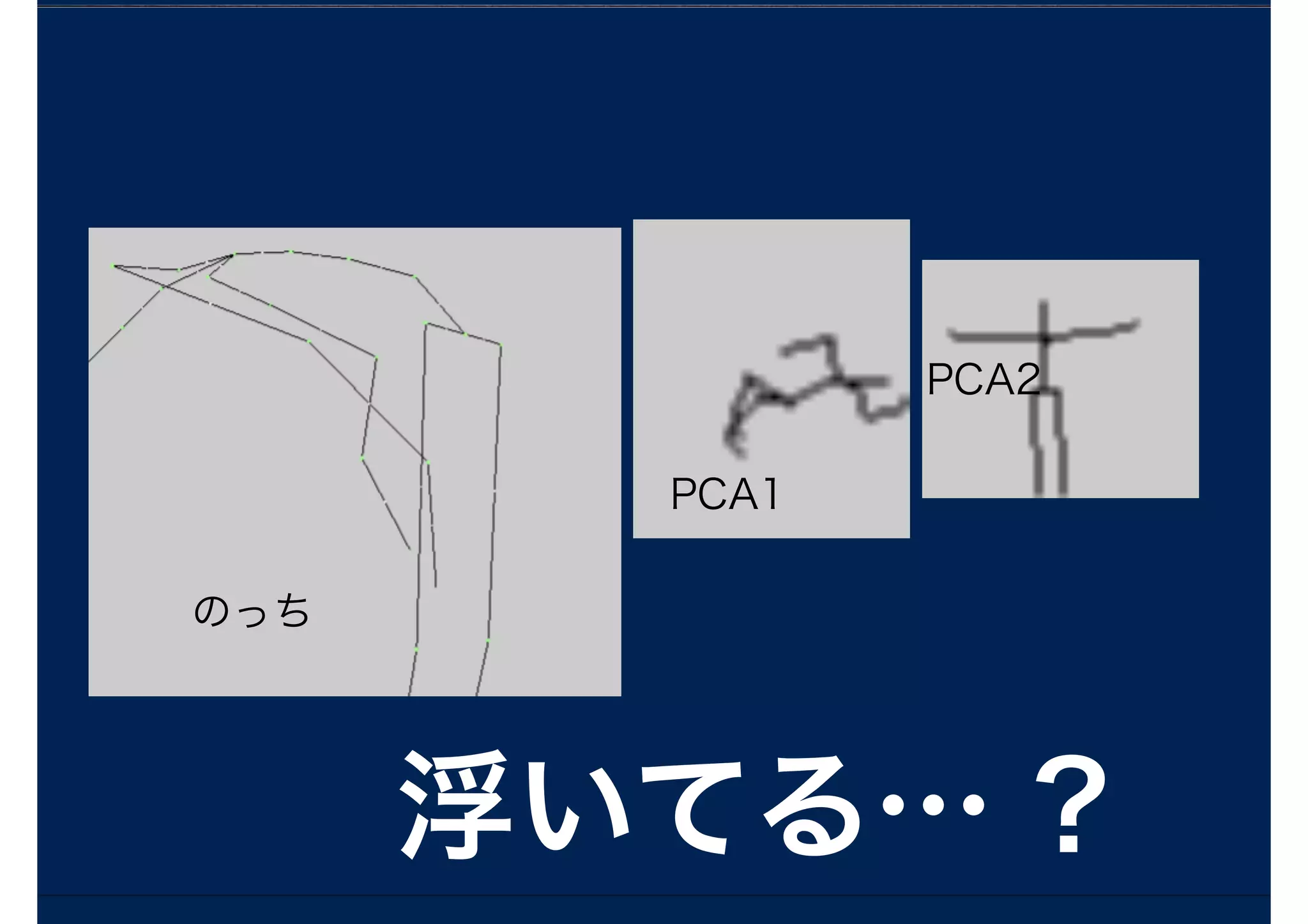
独立成分分析
Newperfume1 = {}
Newperfume2= {}
for( i in c(1:ncol(notch))){
X <- cbind(notch[,i], aachan[,i], kashiyuka[,i])
fastICA <- fastICA(X, 2, alg.typ = "parallel", fun = "logcosh", alpha = 1,
method = "R", row.norm = FALSE, maxit = 200,
tol = 0.0001, verbose = TRUE)
matplot(fastICA$S, type = "l", lty = 1)
fastICA$S[,1] = mean(fastICA$A[1,])*fastICA$S[,1]+mean(X)
fastICA$S[,2] = mean(fastICA$A[2,])*fastICA$S[,2]+mean(X)
Newperfume1 <- cbind(Newperfume1, fastICA$S[,1])
Newperfume2 <- cbind(Newperfume2, fastICA$S[,2])
}
- 79.
- 80.
- 81.
























![{fastICA} - fastICA()
fastICA(X, #[サンプル] x [観測値] の行列
n.comp, #分解するICの数
alg.typ = c(“parallel","deflation"), #ICを同時に推定するか
fun = c("logcosh","exp"), #G関数の形を決める
alpha = 1.0, #neg-entropyの形が"logcosh"のときに設定
#1~2 の間の定数
#αの値によって収束するまでの早さが変わる
method = R, #R→R言語で計算、C→C言語で計算
row.norm = FALSE, #Xが規格化されているかどうか
maxit = 200, #イテレーションの回数のリミット値
tol = 1e-04, #un-mixing matrixが収束したか決めるときのしきい値
verbose = FALSE, #結果をコンソールに表示するかどうか
w.init = NULL #非混合行列の初期値
)](https://image.slidesharecdn.com/random-140614073050-phpapp01/75/Perfume-41-2048.jpg)
![{fastICA} - fastICA()
fastICA(X, #[サンプル] x [観測値] の行列
n.comp, #分解するICの数
alg.typ = c(“parallel","deflation"), #ICを同時に推定するか
fun = c("logcosh","exp"), #G関数の形を決める
alpha = 1.0, #neg-entropyの形が"logcosh"のときに設定
#1~2 の間の定数
#αの値によって収束するまでの早さが変わる
method = R, #R→R言語で計算、C→C言語で計算
row.norm = FALSE, #Xが規格化されているかどうか
maxit = 200, #イテレーションの回数のリミット値
tol = 1e-04, #un-mixing matrixが収束したか決めるときのしきい値
verbose = FALSE, #結果をコンソールに表示するかどうか
w.init = NULL #非混合行列の初期値
)](https://image.slidesharecdn.com/random-140614073050-phpapp01/75/Perfume-42-2048.jpg)







![{fastICA} - fastICA()
fastICA(X, #[サンプル] x [観測値] の行列
n.comp, #分解するICの数
alg.typ = c(“parallel","deflation"), #ICを同時に推定するか
fun = c("logcosh","exp"), #G関数の形を決める
alpha = 1.0, #neg-entropyの形が"logcosh"のときに設定
#1~2 の間の定数
#αの値によって収束するまでの早さが変わる
method = R, #R→R言語で計算、C→C言語で計算
row.norm = FALSE, #Xが規格化されているかどうか
maxit = 200, #イテレーションの回数のリミット値
tol = 1e-04, #un-mixing matrixが収束したか決めるときのしきい値
verbose = FALSE, #結果をコンソールに表示するかどうか
w.init = NULL #非混合行列の初期値
)](https://image.slidesharecdn.com/random-140614073050-phpapp01/75/Perfume-53-2048.jpg)











![perfume データ準備
notch <- read.table("notch.dat", header = F)
aachan <- read.table("aachan.dat", header = F)
kashiyuka <- read.table("kashiyuka.dat", header = F)
X <- cbind(notch[,1], aachan[,1], kashiyuka[,1])
notch Y rot aachan Y rot kashiyuka Y rot](https://image.slidesharecdn.com/random-140614073050-phpapp01/75/Perfume-72-2048.jpg)
![主成分分析
Newperfume1 = {}
Newperfume2 = {}
!
for( i in c(1:ncol(notch))){
X <- cbind(notch[,i], aachan[,i], kashiyuka[,i])
PCA <- prcomp(X)
Newperfume1 <- cbind(Newperfume1,PCA$x[,1])
Newperfume2 <- cbind(Newperfume2,PCA$x[,2])
}
write.table(x=round(Newperfume1,6), file="Newperfume1_PCA.bvh",row.names=F,
col.names=F)
write.table(x=round(Newperfume2,6), file="Newperfume2_PCA.bvh",row.names=F,
col.names=F)
!](https://image.slidesharecdn.com/random-140614073050-phpapp01/75/Perfume-73-2048.jpg)



![独立成分分析
Newperfume1 = {}
Newperfume2 = {}
for( i in c(1:ncol(notch))){
X <- cbind(notch[,i], aachan[,i], kashiyuka[,i])
fastICA <- fastICA(X, 2, alg.typ = "parallel", fun = "logcosh", alpha = 1,
method = "R", row.norm = FALSE, maxit = 200,
tol = 0.0001, verbose = TRUE)
matplot(fastICA$S, type = "l", lty = 1)
fastICA$S[,1] = mean(fastICA$A[1,])*fastICA$S[,1]+mean(X)
fastICA$S[,2] = mean(fastICA$A[2,])*fastICA$S[,2]+mean(X)
Newperfume1 <- cbind(Newperfume1, fastICA$S[,1])
Newperfume2 <- cbind(Newperfume2, fastICA$S[,2])
}](https://image.slidesharecdn.com/random-140614073050-phpapp01/75/Perfume-78-2048.jpg)