=== OpenSim の使い方 ===
・動作確認 編
- OpenSimの正常動作を確認する
- NotePad++をインストールする
・Scaling Tool 編
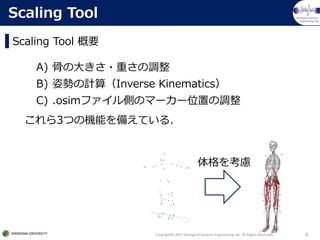
- Scaling Tool(ST) 概要
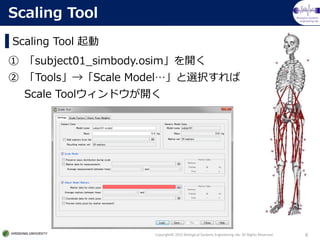
- STの起動
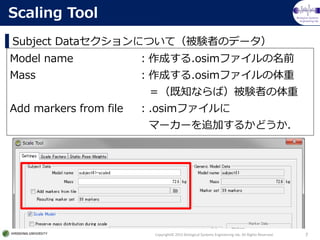
- Subject Dataセクション
- Scale Modelセクション
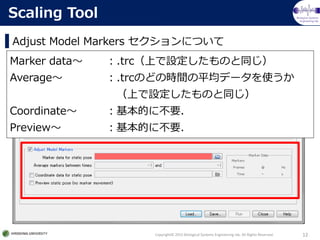
- Adjust Model Markers セクション
- Settingタブ
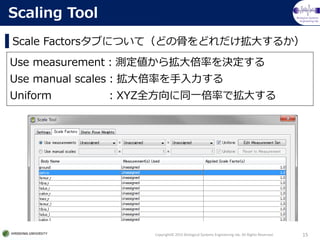
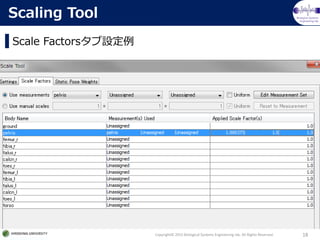
- Scale Factorsタブ
・筋骨格モデル編集 編
- NotePad++による.osimファイルの編集
- 筋,骨,マーカーデータの個別修正
・Inverse Kinematics(motファイル作成)編
- Inverse Kinematics(IK) とは
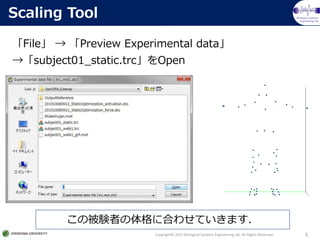
- trcファイル
- .trcファイルから.motファイルの作成
- IK実行例
- マーカーの編集,追加,削除
- .csv → .trc の変換
・Static Opitimization編
- Static Optimization (SO)とは
- SOの起動
- 設定方法
- アクチュエータ,外力の設定
- 実行例
・Plotter 編
- Plotterの起動方法
- 使用例
※※※
本スライドは,日本における筋骨格シミュレータの利用シーンの拡がりを期待して,広島大学工学研究科生体システム論研究室栗田グループの研究室内チュートリアルで利用された資料を一部まとめて公開したものです.
本スライドの作成に主に関わってくれた,岸下優介くん,村田拓也くんに感謝します.
※※※




![Biological Systems
Engineering lab.
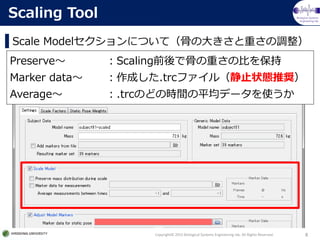
Scale Modelセクションについて(骨の大きさと重さの調整)
Copyright© 2015 Biological Systems Engineering lab. All Rights Reserved. 10
Scaling Tool
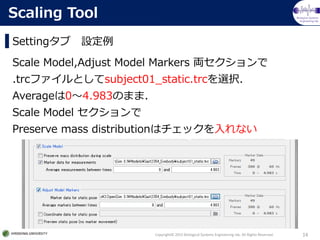
Preserve~ :Scaling前後で骨の重さの比を保持
Marker data~ :作成した.trcファイル(静止状態推奨)
Average~ :.trcのどの時間の平均データを使うか
Scaling前
Scaling後(Preserve~ ONの時) Scaling後(Preserve~ OFFの時)
例:組み込みのモデル を,体重100 [kg]の被験者にScaling
距骨重量 / 踵骨重量 0.08
体重 72.6 [kg]
距骨重量 / 踵骨重量 0.08
体重 100 [kg]
距骨重量 / 踵骨重量 0.0679
体重 125.22 [kg]](https://image.slidesharecdn.com/opensimscaling20160118-160502071046/85/OpenSim-scaling-10-320.jpg)