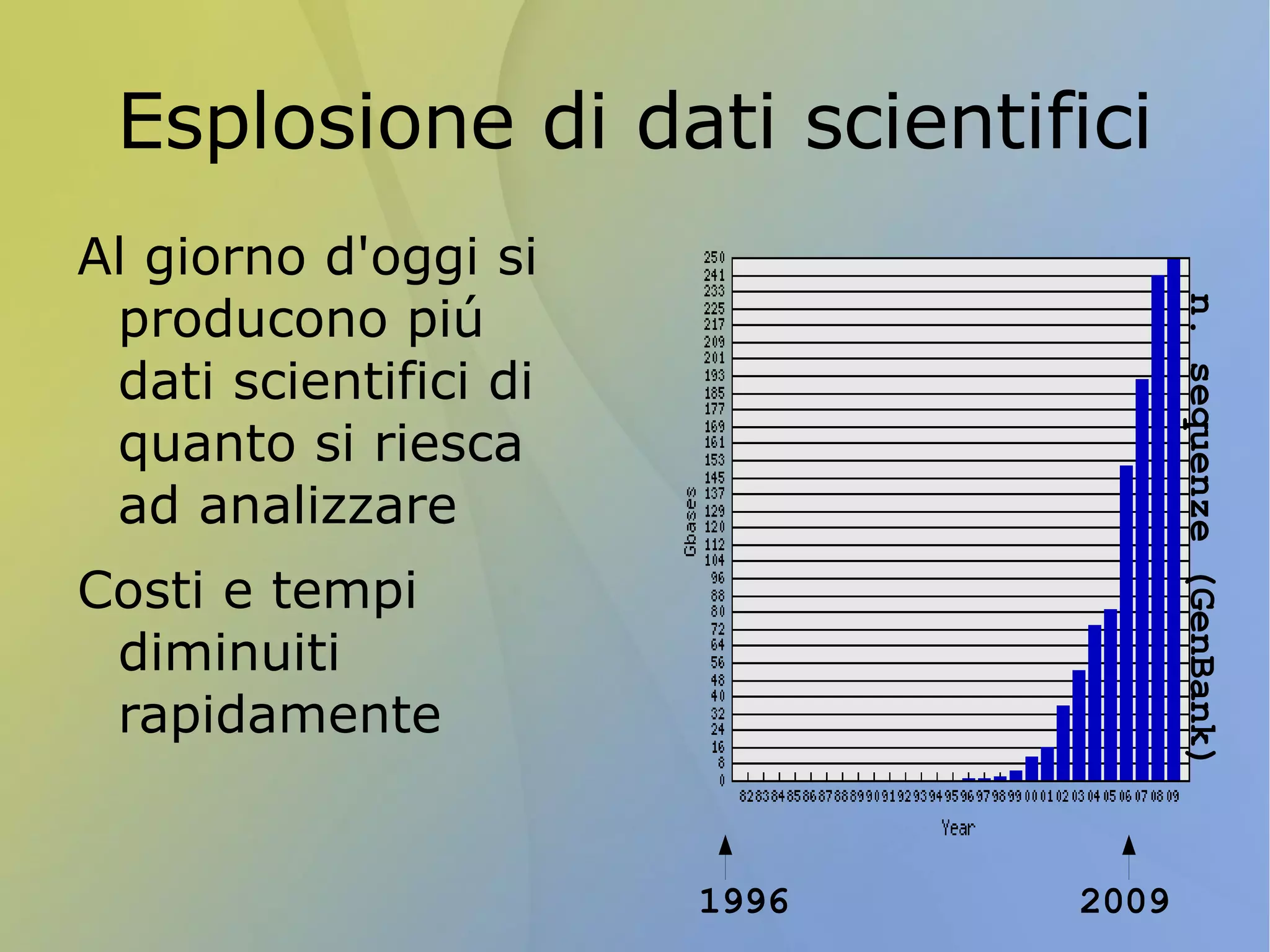
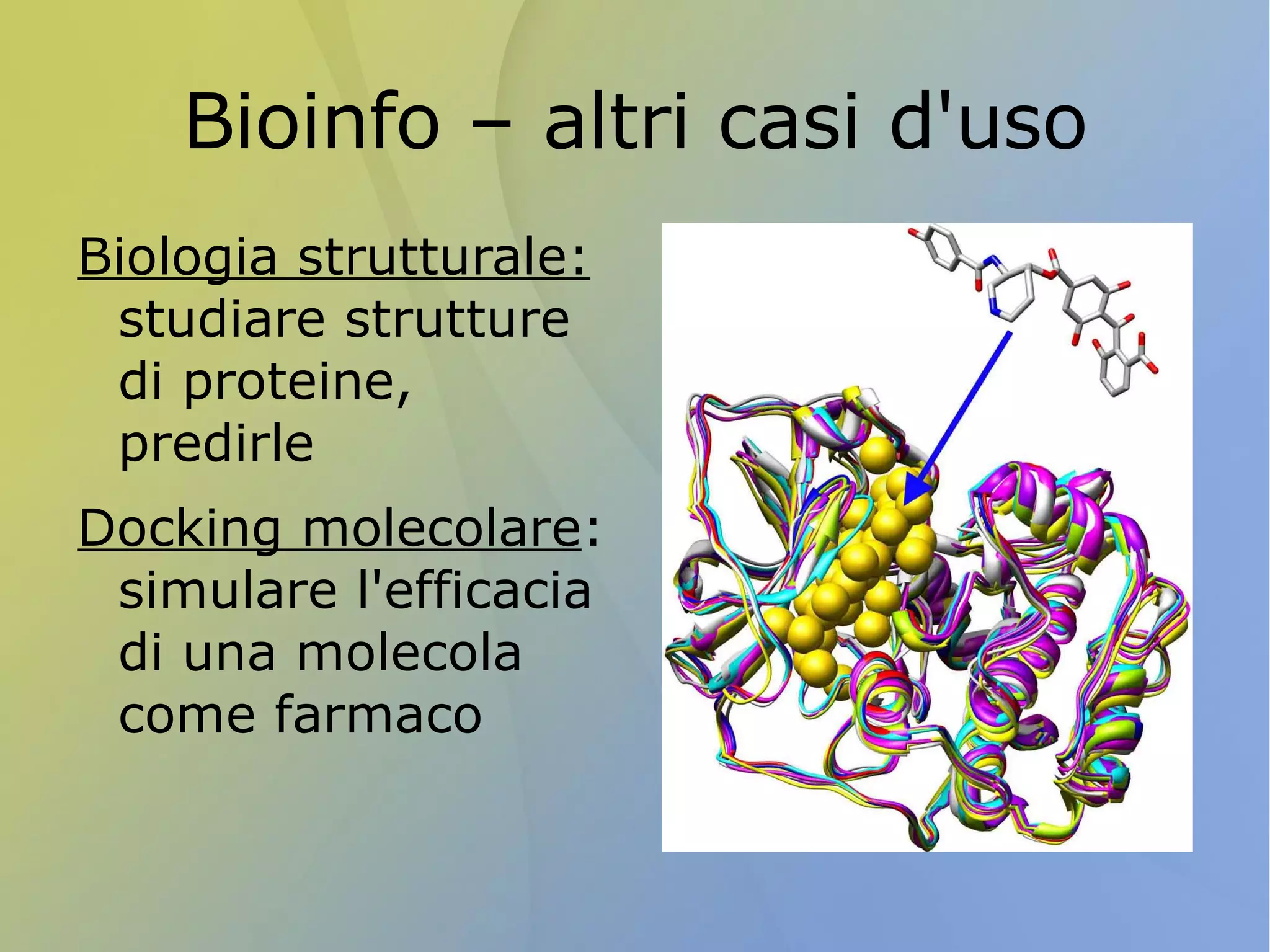
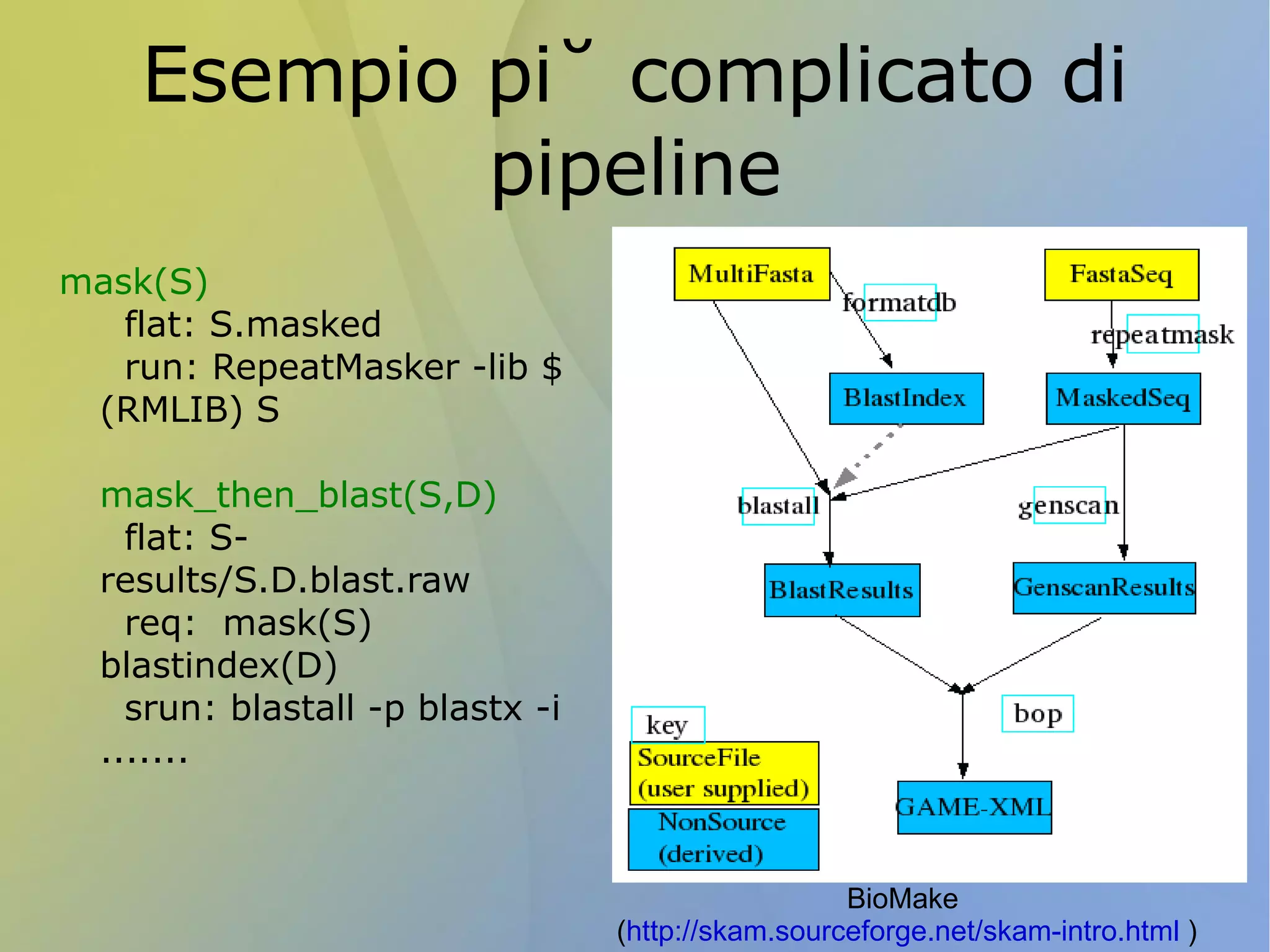
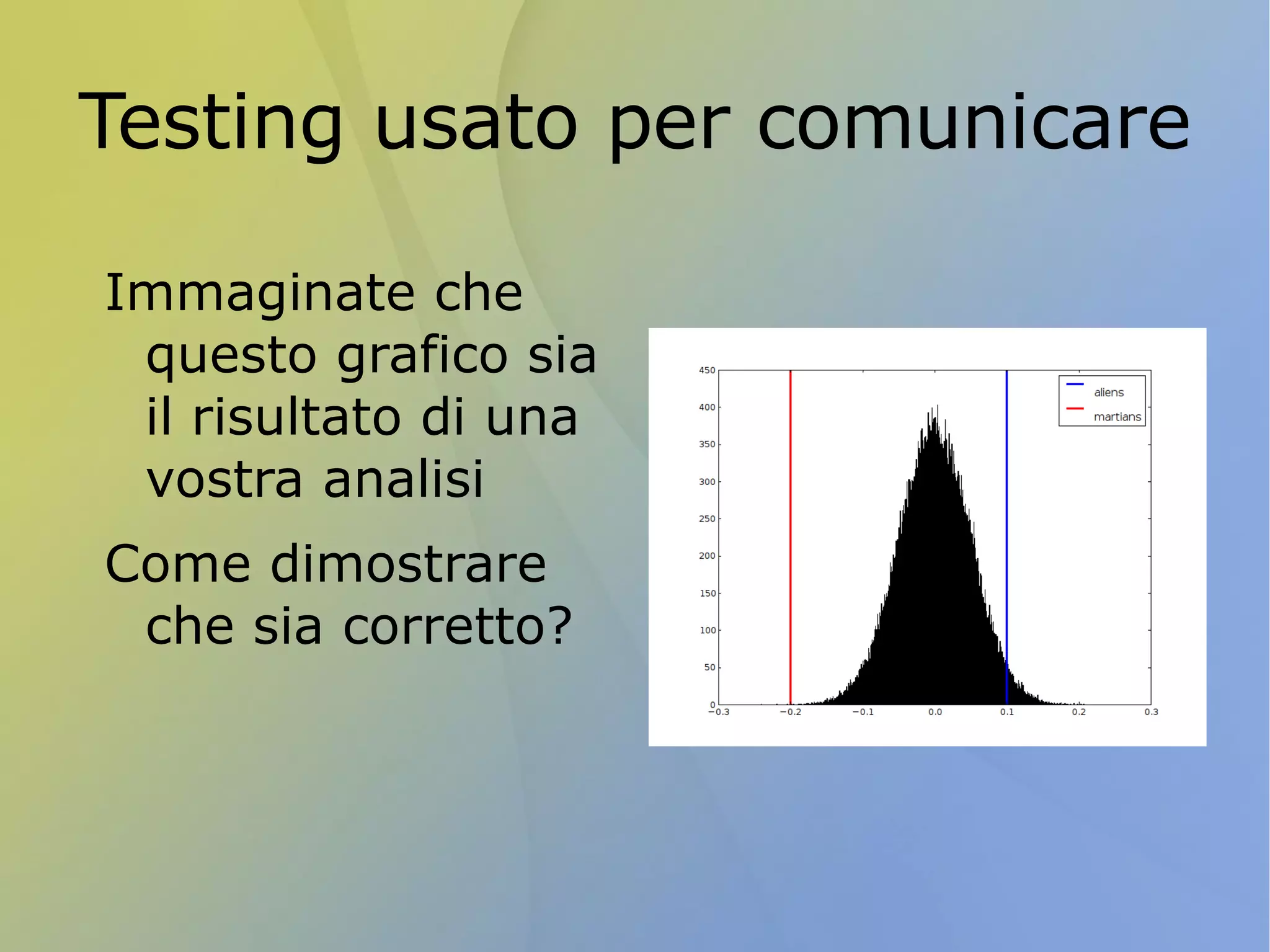
La bioinformatica è una disciplina che analizza e gestisce i dati scientifici attraverso metodi computazionali, essenziale per trattare la vasta quantità di dati generati dalla ricerca, come nel caso del sequenziamento del genoma umano. Python è uno dei linguaggi più utilizzati in questo campo grazie alla sua sintassi chiara, all'accesso a librerie specifiche e alla sua versatilità nel programmare script per l'analisi dei dati biologici. Il lavoro di un bioinformatico include l'integrazione dei dati, la programmazione orientata agli oggetti e il testing, sfruttando strumenti e librerie open source per facilitare le operazioni necessarie.











































![Programmazione a oggetti - esempio genes = { 'gene1' : { 'position' : 10000, 'chromosome': 11, 'sequence' : 'GTAGCCTGATGAACGGGCTAGCATGC....' , 'transcripts' : { 'transcript1' : [......], 'transcript2' : [......], }, }, 'gene1' : { 'position' : ...........}, ..... } def get_subseq(genes, geneid, start, end): ''' get a subsequence of a gene, given a dictionary of gene annotations, a gene id, and start/end position ''' pass](https://image.slidesharecdn.com/pycon-090511033133-phpapp01/75/Pycon-52-2048.jpg)