More Related Content
PPTX
Pythonで機械学習を自動化 auto sklearn PPTX
Raspberry PiとPythonでできること PDF
PPTX
PDF
PPTX
PPT
PPTX
210526 Power Automate Desktop Python What's hot
PPTX
PDF
S01 t1 tsuji_pylearn_ut_01 PPTX
PDF
PPTX
PDF
Lighting talk chainer hands on PPTX
PDF
linterとprettierというコード砂漠に緑をもたらす救世主 ODP
Viewers also liked
PDF
PDF
PDF
Julia 100 exercises #JuliaTokyo PPTX
PDF
Juliaで学ぶ Hamiltonian Monte Carlo (NUTS 入り) PDF
PDF
第6章 2つの平均値を比較する - TokyoR #28 PDF
PDF
数学プログラムを Haskell で書くべき 6 の理由 PPTX
非負値行列分解の確率的生成モデルと�多チャネル音源分離への応用 (Generative model in nonnegative matrix facto... PDF
On the benchmark of Chainer PDF
深層学習ライブラリの環境問題Chainer Meetup2016 07-02 PDF
ヤフー音声認識サービスでのディープラーニングとGPU利用事例 PDF
俺のtensorが全然flowしないのでみんなchainer使おう by DEEPstation PDF
マシンパーセプション研究におけるChainer活用事例 PDF
Chainer Update v1.8.0 -> v1.10.0+ PDF
PDF
NVIDIA 更新情報: Tesla P100 PCIe/cuDNN 5.1 PPTX
PDF
「はじめてでもわかる RandomForest 入門-集団学習による分類・予測 -」 -第7回データマイニング+WEB勉強会@東京 Similar to Julia0.3でランダムフォレスト
PDF
PDF
Lispmeetup #50 cl-random-forest: Common Lispによるランダムフォレストの実装 PPTX
PPTX
32bit Windowsで頑張るRandom Forest PDF
「樹木モデルとランダムフォレスト-機械学習による分類・予測-」-データマイニングセミナー PPTX
PDF
ランダムフォレストとそのコンピュータビジョンへの応用 PPTX
PDF
シリーズML-03 ランダムフォレストによる自動識別 PDF
ライブラリ紹介:Fertilized Forests More from Atsushi Hayakawa
PDF
PDF
PDF
dataclassとtypehintを使ってますか? PDF
トライアスロンとgepuro task views V2.0 Japan.R 2018 PPTX
PPTX
Analyze The Community Of Tokyo.R PPTX
PDF
PDF
simputatoinで欠損値補完 - Tokyo.R #65 PDF
PPTX
PPTX
Rstudio上でのパッケージインストールを便利にするaddin4githubinstall PDF
PDF
Splatoon界での壮絶な戦い&Japan.Rの宣伝 PDF
PDF
PDF
nginxのログを非スケーラブルに省メモリな方法で蓄積する PDF
PDF
PDF
Julia0.3でランダムフォレスト
- 1.
- 2.
- 3.
DecisionTree.jl
● bensadeghiさんが開発
○ 他には、
■MineSweeperSolver.jl
■ METADATA.jl
■ pyplot.jl
○ なども開発に関わっている。
● MITライセンス
● 決定木はID3 algorithmで実装されている。
CARTで実装されたランダムフォレストは、
@bicycle1885さんが開発している。
https://github.com/bicycle1885/RandomForests.jl
- 4.
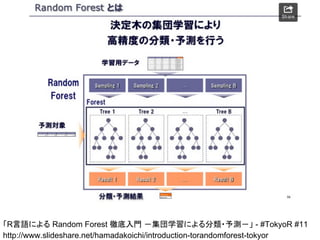
「R言語による Random Forest徹底入門 -集団学習による分類・予測-」 - #TokyoR #11
http://www.slideshare.net/hamadakoichi/introduction-torandomforest-tokyor
- 5.
- 6.
- 7.
- 8.
- 9.
- 10.
ソースコードを覗いてみる(分類)
function build_forest(labels::Vector, features::Matrix,nsubfeatures::Integer,
ntrees::Integer, partialsampling=0.7)
partialsampling = partialsampling > 1.0 ? 1.0 : partialsampling
Nlabels = length(labels)
Nsamples = int(partialsampling * Nlabels)
forest = @parallel (vcat) for i in [1:ntrees]
inds = rand(1:Nlabels, Nsamples)
build_tree(labels[inds], features[inds,:], nsubfeatures)
end
return Ensemble([forest])
end
● バージョン0.2の頃に合わせているのか、データの持ち方がVectorとMtrixになってる。
● arrayとの違いはなんだろうか?
● 引数は、nsubfeatrues, ntrees, partialsamplingの3つ
● partialsamplingは、指定しなくても動作する
● 並列処理に対応しているっぽい
- 11.
ソースコードを覗いてみる(回帰)
function build_forest{T<:FloatingPoint,U<:Real}(labels::Vector{T},features::Matrix{U},
nsubfeatures::Integer, ntrees::Integer,maxlabels=0.5, partialsampling=0.7)
partialsampling = partialsampling > 1.0 ? 1.0 : partialsampling
Nlabels = length(labels)
Nsamples = int(partialsampling * Nlabels)
forest = @parallel (vcat) for i in [1:ntrees]
inds = rand(1:Nlabels, Nsamples)
build_tree(labels[inds], features[inds,:], maxlabels, nsubfeatures)
end
return Ensemble([forest])
end
● Javaでいうオーバーライドが出来るのかな?
● 関数を宣言した直後に型を指定?
● パラメータにmaxlabelsが追加されている。葉あたりの平均サンプル数を指定する
- 12.





![データの準備
iris = dataset("datasets", "iris")
features = array(iris[:, 1:4])
labels = array(iris[:, 5])](https://image.slidesharecdn.com/julia0-140705040027-phpapp02/85/Julia0-3-7-320.jpg)


![ソースコードを覗いてみる(分類)
function build_forest(labels::Vector, features::Matrix, nsubfeatures::Integer,
ntrees::Integer, partialsampling=0.7)
partialsampling = partialsampling > 1.0 ? 1.0 : partialsampling
Nlabels = length(labels)
Nsamples = int(partialsampling * Nlabels)
forest = @parallel (vcat) for i in [1:ntrees]
inds = rand(1:Nlabels, Nsamples)
build_tree(labels[inds], features[inds,:], nsubfeatures)
end
return Ensemble([forest])
end
● バージョン0.2の頃に合わせているのか、データの持ち方がVectorとMtrixになってる。
● arrayとの違いはなんだろうか?
● 引数は、nsubfeatrues, ntrees, partialsamplingの3つ
● partialsamplingは、指定しなくても動作する
● 並列処理に対応しているっぽい](https://image.slidesharecdn.com/julia0-140705040027-phpapp02/85/Julia0-3-10-320.jpg)
![ソースコードを覗いてみる(回帰)
function build_forest{T<:FloatingPoint,U<:Real}(labels::Vector{T},features::Matrix{U},
nsubfeatures::Integer, ntrees::Integer, maxlabels=0.5, partialsampling=0.7)
partialsampling = partialsampling > 1.0 ? 1.0 : partialsampling
Nlabels = length(labels)
Nsamples = int(partialsampling * Nlabels)
forest = @parallel (vcat) for i in [1:ntrees]
inds = rand(1:Nlabels, Nsamples)
build_tree(labels[inds], features[inds,:], maxlabels, nsubfeatures)
end
return Ensemble([forest])
end
● Javaでいうオーバーライドが出来るのかな?
● 関数を宣言した直後に型を指定?
● パラメータにmaxlabelsが追加されている。葉あたりの平均サンプル数を指定する](https://image.slidesharecdn.com/julia0-140705040027-phpapp02/85/Julia0-3-11-320.jpg)
