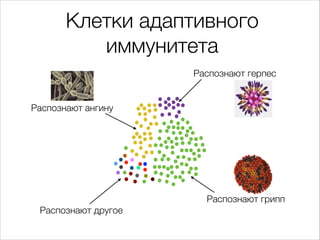
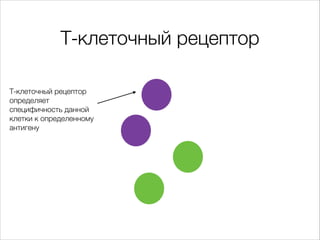
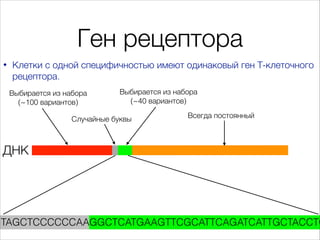
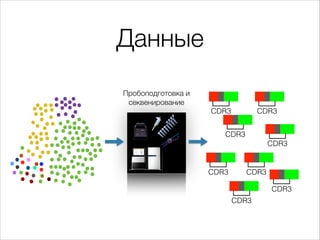
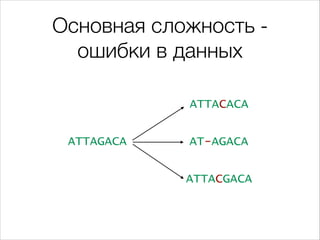
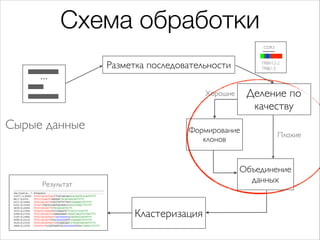
Документ описывает первичный анализ данных иммунома человека, сосредотачиваясь на клетках адаптивного иммунитета и их способности распознавать различные антигены. Он детализирует механизмы работы т-клеточных рецепторов, включая их генетическую специфичность и разнообразие. Также документ упоминает о методах секвенирования и обработке данных для восстановления информации о клональных популяциях клеток.











































![Прототип
Clones
DataSets
ATTAGACA
Int[] datasets
ID
Name
{1,3,30,40}
1
Control_1
40
String CDR3
Flu_1
Indexed using GiST or GIN](https://image.slidesharecdn.com/ya-131111144847-phpapp01/85/Ya-mi-tcr-final-50-320.jpg)


