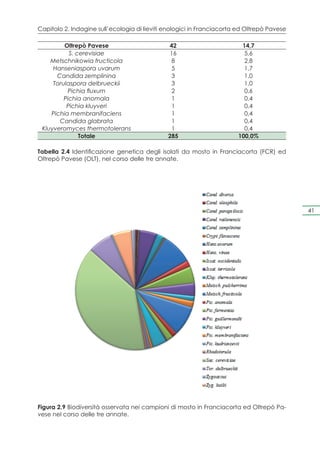
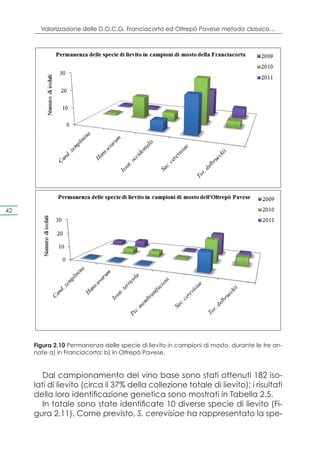
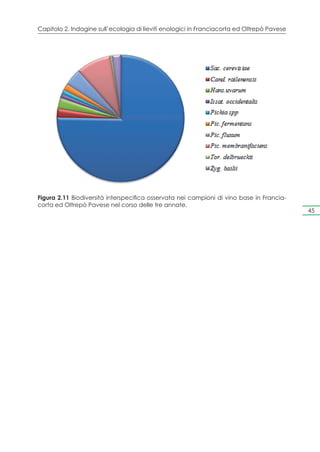
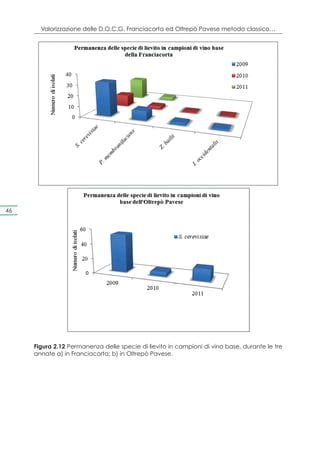
Il documento presenta un progetto di ricerca volto alla valorizzazione dei vini spumanti D.O.C.G. Franciacorta e Oltrepò Pavese attraverso l'uso di lieviti autoctoni, mirato a migliorare la qualità e a certificare l'origine territoriale dei prodotti. La Lombardia, con la sua alta produzione di vino spumante, sta affrontando sfide legate alle trasformazioni del mercato e alla concorrenza internazionale. Il progetto mira a preservare le varietà locali di lieviti e a sviluppare un protocollo analitico per garantire la qualità e l'autenticità dei vini lombardi.























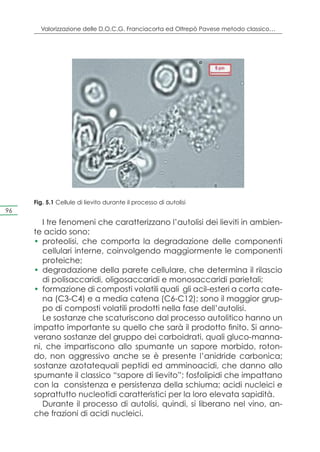
![Capitolo 2. Indagine sull’ecologia di lieviti enologici in Franciacorta ed Oltrepò Pavese
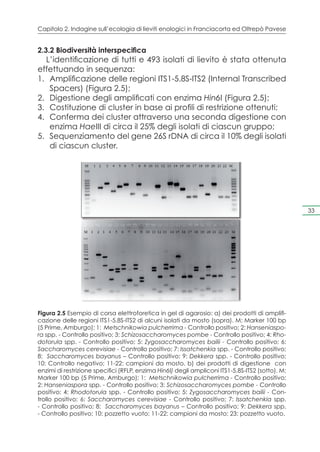
Figura 2.2 Air Sampler “MAS 100 Eco” di VWR International utilizzato per il campiona-
mento.
La piastra viene posta nell’apposito supporto, priva di coper-
chio, con il terreno colturale rivolto verso la parte forata dell’ap-
parecchio. Da quest’ultima, costruita in acciaio e disinfettata
prima di ciascun campionamento con alcol etilico, si effettua l’a-
spirazione dell’aria che andrà ad impattare, con quanto in essa
eventualmente disperso (microrganismi, spore) sul terreno nutriti- 23
vo della piastra. Questo campionatore permette il prelevamen-
to di precisi volumi, quindi l’ottenimento di risultati non soltanto
qualitativi, ma anche quantitativi. Durante il campionamento lo
strumento veniva trasportato manualmente tra i filari mantenen-
dolo ad una distanza di circa 30 cm dalle piante e di 1-1,20 m
dal suolo (in corrispondenza dei grappoli). Per ogni anno, gli stessi
vigneti (tredici in Franciacorta e undici in Oltrepò Pavese) sono
stati analizzati e, in ogni vigneto, due aliquote da 100 e 500 litri
di aria ciascuno, replicate per due volte sono stati raccolti per le
successive analisi microbiologiche. L’isolamento dei lieviti è stato
effettuato utilizzando il terreno colturale YEPD [1% (w/v) Estratto di
lievito, 2% (w/v) Peptone, 2% (w/v) Glucosio] addizionato con 2%
agar e modificato nelle seguenti caratteristiche: pH 3,6, 200 mg/L
K2S2O5 e 100 mg/L cloramfenicolo. Le piastre, ottenute dal cam-
pionamento dell’aria, sono state incubate a 25°C in condizioni
anaerobiche (Microbiologie Anaerocult®A; Merck, Darmstadt,
Germania) per 5 giorni. Questa modifica è stata effettuata per
evitare la crescita di muffe.](https://image.slidesharecdn.com/qdr148-130128095911-phpapp02/85/VALORIZZAZIONE-DELLE-D-O-C-G-24-320.jpg)




















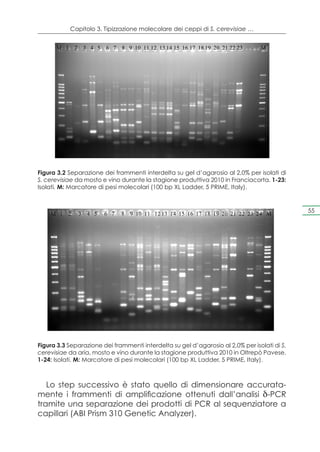
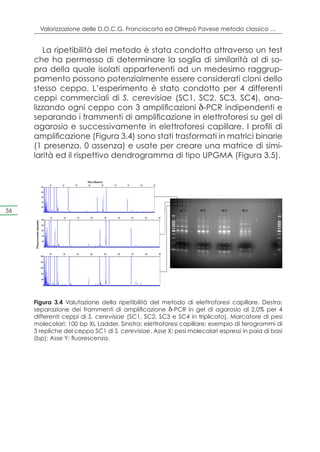
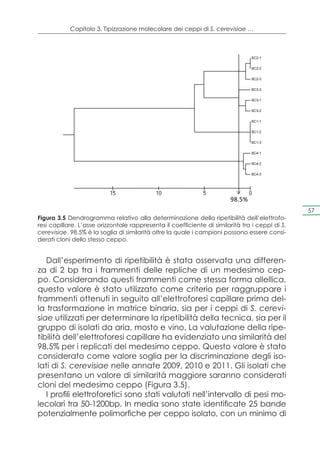


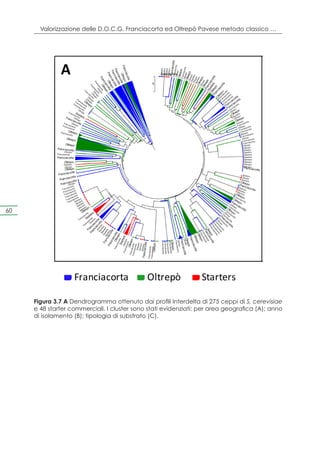
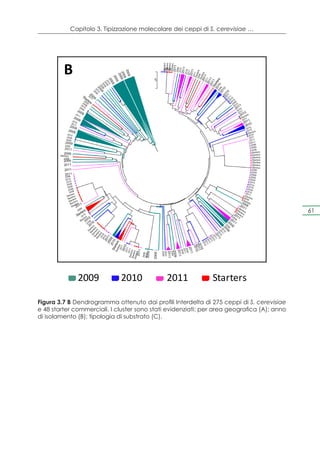
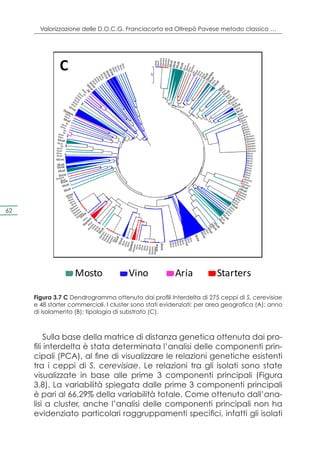



![Capitolo 4. Selezione dei ceppi e prove di microvinificazione
posto a sterilizzazione per eliminare eventuali microrganismi che
avrebbero potuto condizionare i risultati. Ciascuna coltura di
lievito è stata inoculata ad una concentrazione pari a 106 UFC/
ml, a partire da una pre-coltura sviluppatasi nello stesso tipo di
mosto per 48 ore. La prova è stata condotta alla temperatura di
18 °C.
Determinazione dei caratteri tecnologici
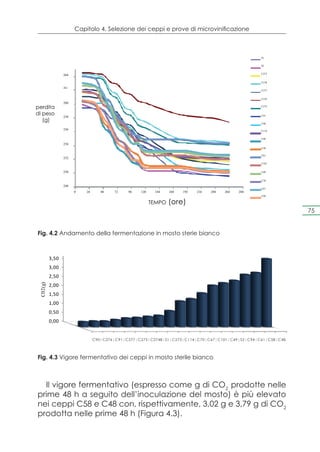
Per determinare il potere ed il vigore fermentativo dei ceppi
impiegati nell’analisi è stata misurata ogni giorno la perdita di
peso delle provette Erlenmeyer fino all’ottenimento di un peso
costante per due giorni consecutivi. Il vigore fermentativo è sta-
to espresso in termini di grammi di CO2 prodotte nelle prime 48
ore, seguenti l’inoculo del mosto, mentre il potere fermentativo
è stato espresso come % v/v di etanolo prodotto, impiegando la
seguente formula:
[ ( Δpeso (g) / 44 (g/mol CO2 ) x 46,7 (g/mol EtOH) ] / 0,789 (g/ml EtOH) 67
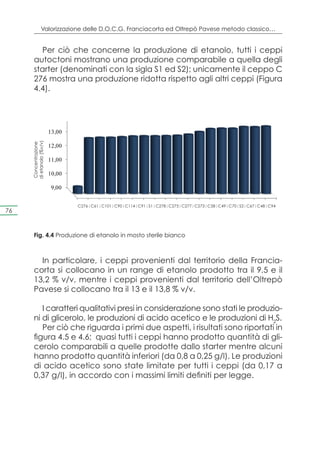
Determinazione dei caratteri qualitativi
I caratteri qualitativi valutati sono stati: produzione di glicerolo,
produzione di acido acetico e produzione di H2S.
Al termine della fermentazione, aliquote di campioni di vino
sono state centrifugate (Hettich zentrifugen, rotina 380r, Ger-
many) a 3500 g e il surnatante è stato sottoposto ad analisi; per
ciascun campione, valutato in tre repliche, la produzione di gli-
cerolo e di acido acetico sono state misurate attraverso l’impie-
go di kit enzimatici (kit 148270 e 148261, rispettivamente; Boehrin-
ger-Mannheim, Germany).
La purezza della fermentazione è stata espressa in termini di
grammi di acido acetico/100 ml di etanolo.
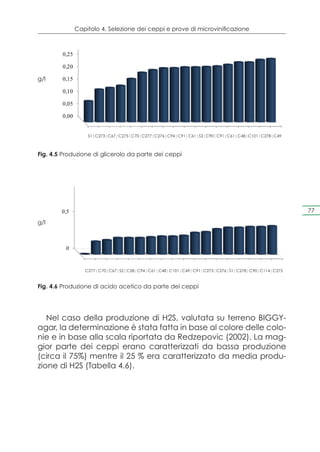
Per ciò che riguarda la produzione di H2S, tutti i ceppi sono stati
piastrati su terreno Bismuth Sulfite Glucose Glycine Yeast agar-
BiGGY (BD, France). In questo terreno il colore delle colonie è
determinato dalla riduzione del solfito a solfuro che legandosi al
bismuto, provoca cambiamento di colore delle colonie (da bei-
ge a nero). In accordo con Redzepovic (2002) il grado di colo-
razione, associato alla crescita delle colonie di lievito in BIGGY-
agar, è determinato dalla seguente scala:](https://image.slidesharecdn.com/qdr148-130128095911-phpapp02/85/VALORIZZAZIONE-DELLE-D-O-C-G-68-320.jpg)








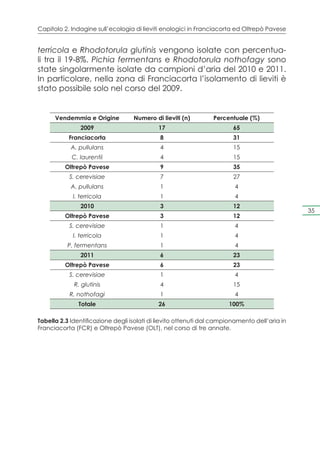
![Valorizzazione delle D.O.C.G. Franciacorta ed Oltrepò Pavese metodo classico…
Per ciò che riguarda la gradevolezza dei ceppi provenienti
dall’Oltrepò Pavese, sono stati il ceppo C48 e il C58 a mostrarsi
significativamente diversi dagli altri [intervallo di confidenza 95%,
(p<0,05].
Nel caso dell’intensità, per ciò che riguarda il territorio della
Franciacorta, i campioni significativamente diversi sono stati il C
276 e il C91 mentre per il territorio dell’Oltrepò Pavese, il campio-
ne più intenso è stato il C49.
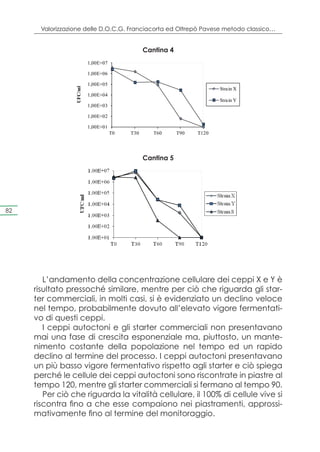
4.8 Risultati delle prove di rifermentazione
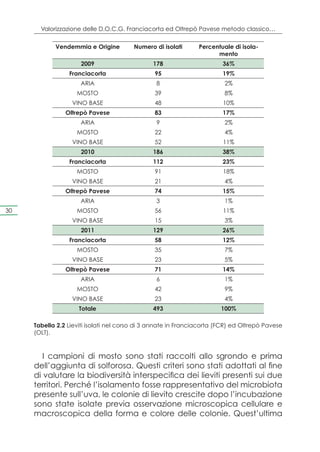
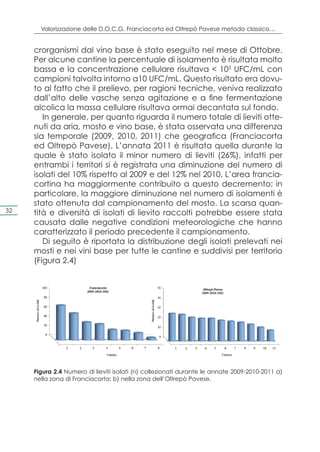
Sulla base dei risultati descritti precedentemente, sono stati se-
lezionati i ceppi autoctoni per le prove di rifermentazione dei vini
base 2011 denominati rispettivamente X (C273 in Franciacorta,
C58 in Oltrepò Pavese) e Y (C275 in Franciacorta, C48 in Oltre-
pò Pavese). Questi sono stati impiegati in 5 cantine del territorio
80 della Franciacorta e in 3 cantine dell’Oltrepò pavese. Gli star-
ter commerciali impiegati in queste prove (denominati ceppi S)
sono stati impiegati come controllo.
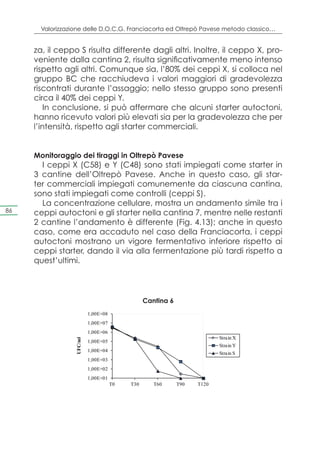
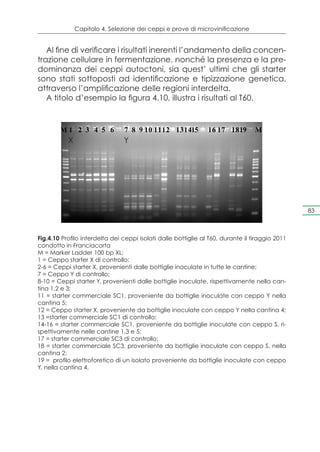
Monitoraggio dei tiraggi in Franciacorta
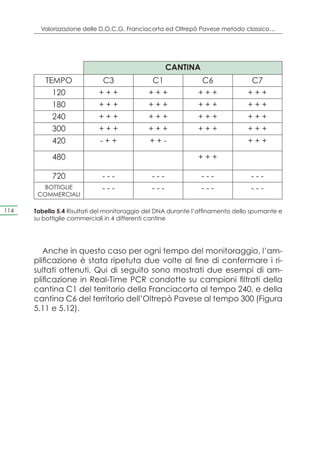
La rifermentazione è stata monitorata a differenti intervalli di
tempo a partire dal T0, che corrisponde al primo giorno di imbot-
tigliamento del vino; da ciascuna cantina sono state prelevate
2 bottiglie per ciascun ceppo test su cui sono state condotte le
seguenti analisi: valutazione della concentrazione cellulare, va-
lutazione della vitalità cellulare ed identificazione genetica.
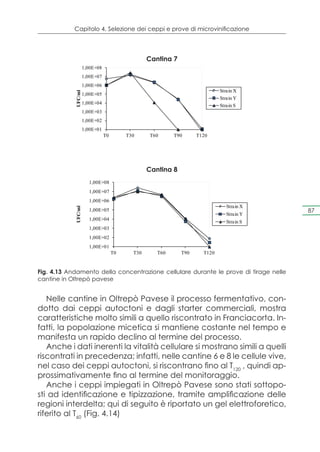
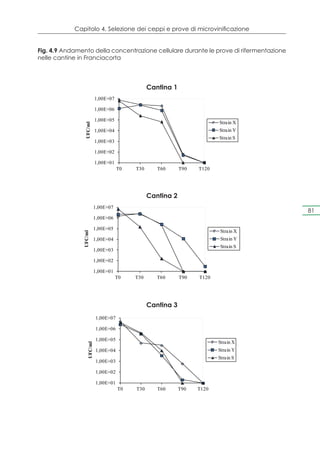
Per ciò che riguarda la concentrazione cellulare, essa è stata
valutata tramite piastramenti in terreno WL, in duplice copia. I
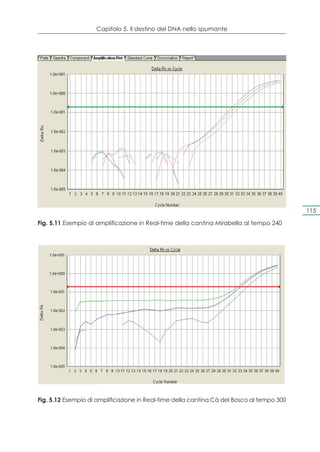
risultati per ciascuna cantina, della Franciacorta, sono riportati in
Figura 4.9.](https://image.slidesharecdn.com/qdr148-130128095911-phpapp02/85/VALORIZZAZIONE-DELLE-D-O-C-G-81-320.jpg)

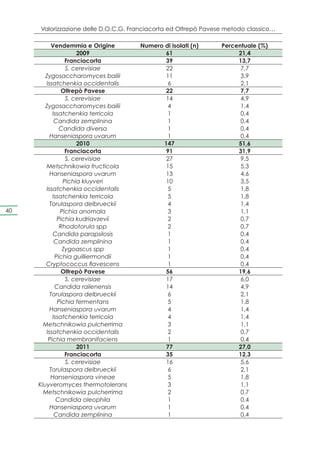
![Capitolo 4. Selezione dei ceppi e prove di microvinificazione
Pleasantness of Franciacorta wine samples at 6 months of tirage proves
10
9
8
7
C
6 BC
punteggio
ABC
score
5 AB
4 A
3
2
1
0
S2 Y2 X1 Y3 S3 Y4 Y1 X5 Y5 S1 X2 X3 X4 X5 S4
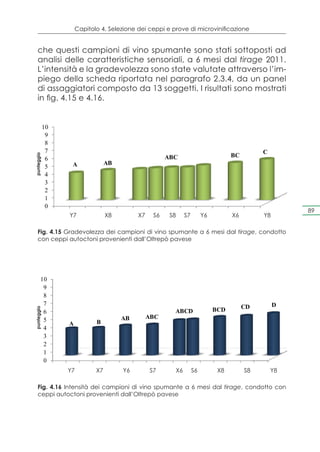
Fig. 4.11 Gradevolezza dei campioni di vino spumante a 6 mesi dal tirage, condotto
con ceppi autoctoni provenienti dalla Franciacorta
Intensity of Franciacorta wine samples at 6 months of tirage proves
10
9
8
7 BCD CD D 85
ABCD
punteggio
6 AB ABC
A
score
5
4
3
2
1
0
X2 Y2 S3 Y1 Y3 Y4 X1 X4 S5 X3 X5 Y5 S2 S1 S4
Fig. 4.12 Intensità dei campioni di vino spumante a 6 mesi dal tirage, condotto con
ceppi autoctoni provenienti dalla Franciacorta
Per ciò che riguarda la gradevolezza, per valutare le differenze
significative tra i vari campioni, è stato impiegato il metodo LSD
[intervallo di confidenza del 95%, (p<0,05].
Dall’analisi dei risultati di gradevolezza è stato evidenziato che
unicamente il ceppo S, proveniente dalla cantina 4 risulta diffe-
rente dagli altri. Infatti, gli altri ceppi si distribuiscono in un gruppo
omogeneo (A, B e C).
I valori di intensità, invece, evidenziano un gruppo omogeneo
(A, B, C e D), e in accordo con i risultati inerenti alla gradevolez-](https://image.slidesharecdn.com/qdr148-130128095911-phpapp02/85/VALORIZZAZIONE-DELLE-D-O-C-G-86-320.jpg)