More Related Content
PDF
LODチャレンジ Japan 2013 審査員特別賞 環境LOD賞 PDF
LODチャレンジ Japan 2013 審査員特別賞 ベストプレゼンテーション賞 PDF
LODチャレンジ Japan 2013 スポンサー賞 ヒューマンセントリック賞 PDF
LODチャレンジ Japan 2013 基盤技術部門 優秀賞 PDF
LODチャレンジ Japan 2013 基盤提供パートナー賞 Remote Testkit賞 PDF
LODチャレンジ Japan 2013 基盤技術部門 最優秀賞 PDF
20210922応用生態工学会データベース自由集会 PDF
What's hot
PDF
環境科学の研究者から見たオープンデータの利点と現状 PDF
生物多様性情報の標準データフォーマットDarwin Core Archiveと生態学データに適合させる拡張形式"Sample-based Data" PDF
PDF
PDF
20201125バイオロギング研究会ワークショップ【オープンデータ】 PDF
20201003生物多様性情報を使い倒す~GBIF入門~ PDF
LODチャレンジ Japan 2013 アプリケーション部門 最優秀賞 PDF
2016/12/21NISTEPホライズン・セミナースライド PDF
キラリマッピングプロジェクトOsm実習20141018 Viewers also liked
PDF
LODチャレンジ Japan 2013 スポンサー賞 日本マイクロソフト賞 PDF
LODチャレンジ Japan 2013 ビジュアライゼーション部門 最優秀賞 PDF
LODチャレンジ Japan 2013 スポンサー賞 goo賞 PDF
LODチャレンジ Japan 2013 審査員特別賞 ベストファシリテータ賞 PDF
LODチャレンジ Japan 2013 データ提供パートナー賞 公益財団法人横浜市芸術文化振興財団賞 PDF
LODチャレンジ Japan 2013 審査員特別賞 子育てライフ賞 PDF
LODチャレンジ Japan 2013 審査員特別賞 学生奨励賞2 PDF
LODチャレンジ Japan 2013 データセット部門 最優秀賞 PDF
LODチャレンジ Japan 2013 スポンサー賞 データクリエータ賞 PDF
LODチャレンジ Japan 2013 データセット部門 優秀賞 PDF
LODチャレンジ Japan 2013 データ提供パートナー賞 Statistics Japan賞 PDF
LODチャレンジ Japan 2013 審査員特別賞 データカタログ賞 PDF
LODチャレンジ Japan 2013 審査員特別賞 学生奨励賞1 PDF
LODチャレンジ Japan 2013 審査員特別賞 公共LOD賞 PDF
LODチャレンジ Japan 2013 アイディア部門 優秀賞 PDF
LODチャレンジ Japan 2013 審査員特別賞 LOD推進賞 PDF
LODチャレンジ Japan 2013 審査員特別賞 チャレンジデー賞 PDF
LODチャレンジ Japan 2013 基盤提供パートナー賞 アプリクリエータ賞 PDF
LODチャレンジ Japan 2013 データ提供パートナー賞 アイデア無限賞 More from Linked Open Dataチャレンジ実行委員会
PDF
三原さん20150926 lodチャレンジ2015キックオフ PDF
栗原先生20150926 lodチャレンジ2015キックオフ PDF
PDF
PDF
OpenData_DataCurator_20140927 PPTX
20140927 lodチャレンジ@キックオフイベントlast PDF
LODチャレンジ Japan 2013 アプリケーション部門 優秀賞 PDF
LODチャレンジ Japan 2013 アイディア部門 最優秀賞 LODチャレンジ Japan 2013 審査員特別賞 ライフサイエンス賞
- 1.
- 2.
◆受賞者◆
込山悠介, 番野雅城, ガル・サード,鑓水優行,
植木快, 清水謙多郎
◆推薦の言葉◆
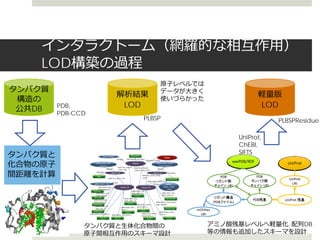
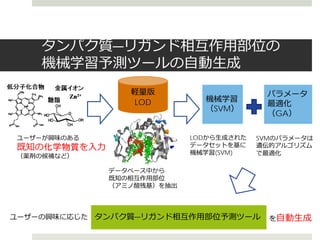
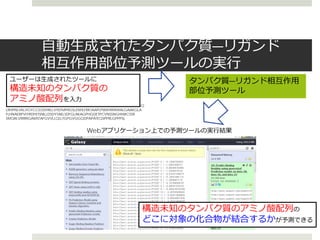
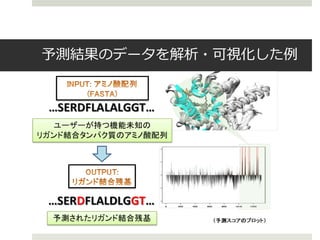
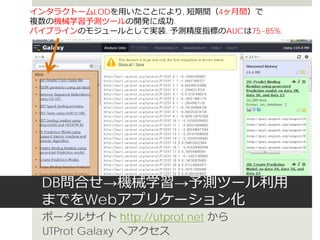
創薬における重要課題のひとつであるリガンドの結合部位予測に
向け、構造生物学の成果データをLODとして再構成することで、
更に幅広い用途に利活用できるデータベースを構築しました。
ライフサイエンス分野で現在進められているRDFによるデータ
ベースの統合化推進に大きく貢献することが今後期待されます。
- 3.
- 4.
- 5.
- 6.
- 7.
- 8.
- 9.
- 10.
謝辞
国立情報学研究所
Thiswork was supported by JSPS KAKENHI Grant
Number 12J07771 and 23300109.
This research was supported by JST, NBDC.