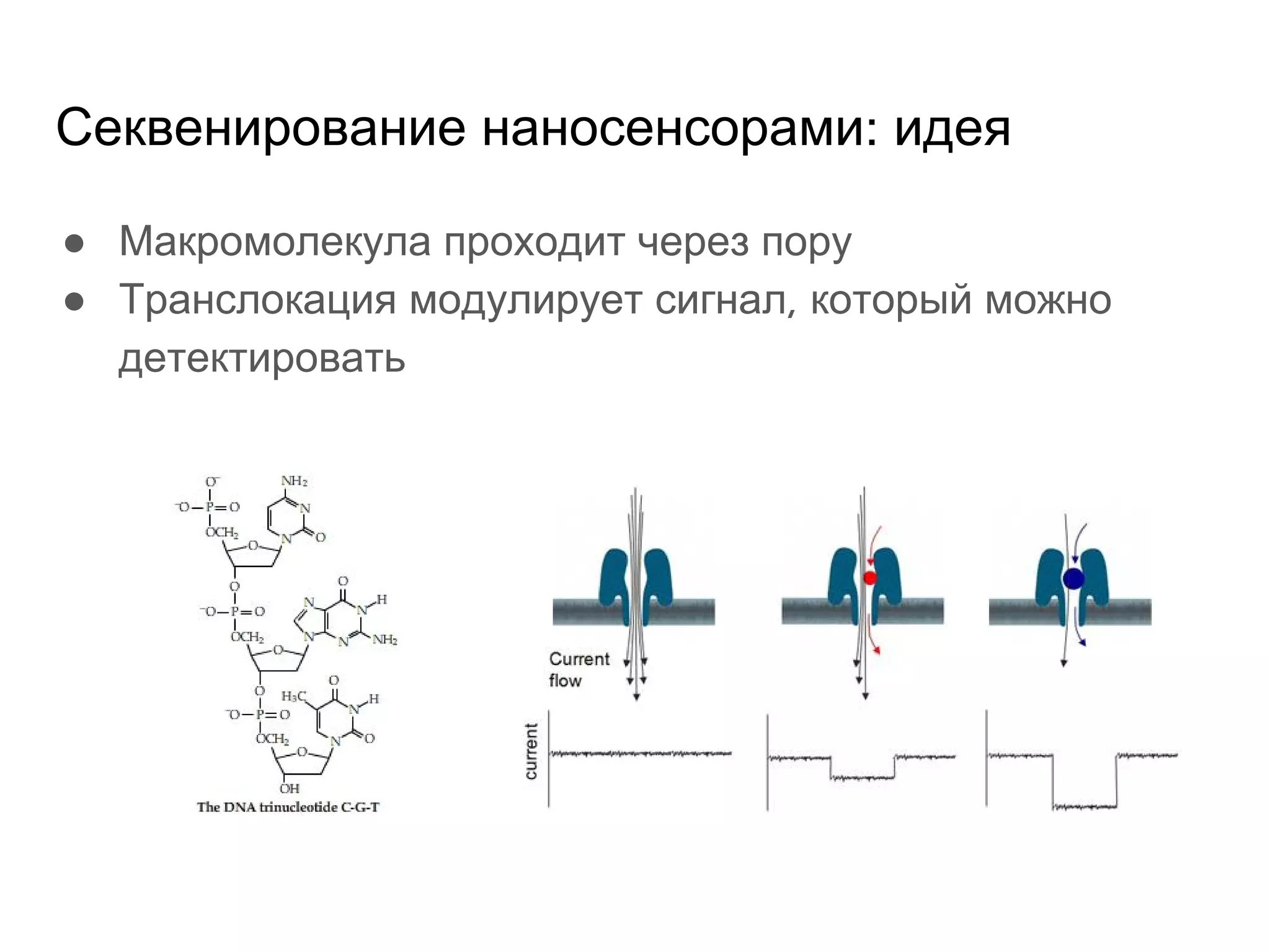
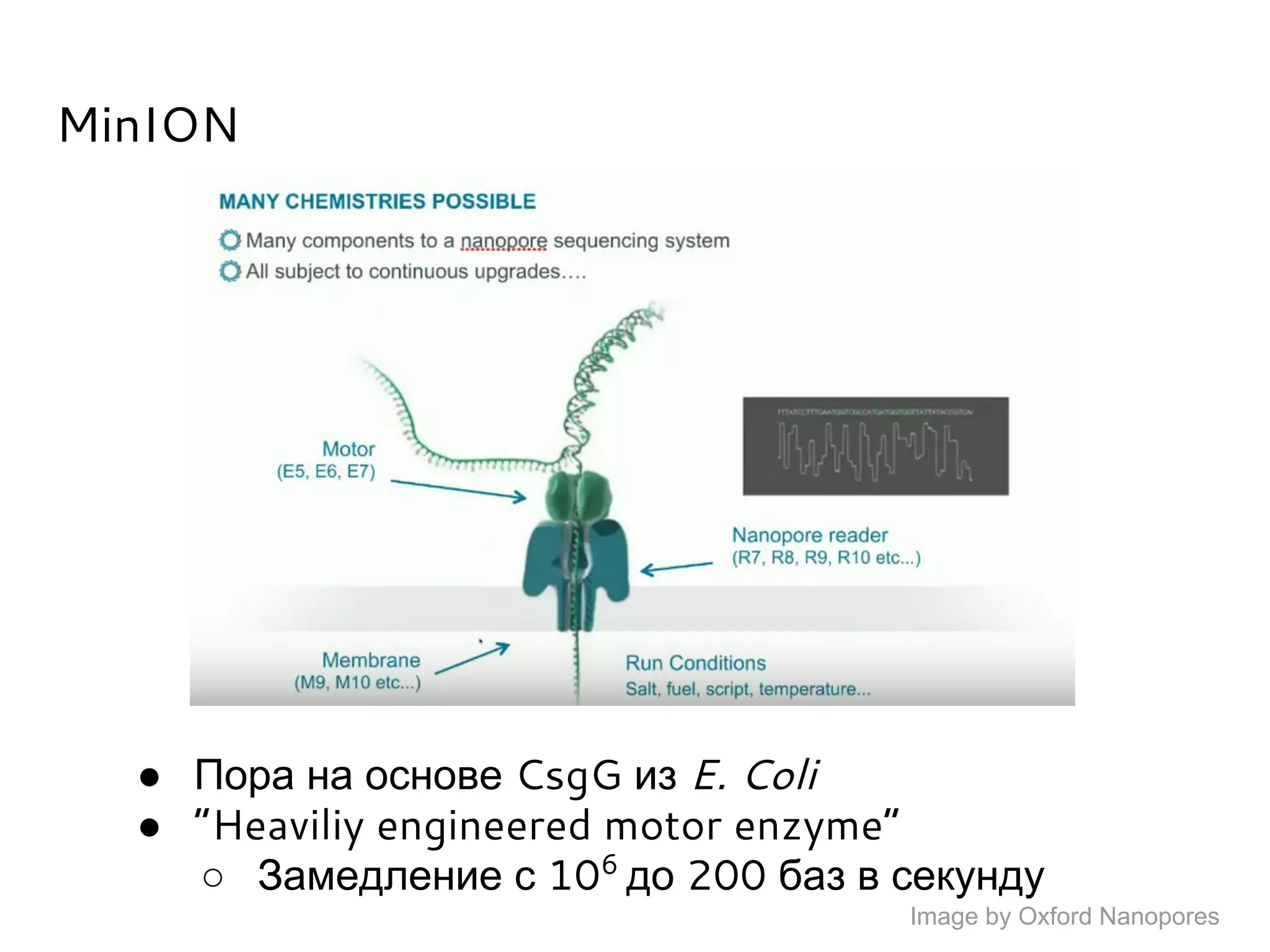
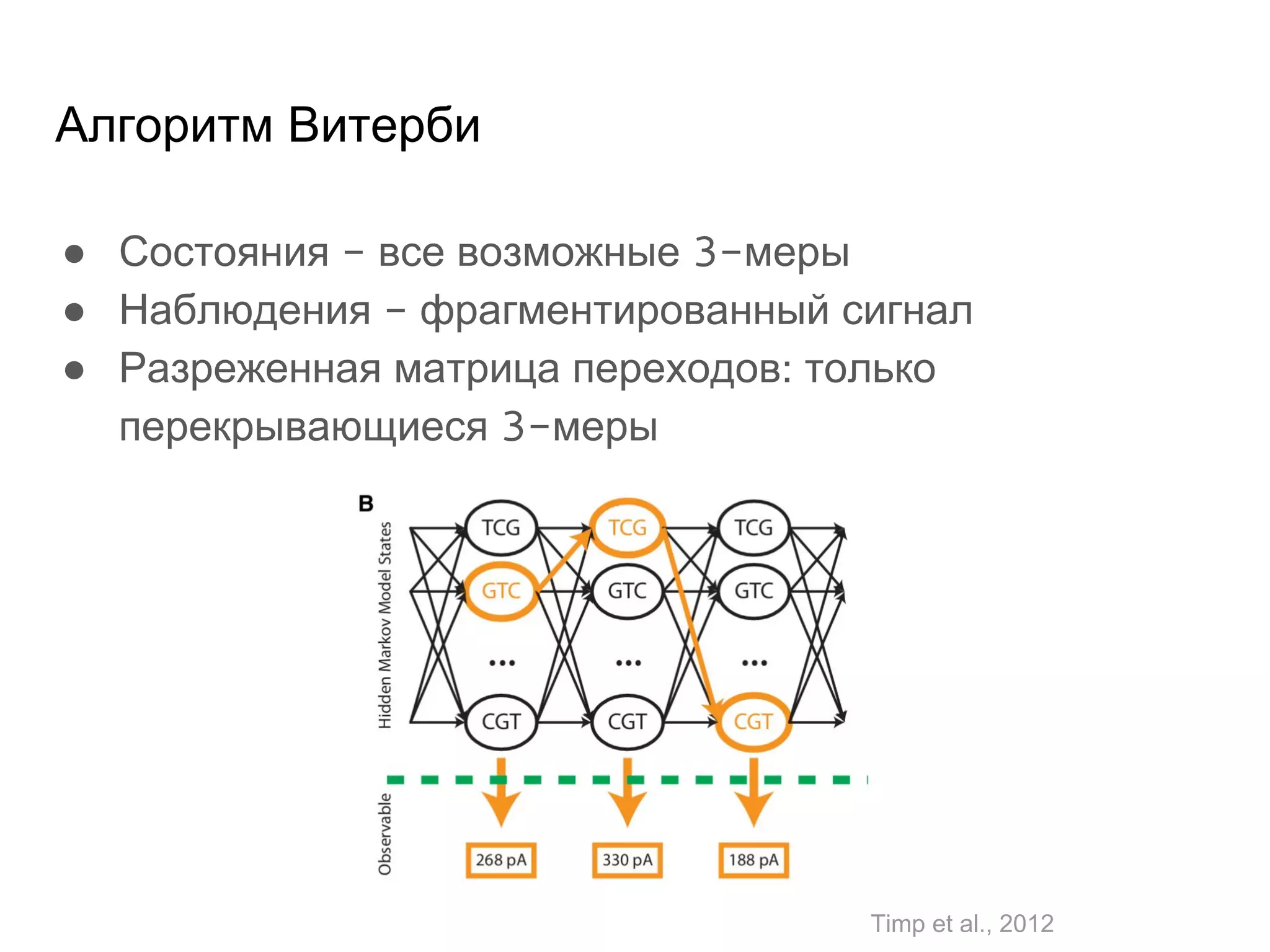
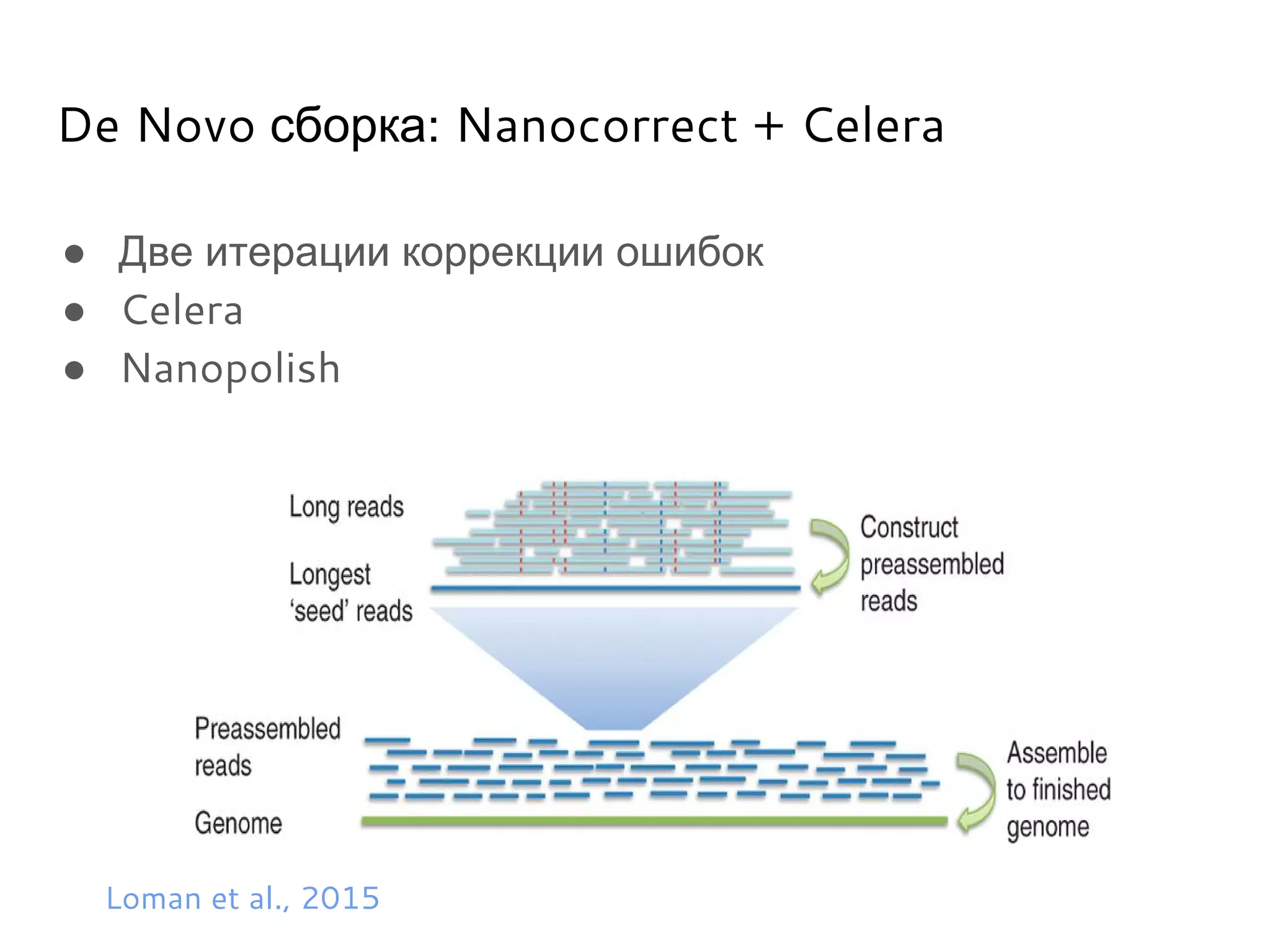
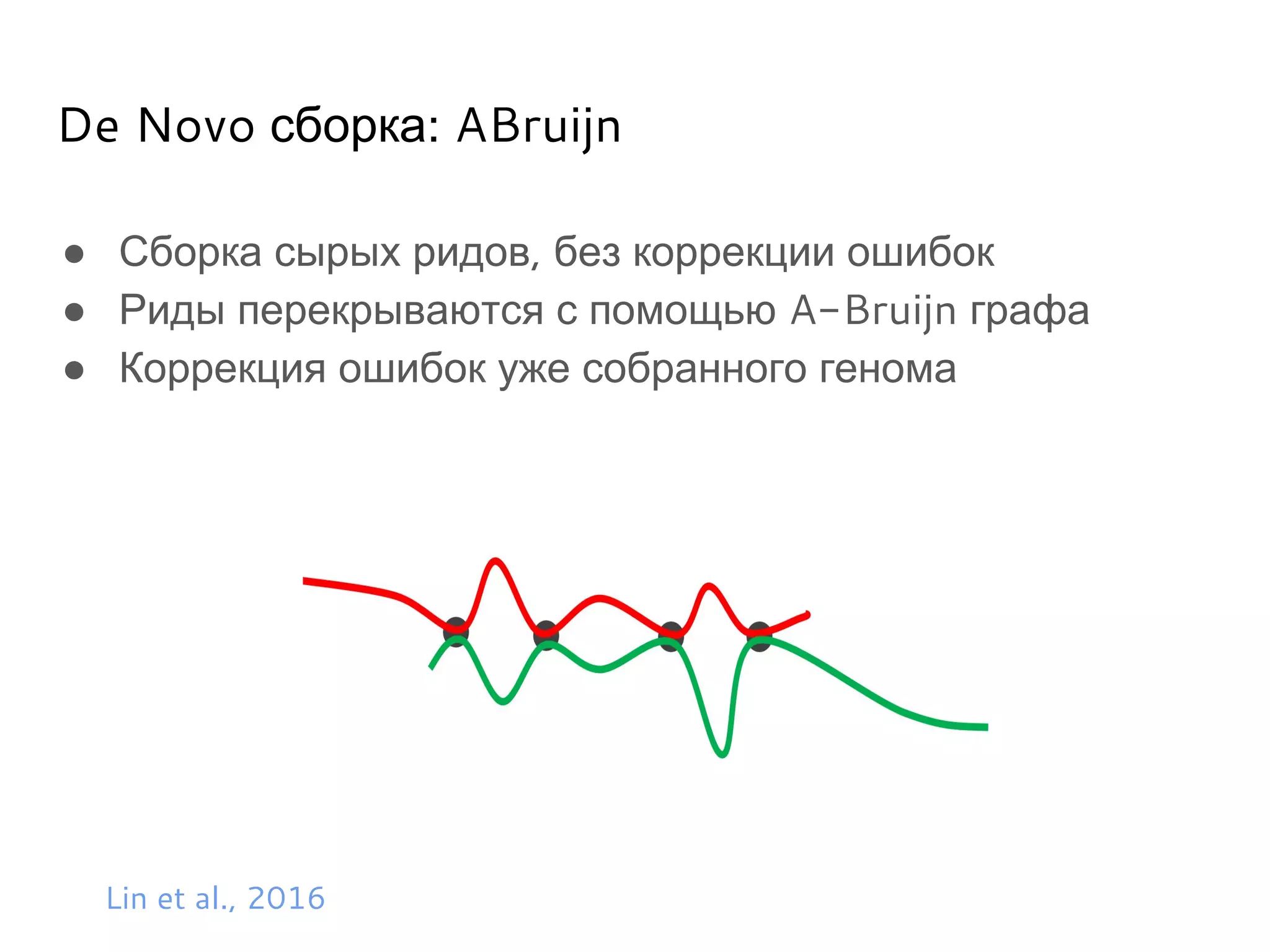
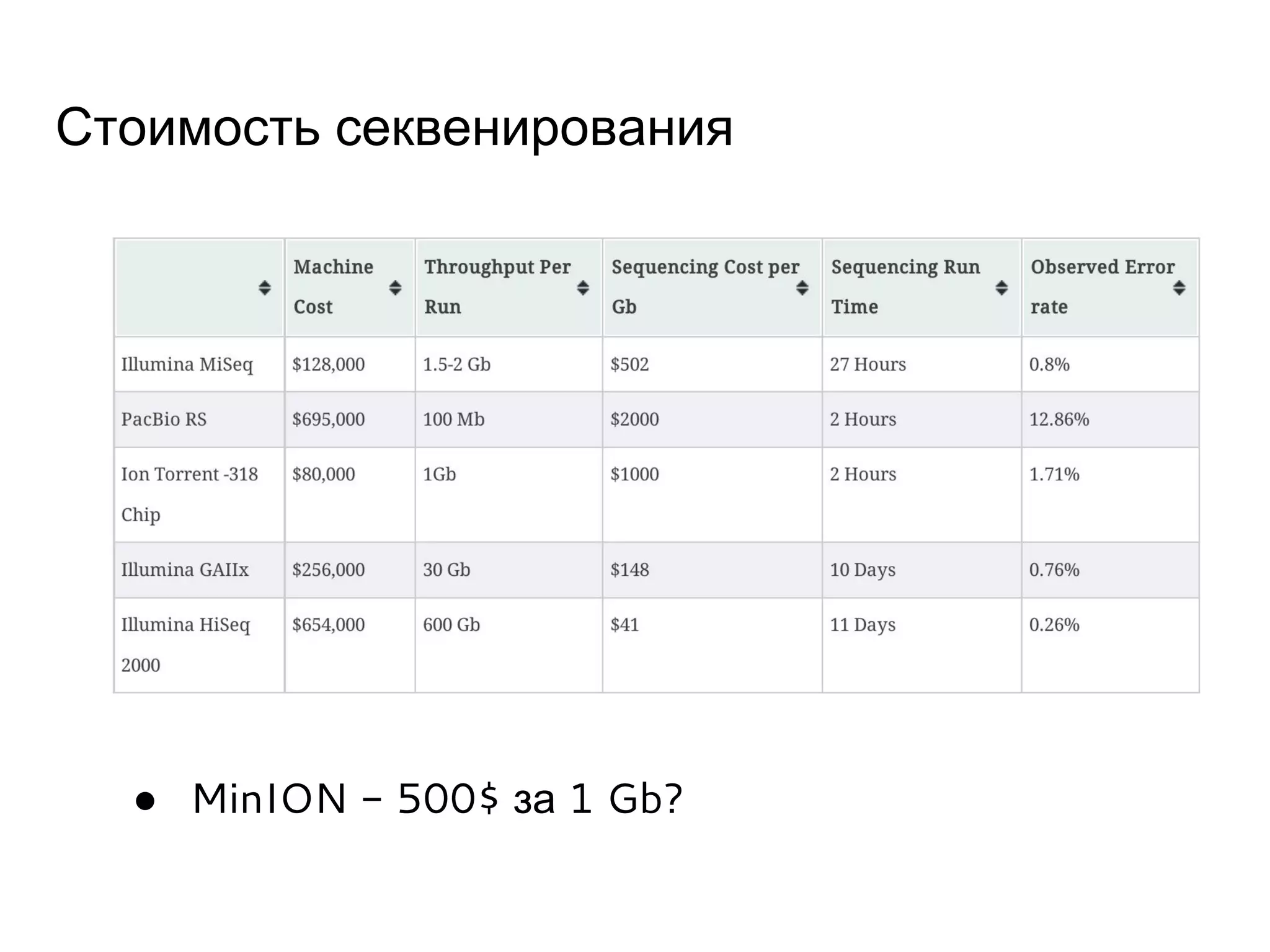
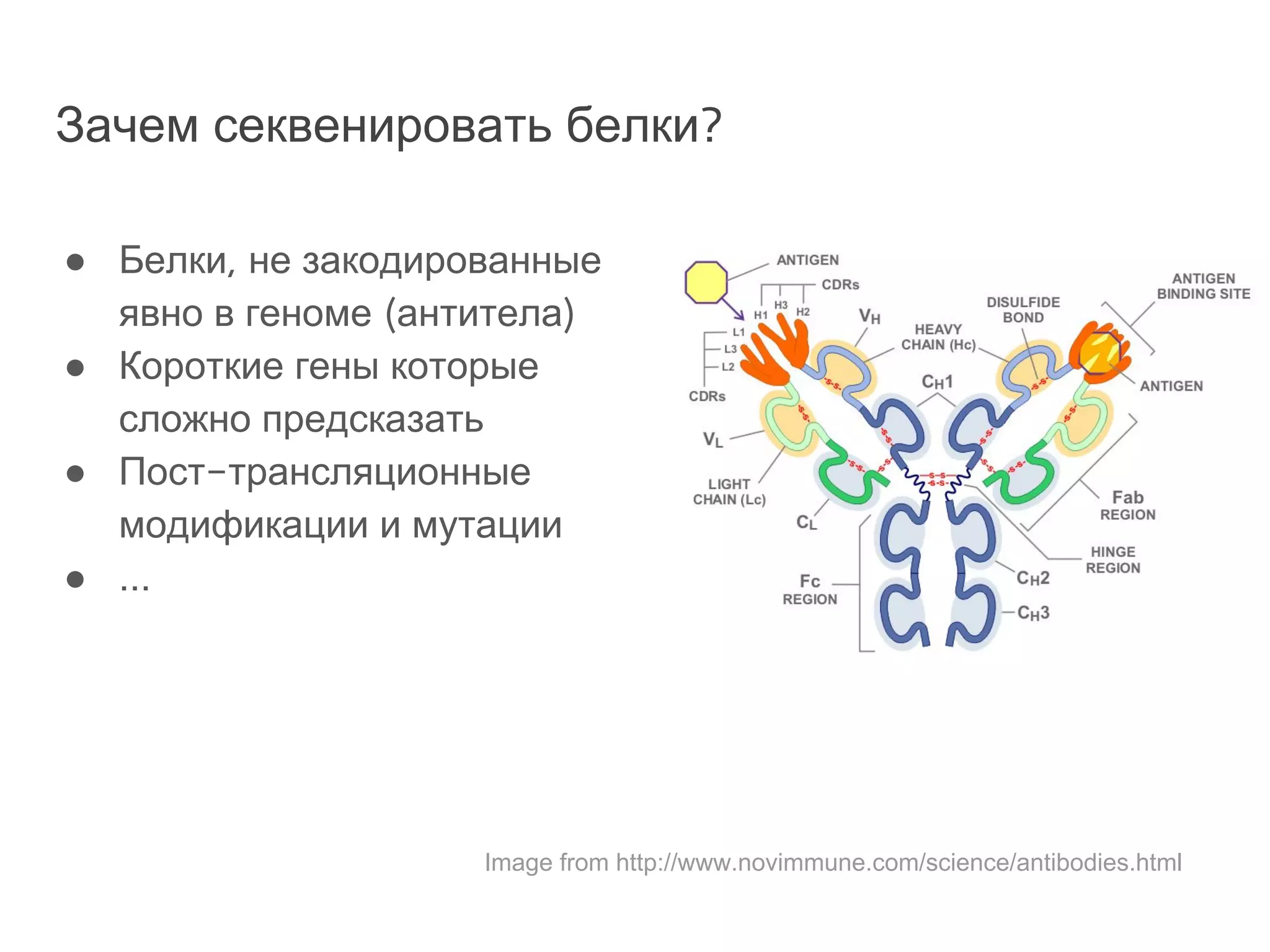
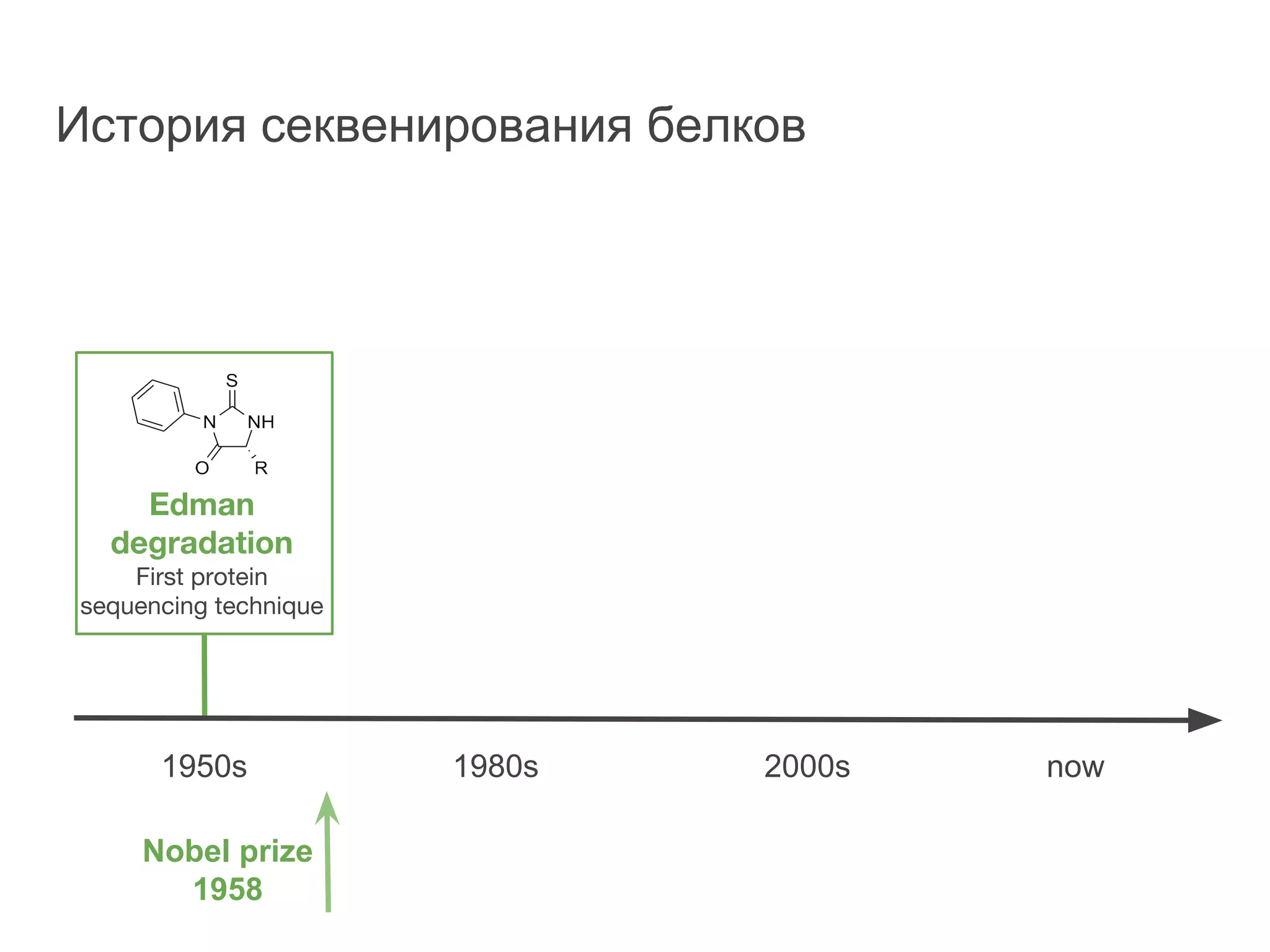
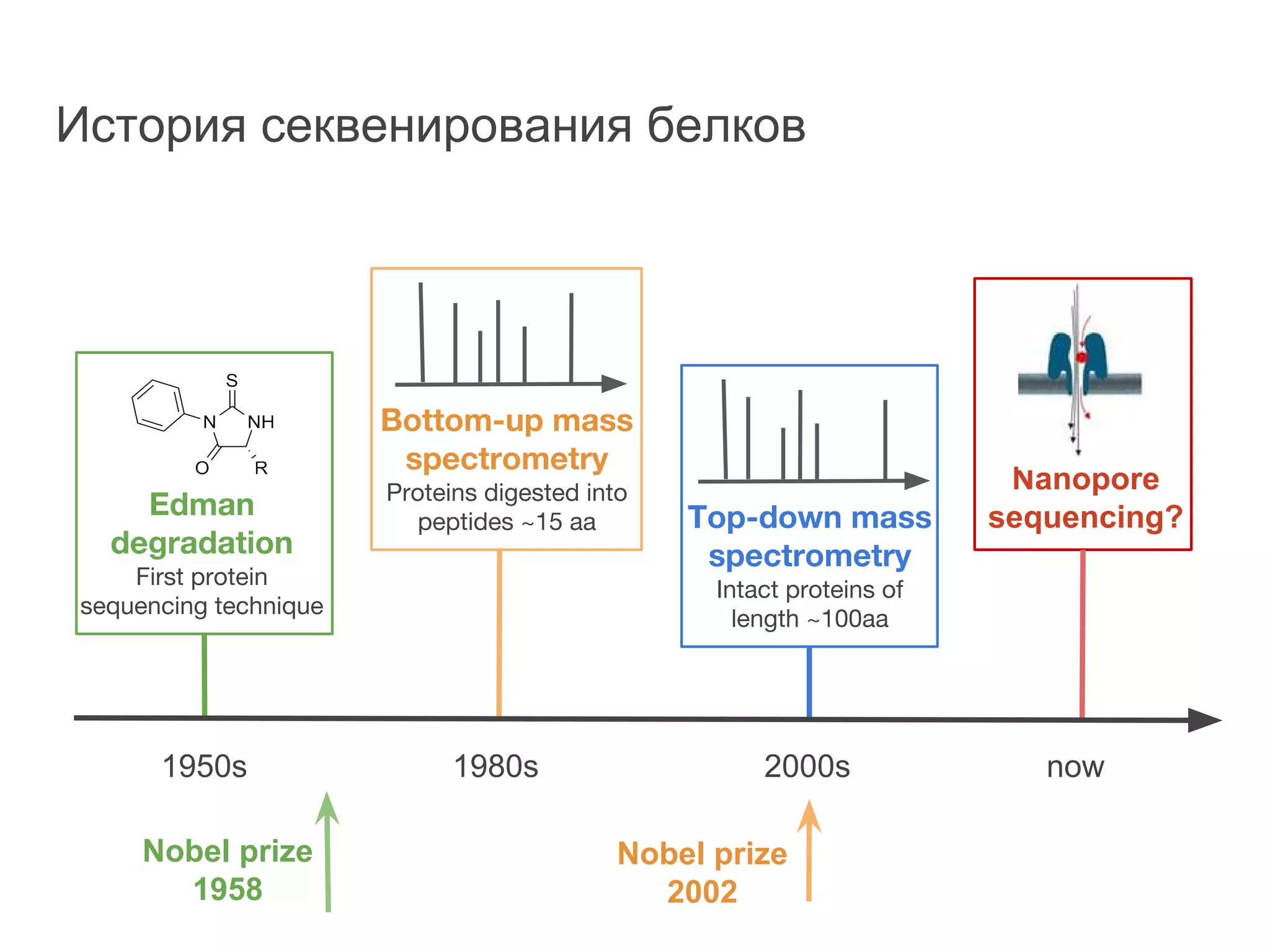
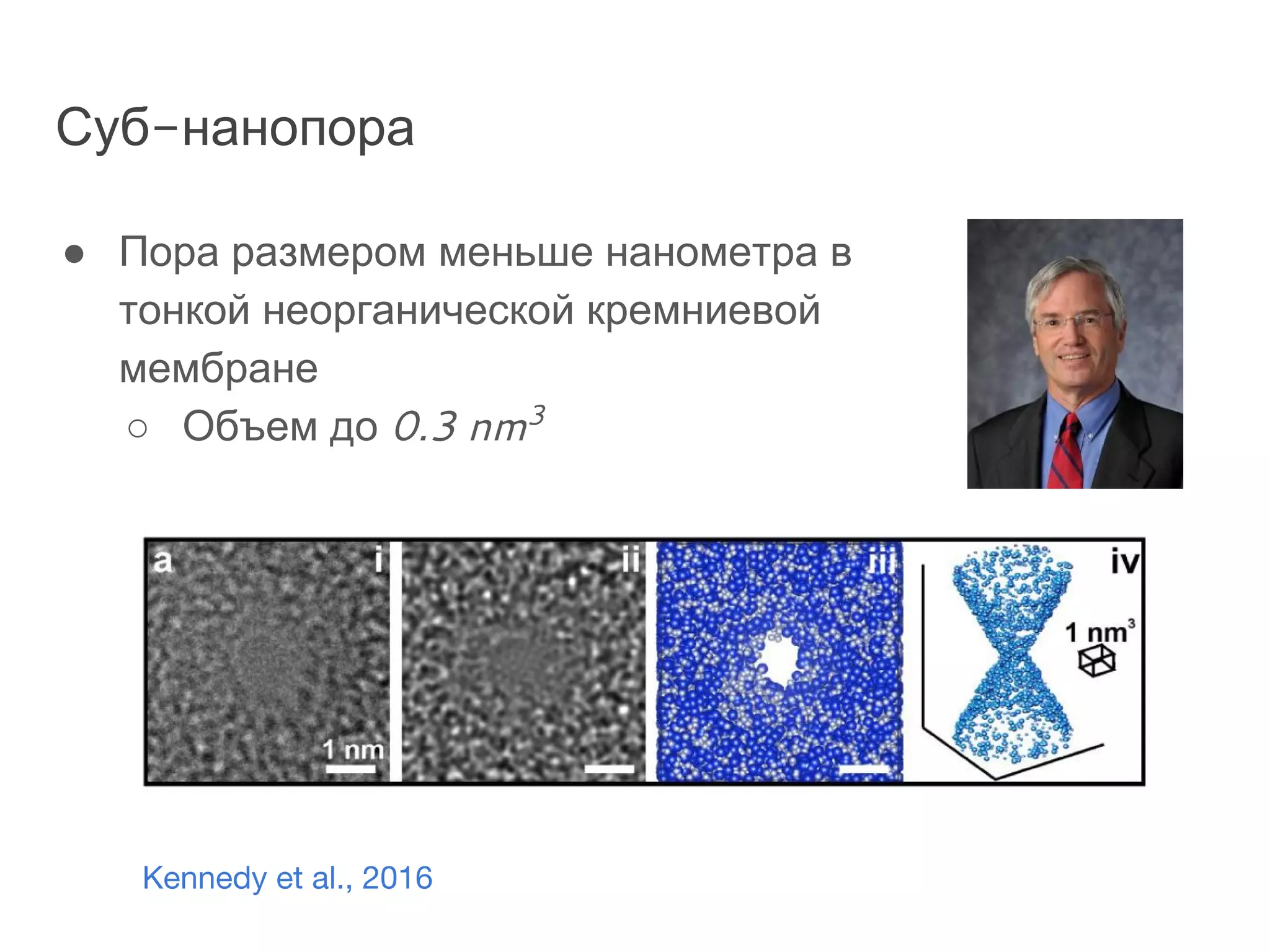
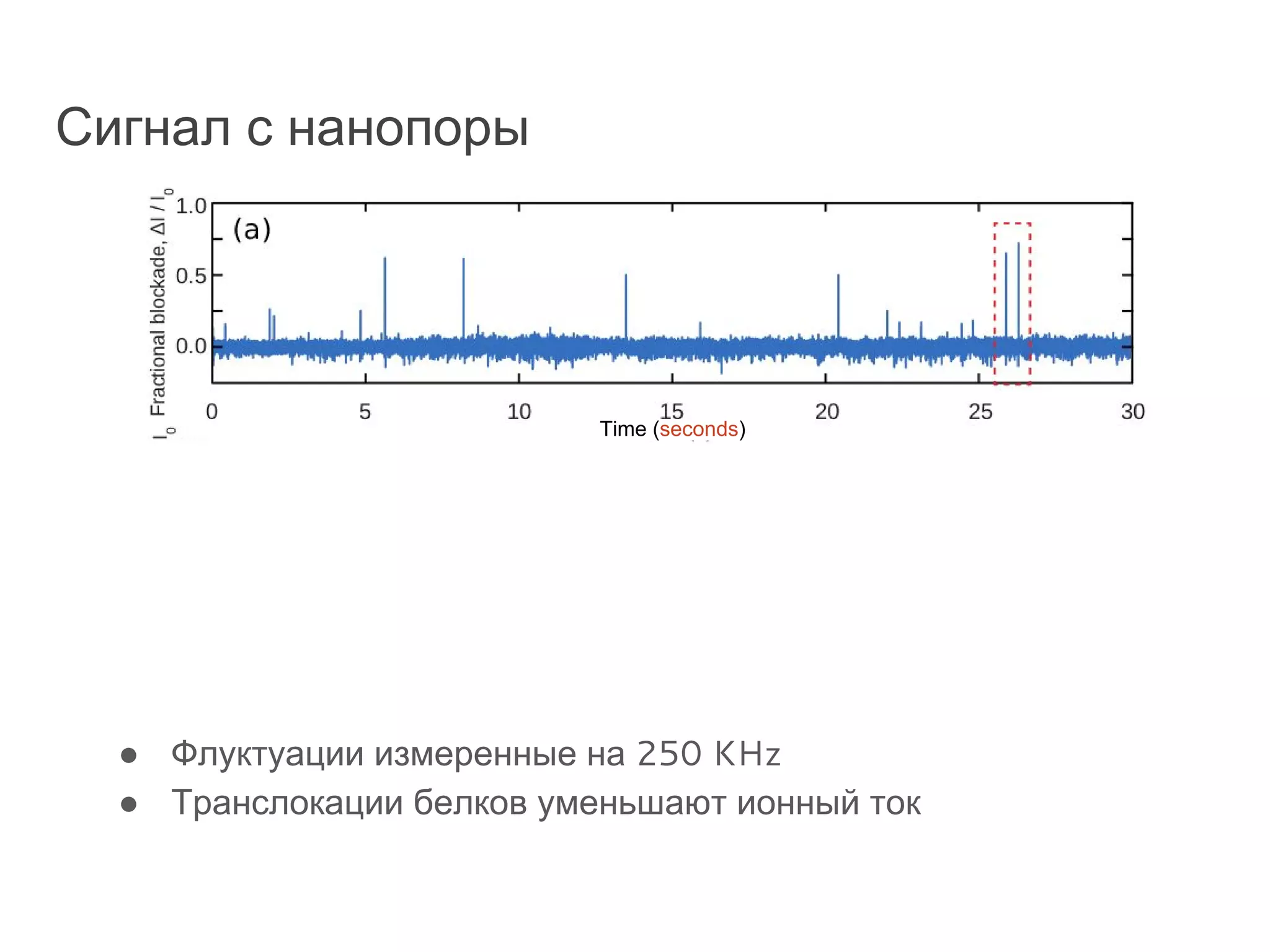
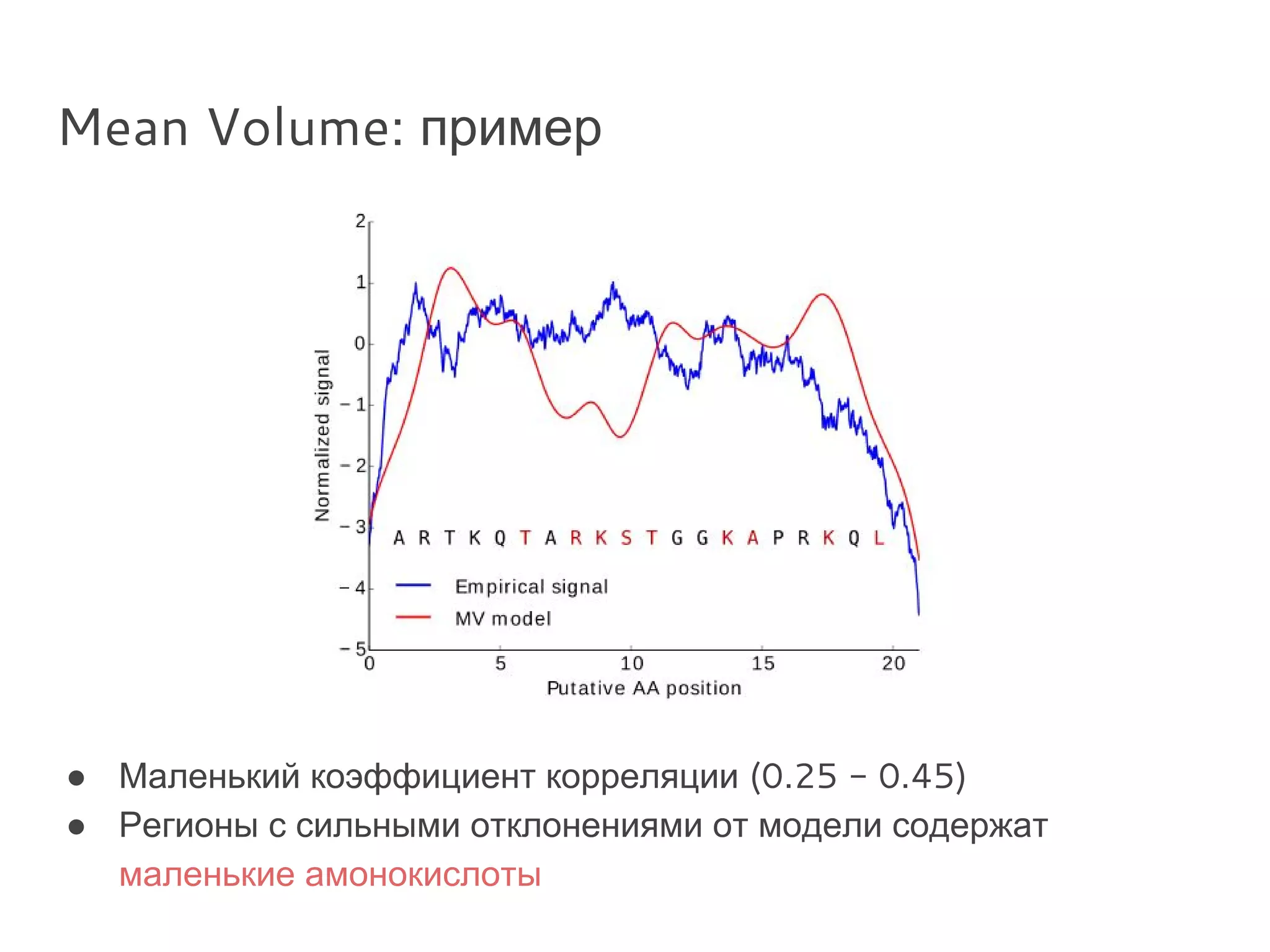
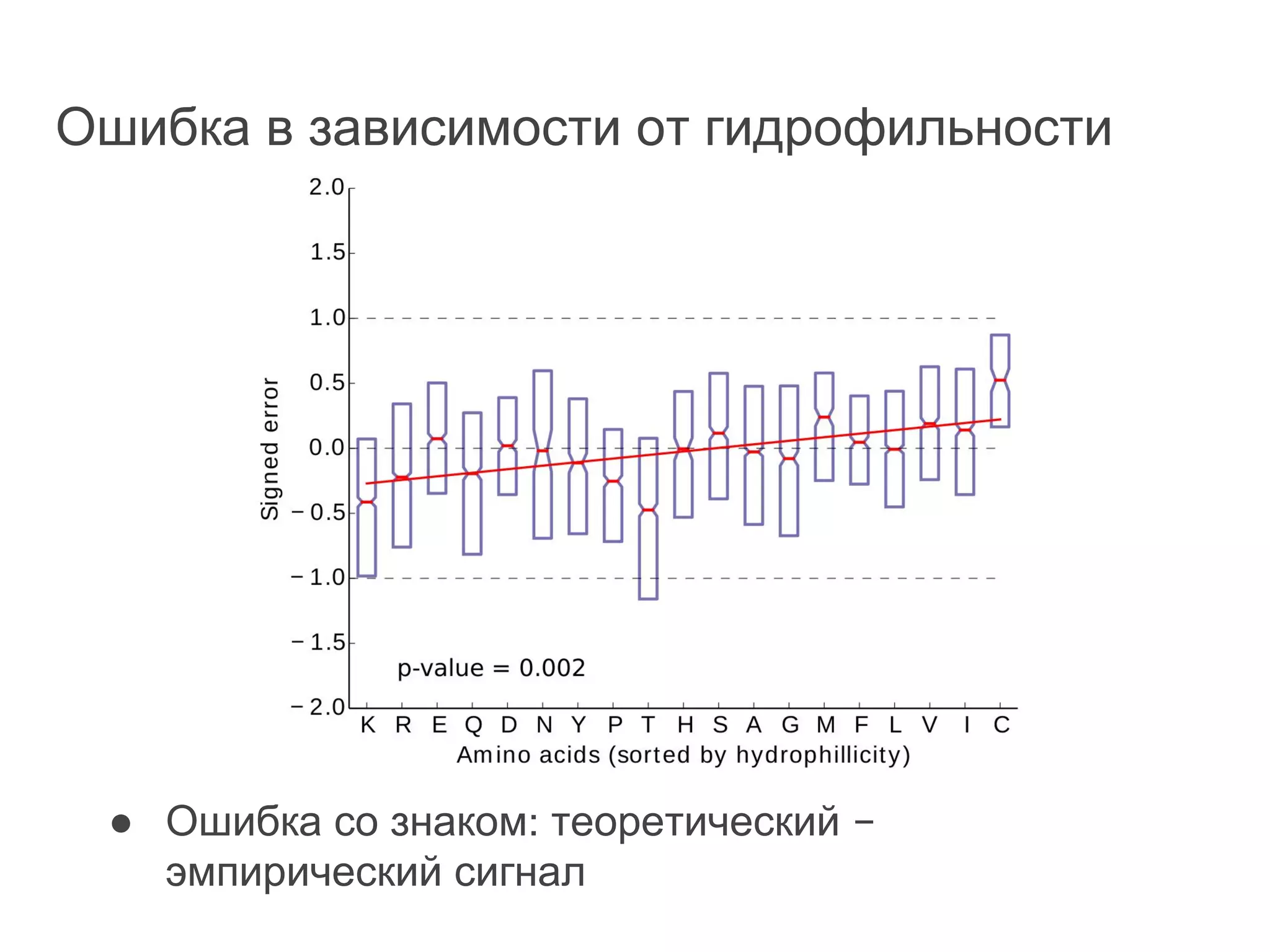
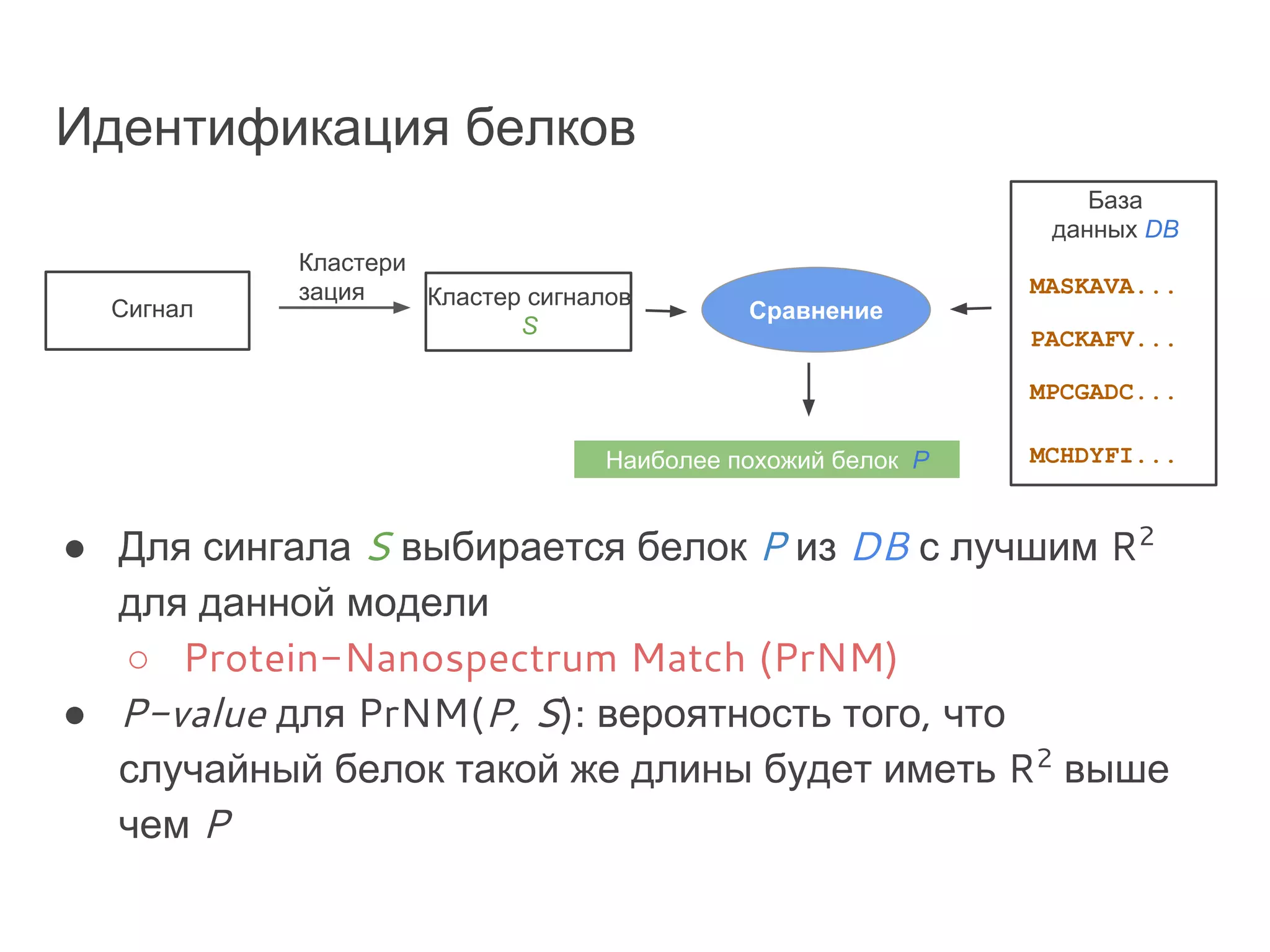
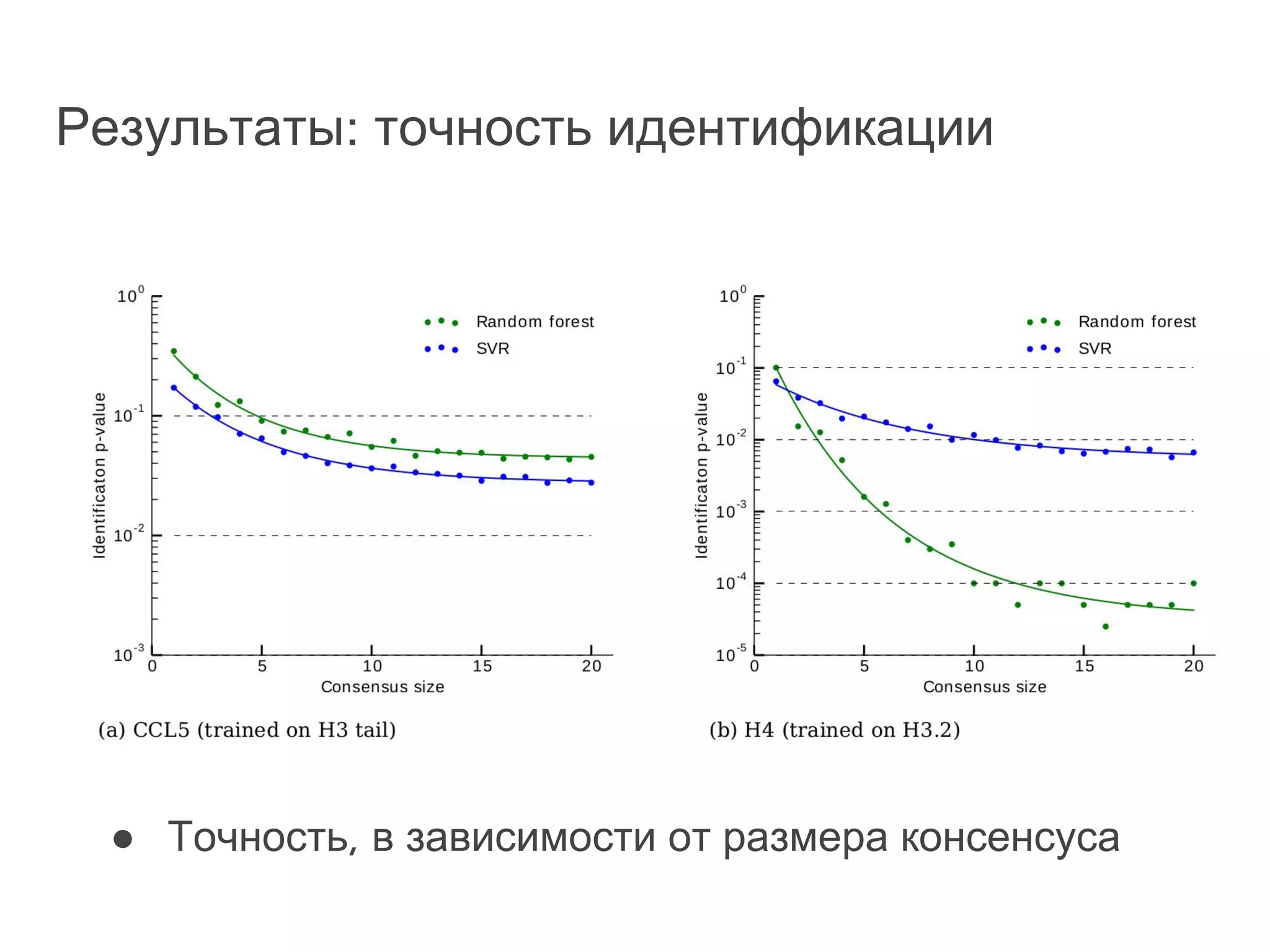
Документ охватывает методы секвенирования с использованием нанопор, включая их преимущества и недостатки по сравнению с традиционными методами, такими как Sanger и NGS. Также рассматривается секвенирование белков и сложности, связанные с анализом белковых последовательностей. Включены данные о стоимости оборудования и точности секвенирования, а также различные подходы к обработке полученных сигналов.