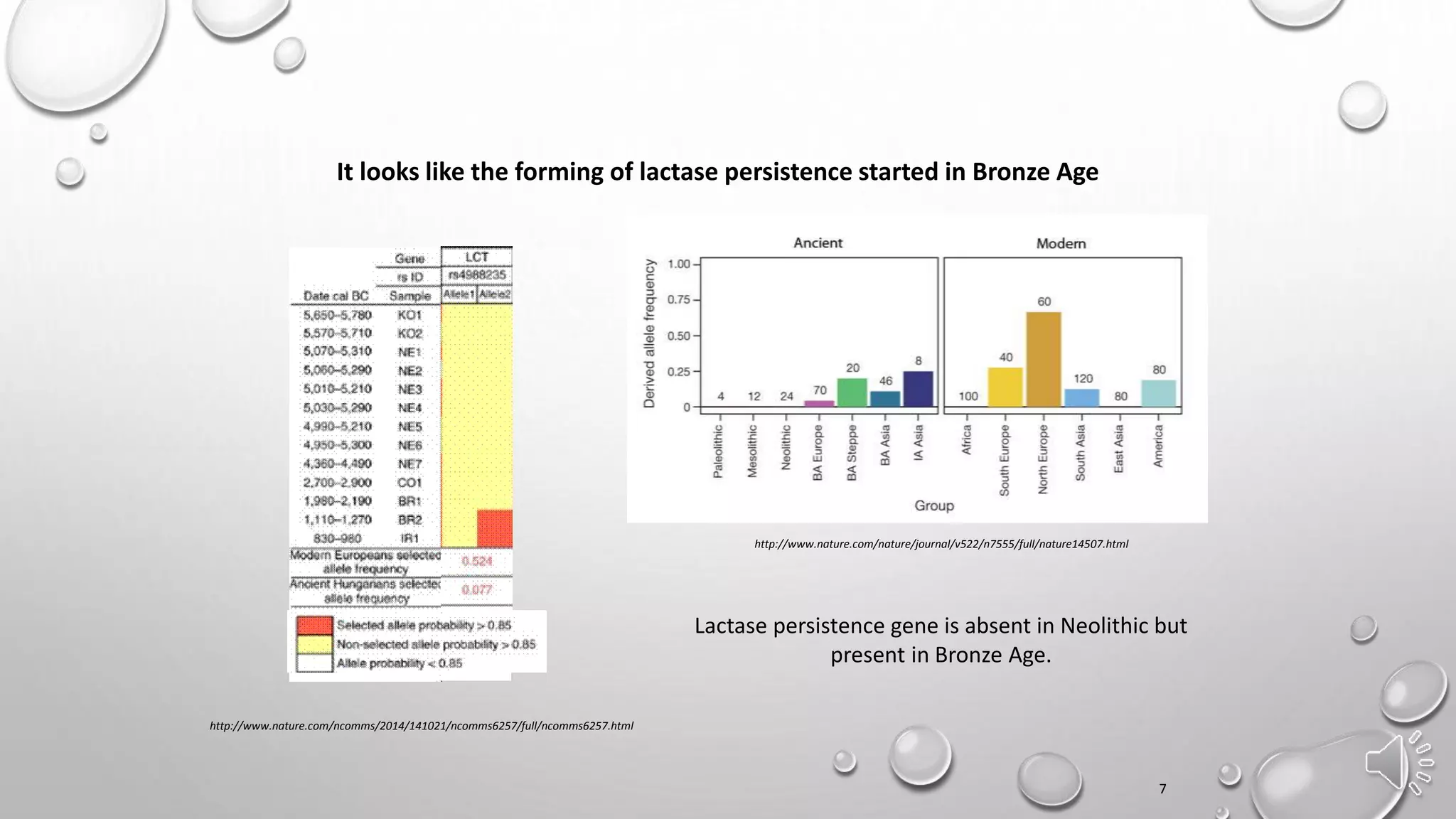
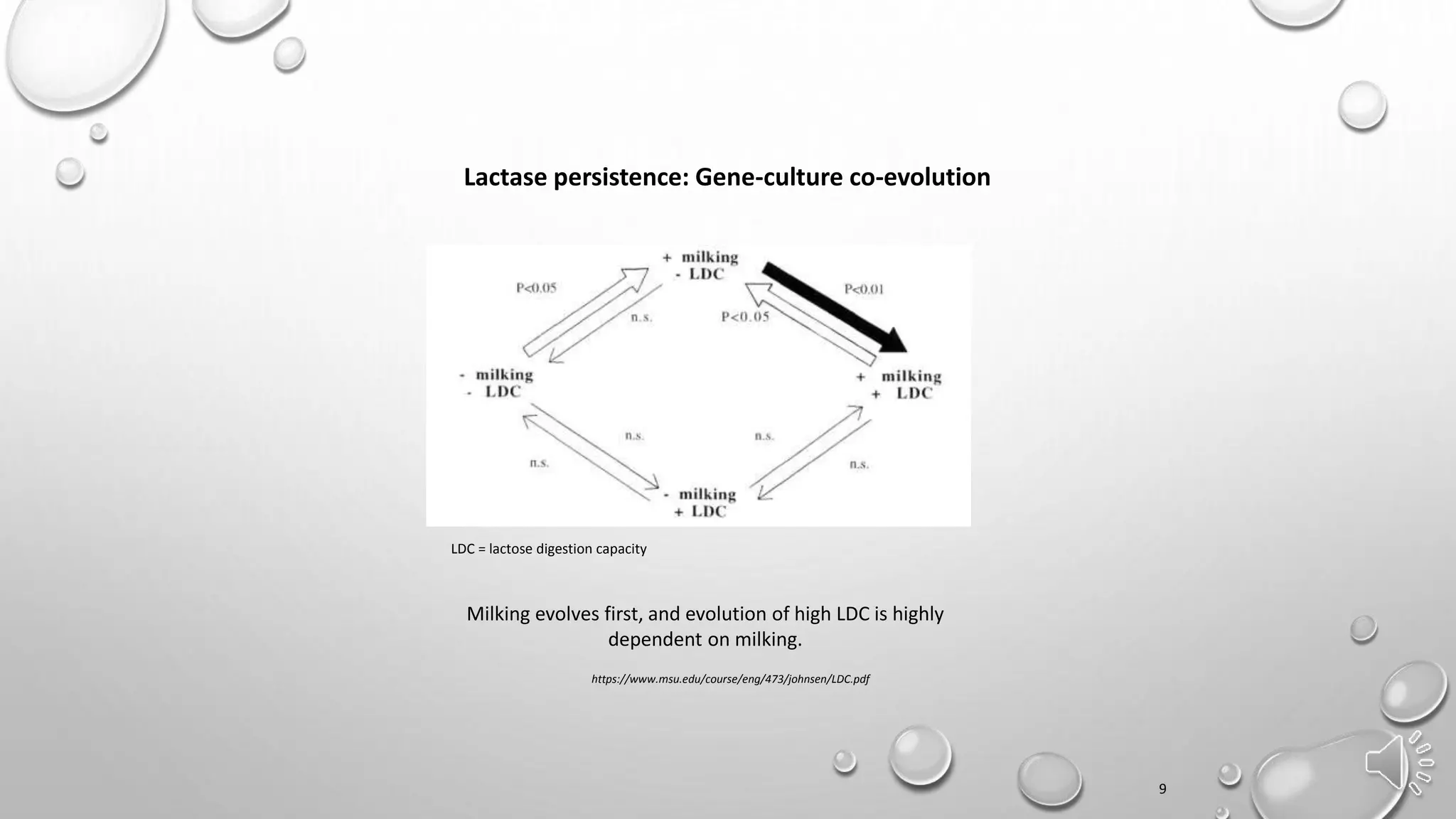
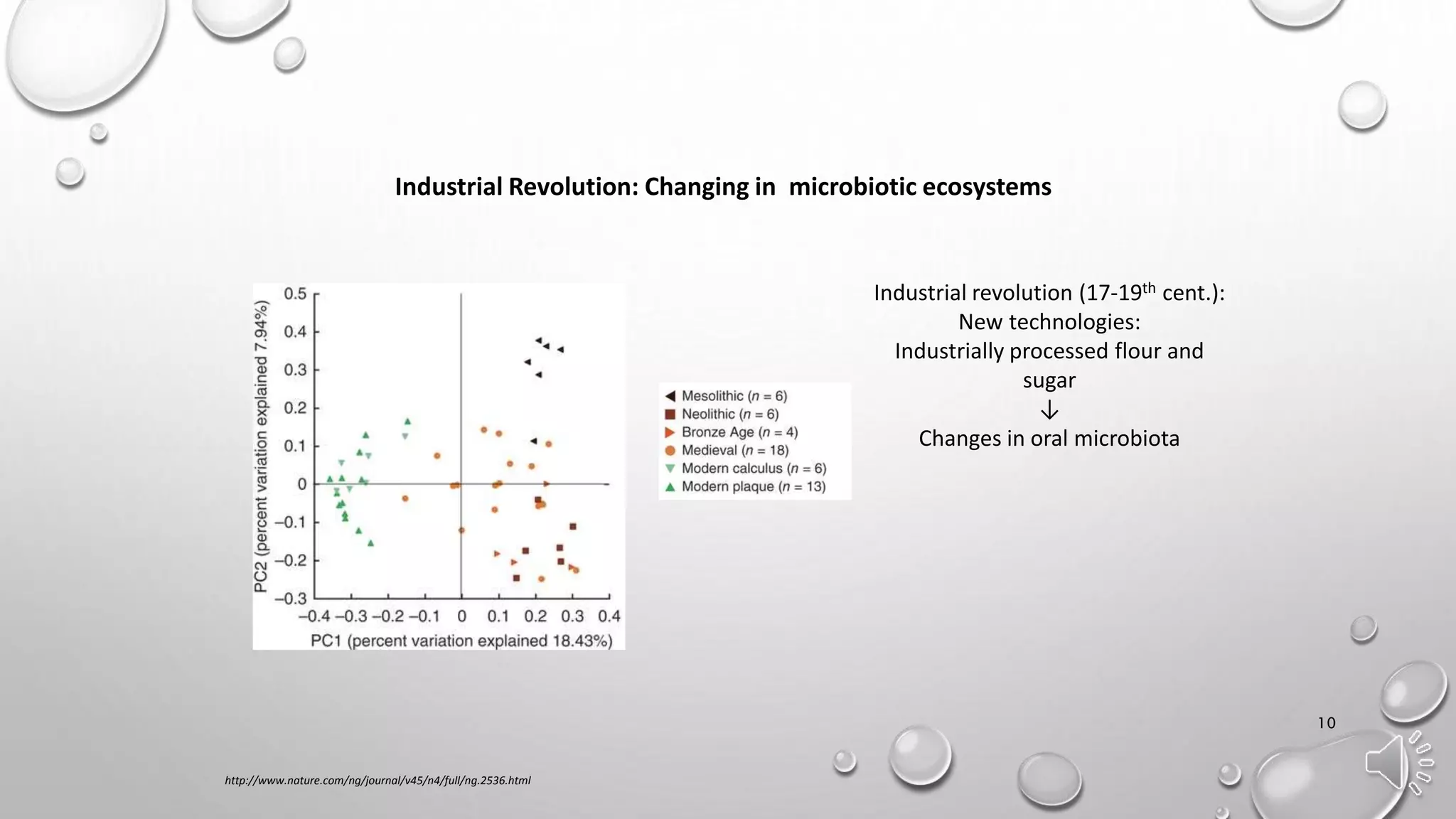
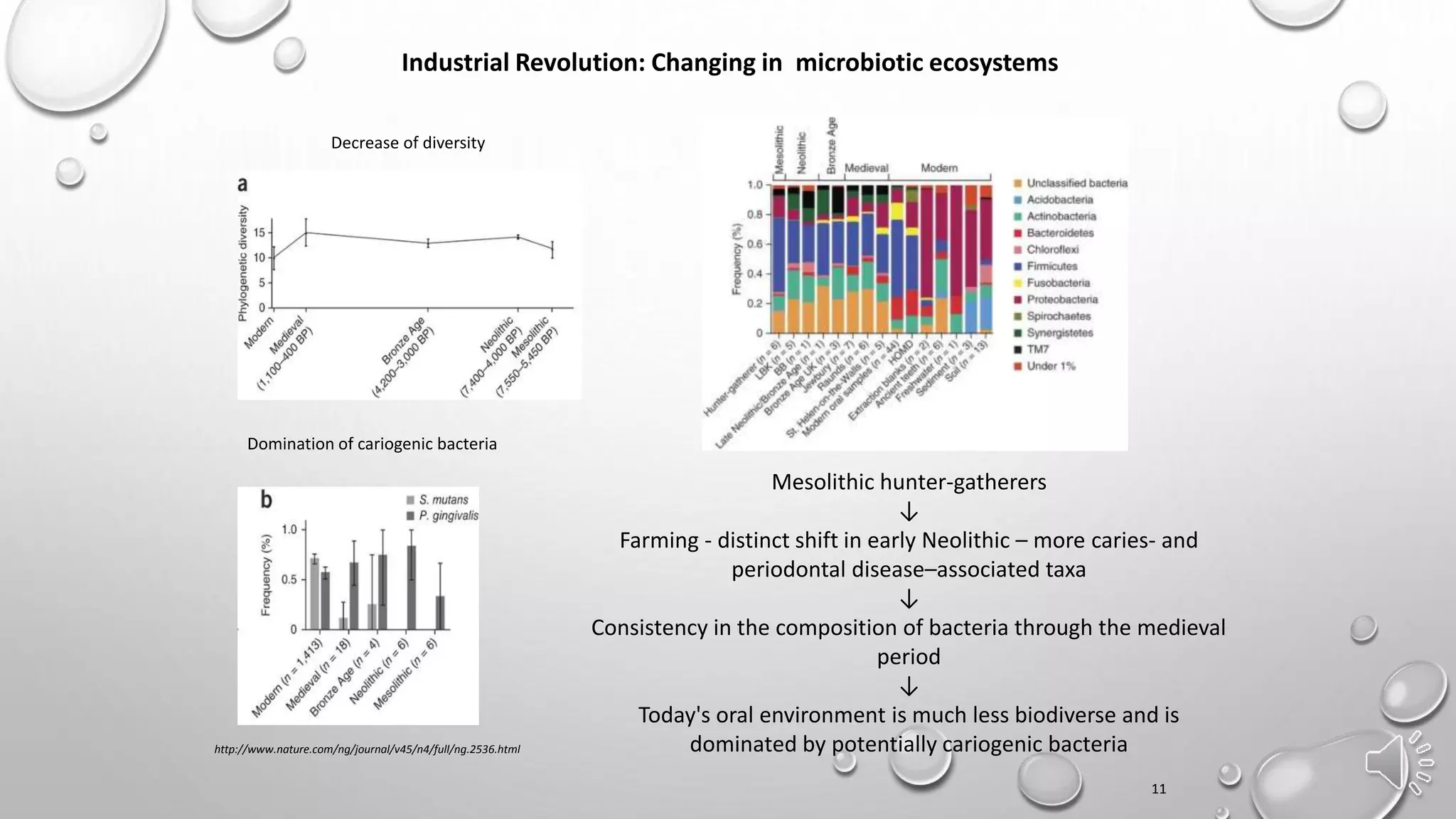
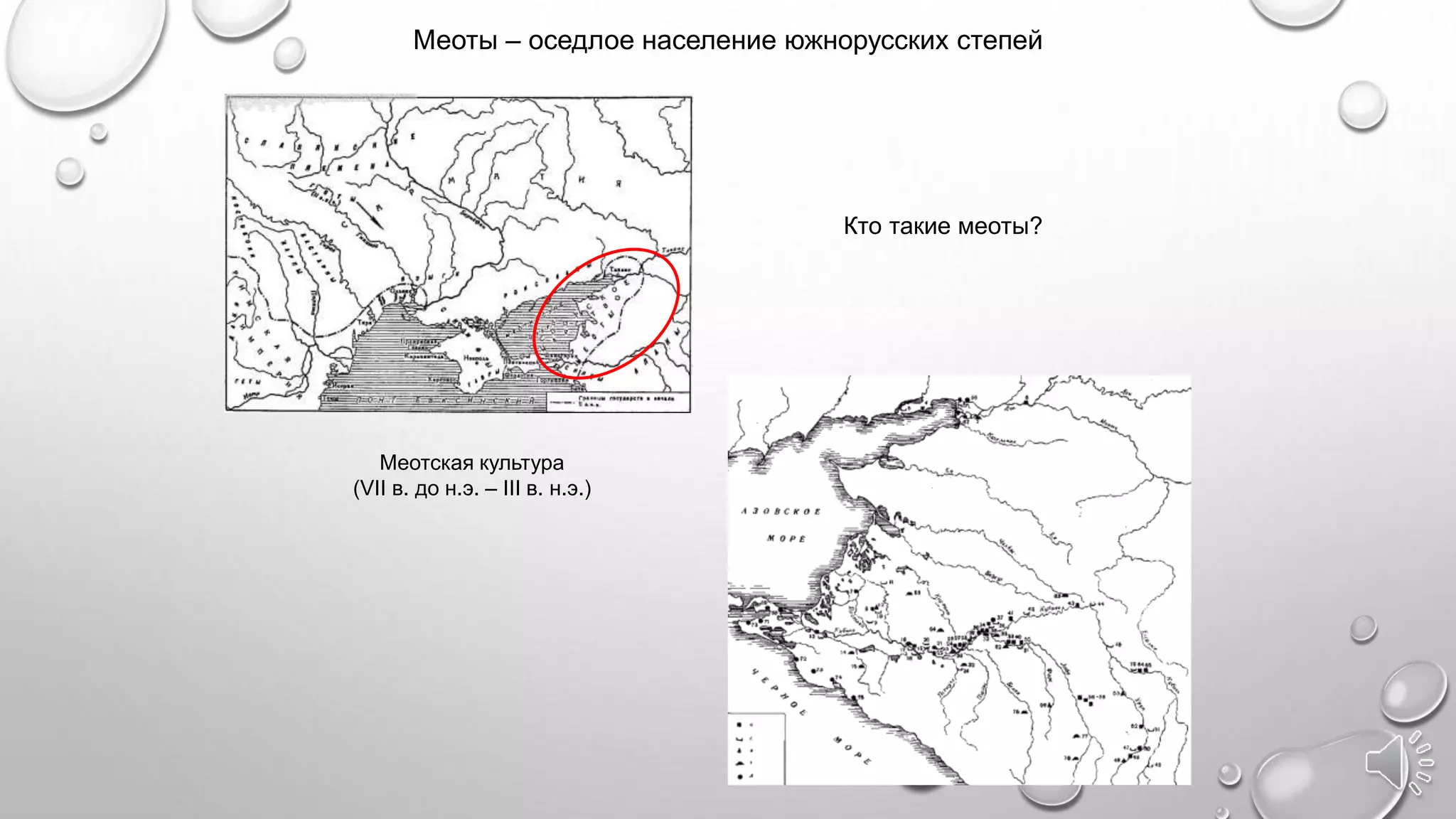
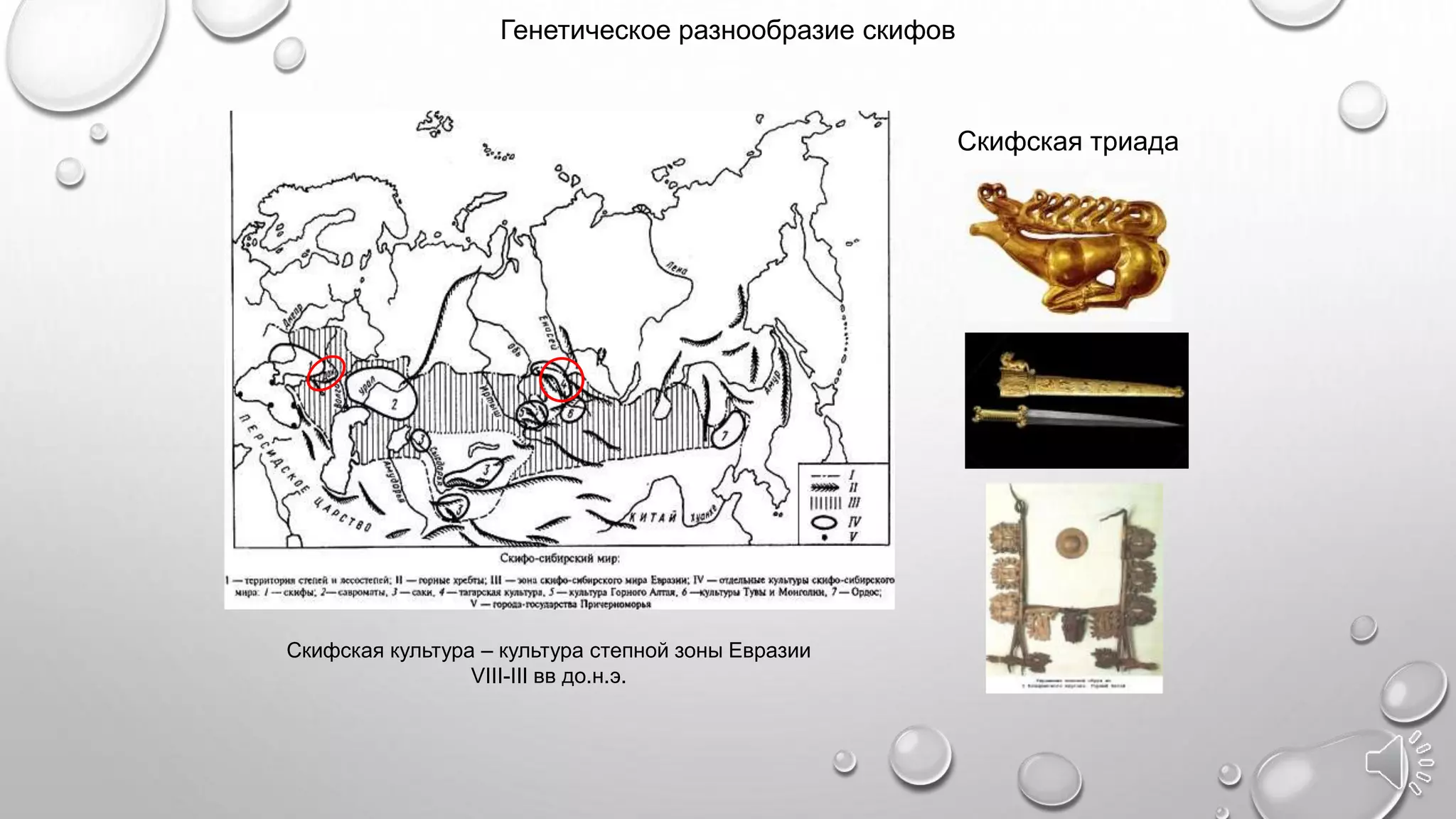
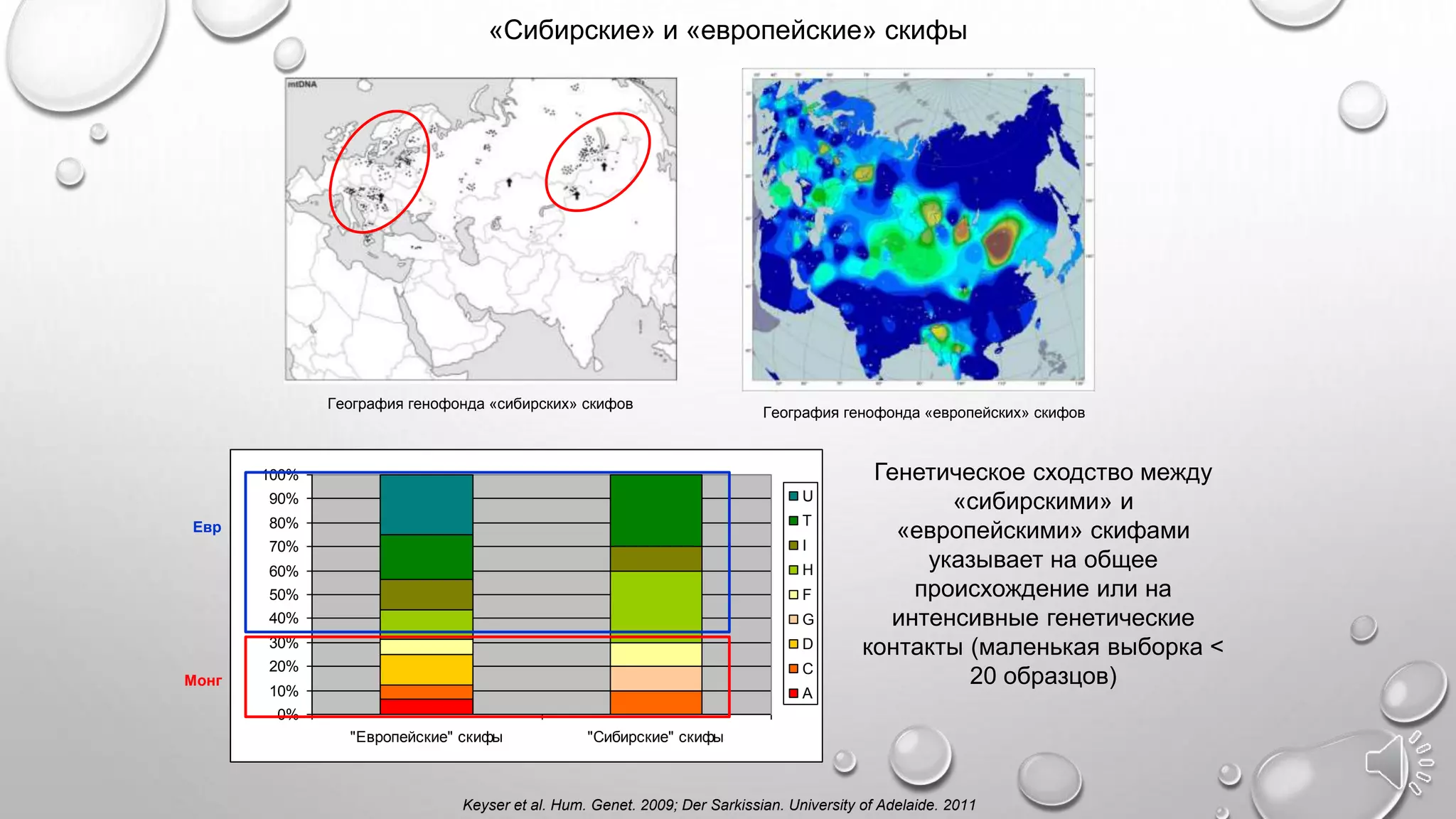
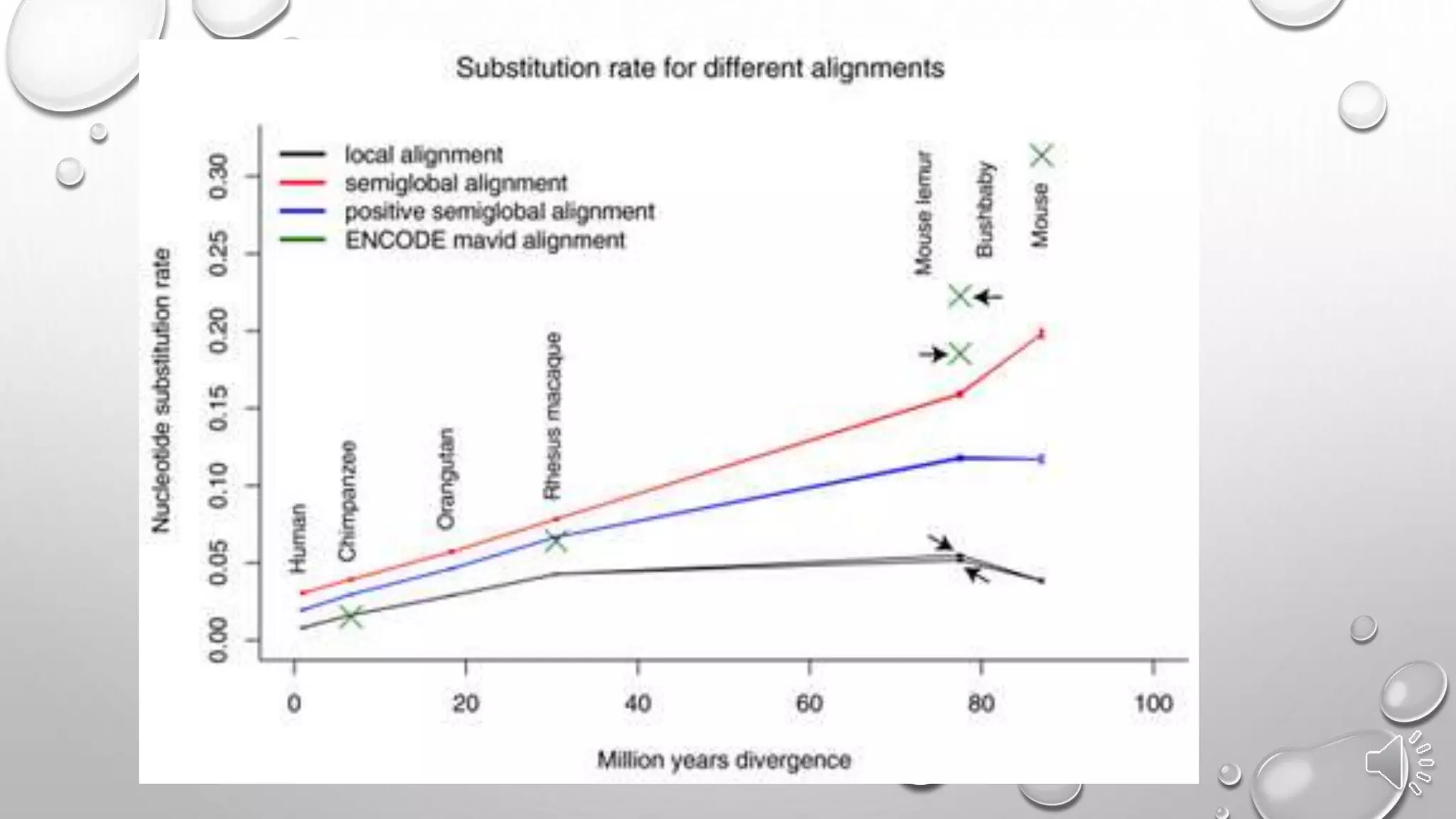
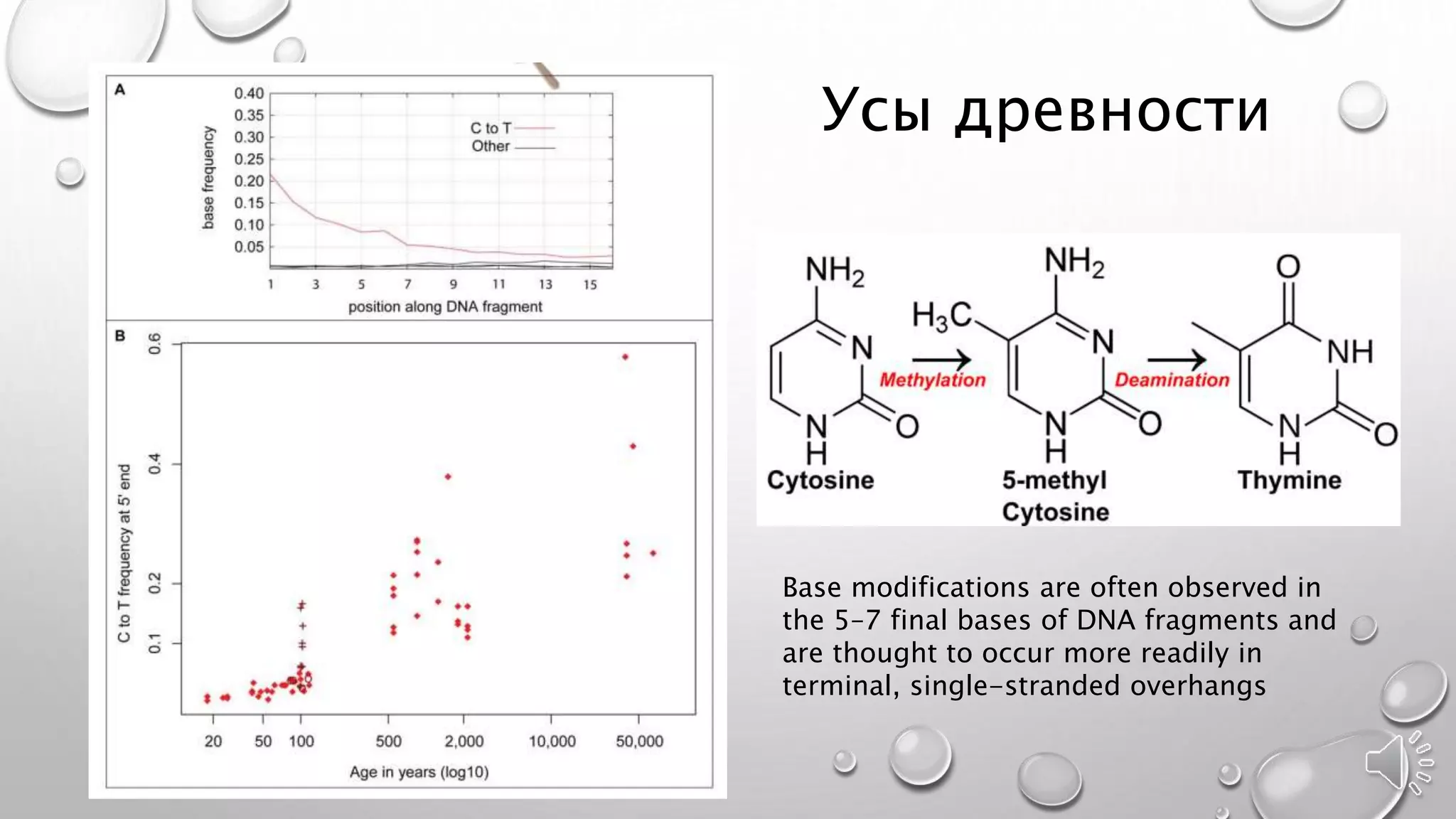
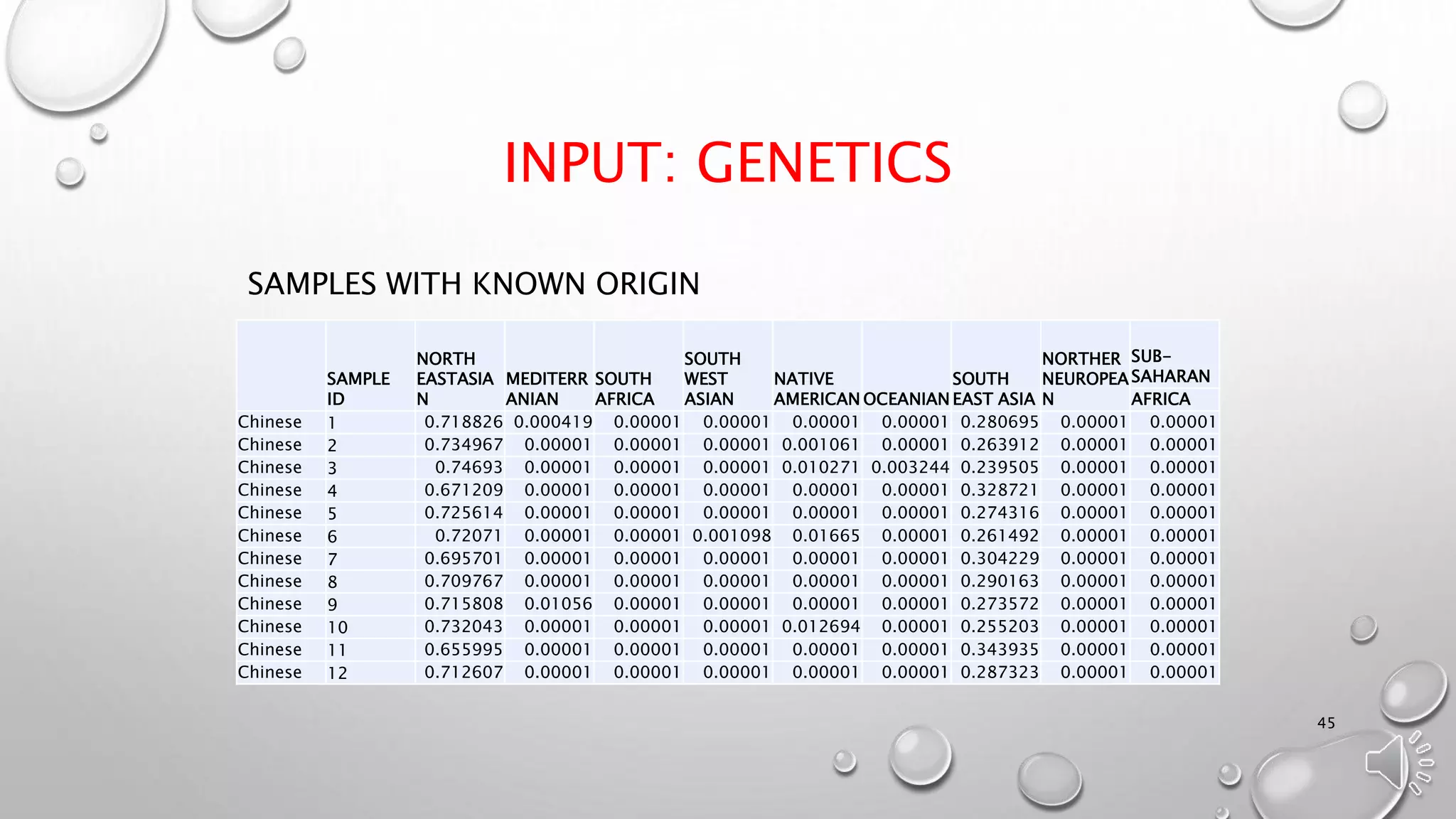
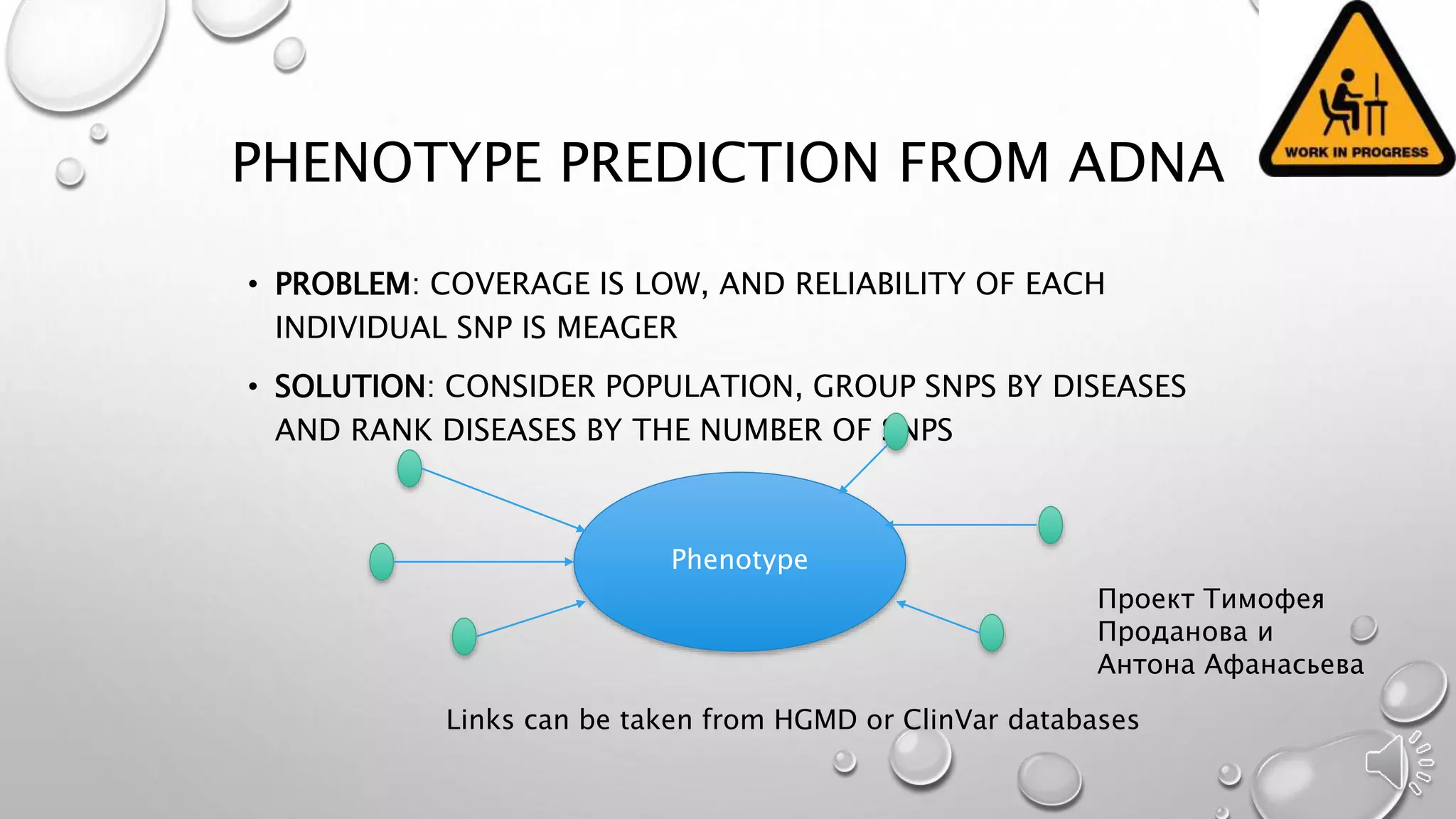
Документ обсуждает важность древней ДНК для понимания истории и эволюции, исследуя генетический вклад неандертальцев, денисовцев и других древних видов в современных людях. Методы анализа древней ДНК помогают освещать миграцию, родственные связи и физические характеристики, такие как цвет кожи и адаптация к климату. Проблемы, связанные с загрязнением и фрагментацией ДНК, подчеркивают необходимость точных методов исследования для извлечения значимой информации из древних образцов.