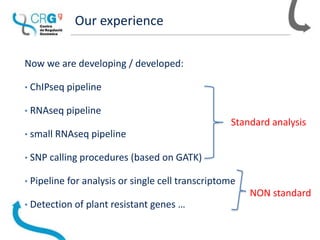
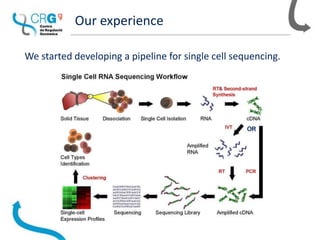
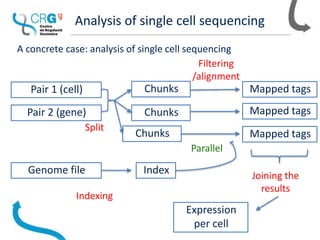
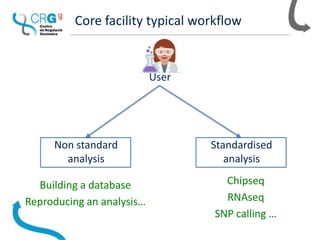
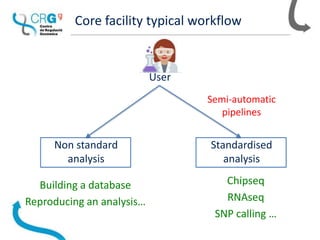
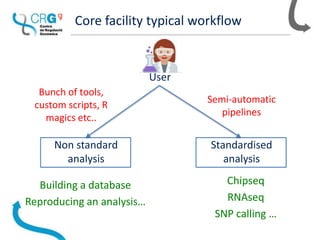
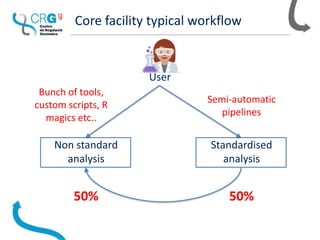
The document outlines the workflow and experiences of a bioinformatics core facility using Nextflow for genomic analyses, highlighting the importance of automation, parallelization, and reproducibility. The facility has developed several pipelines for RNA-seq, SNP calling, and single-cell transcriptomes while discussing the benefits of using containers to address compatibility issues. Future ideas include creating semi-automatic reports and a content management system to enhance data handling.


![Why nextflow?
• Standard analysis:
• Automation, parallelization, portability, reproducibility
(together with containers).
• NF allows adding new steps without pain (thanks to isolation
of processes) in a collaborative way
[After 2 years] Can you redo the SAME analysis with new samples?](https://image.slidesharecdn.com/cozzutonextflow-170915131502/85/From-Zero-to-Nextflow-2017-9-320.jpg)