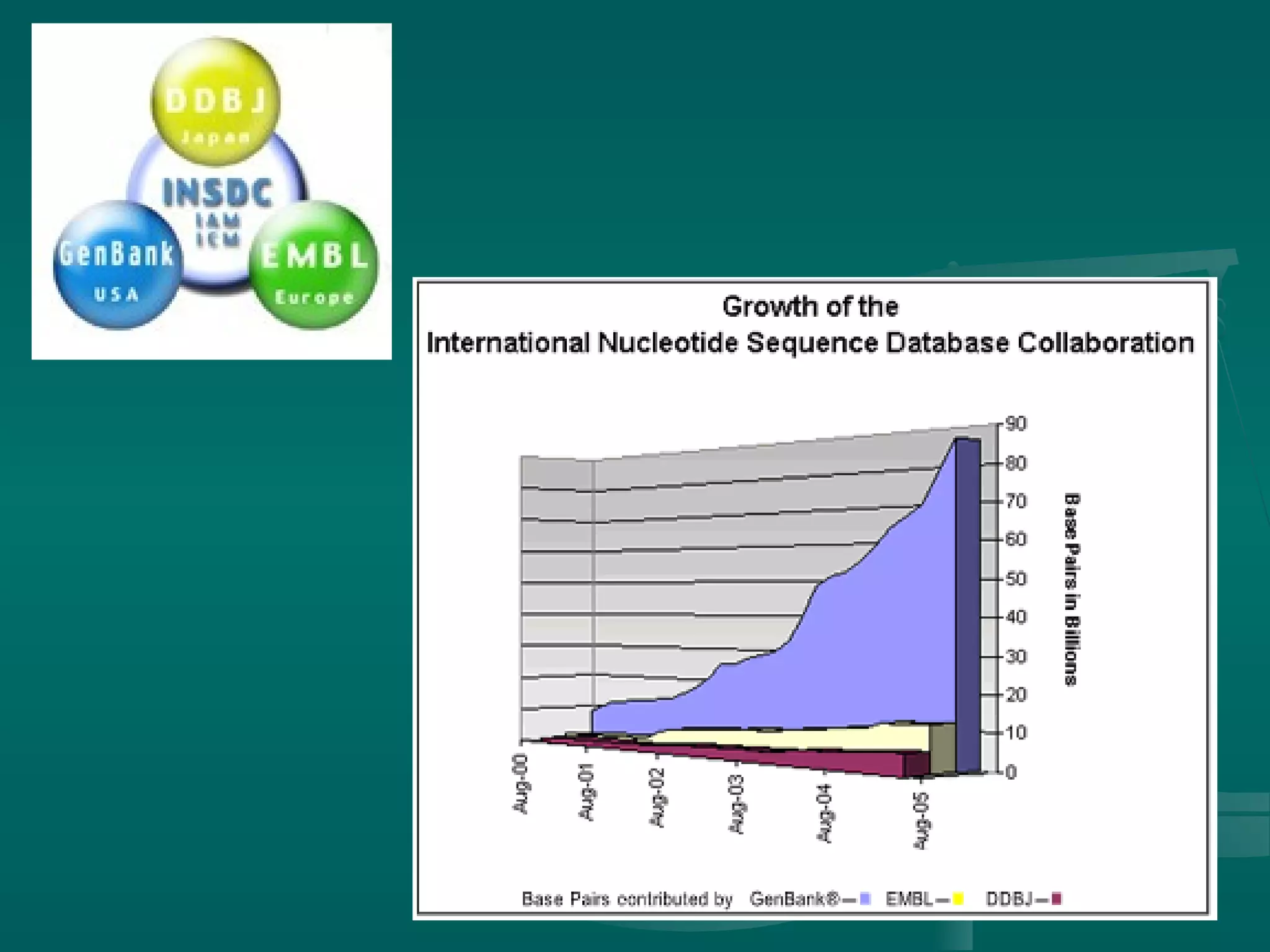
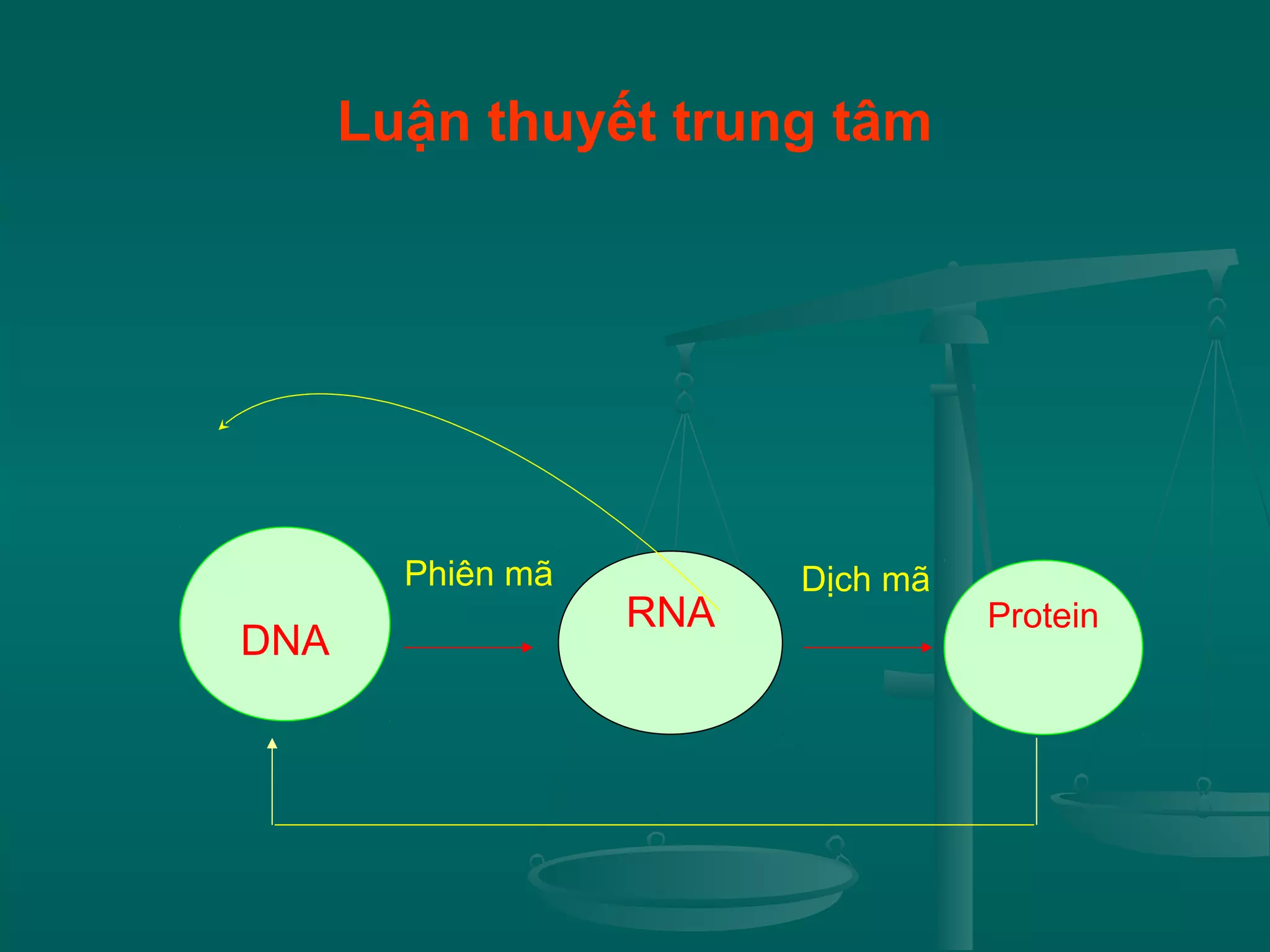
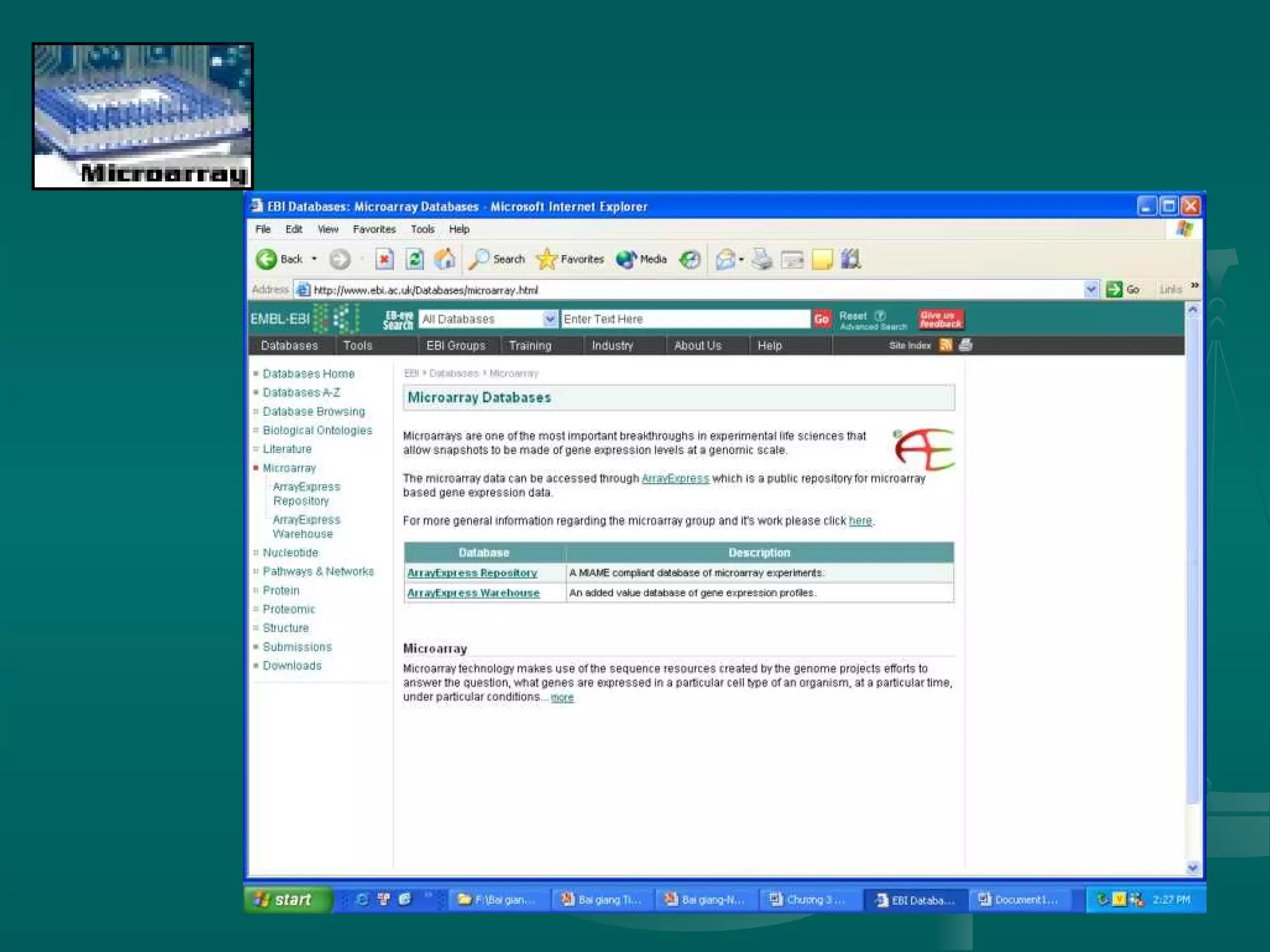
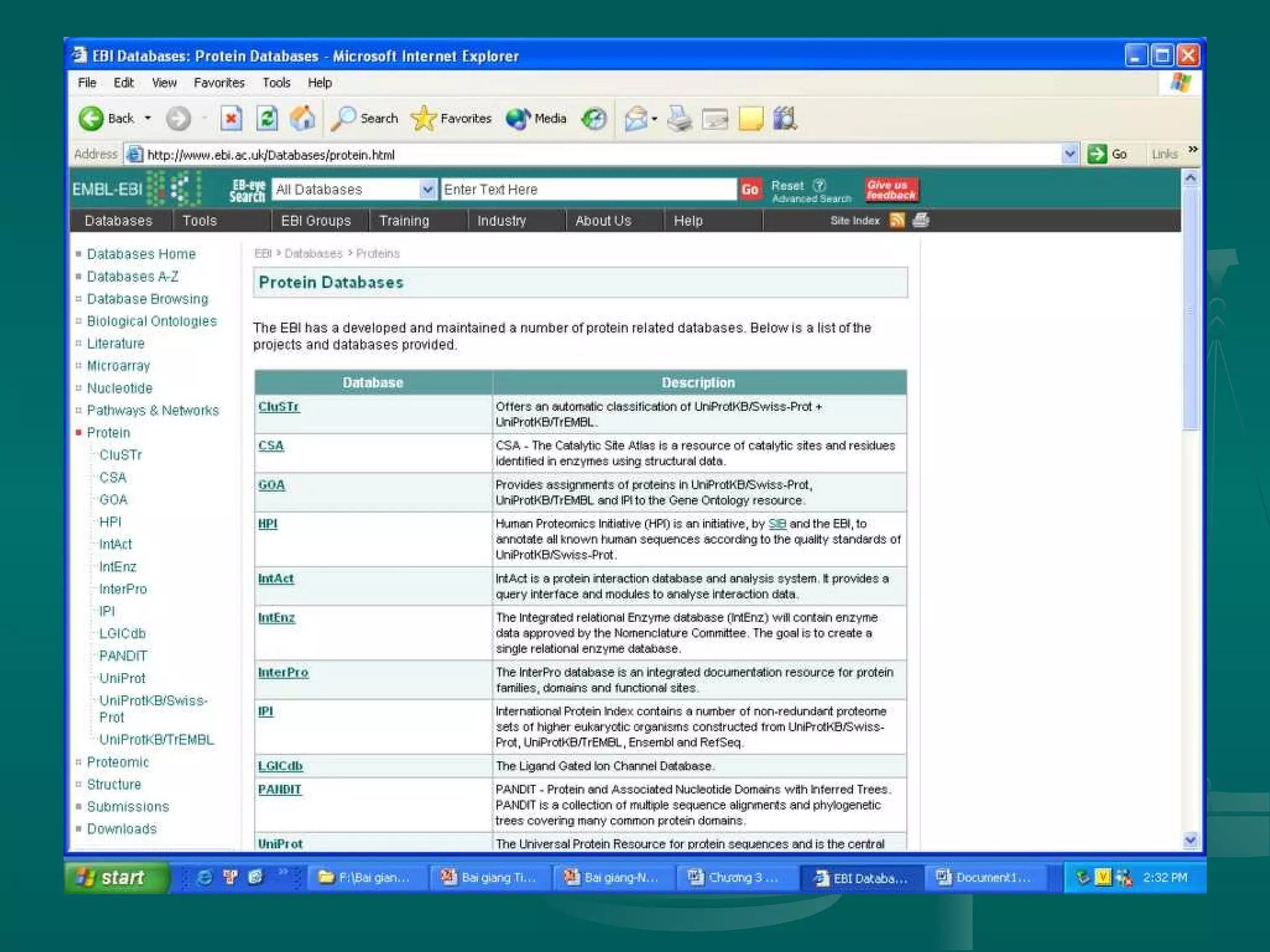
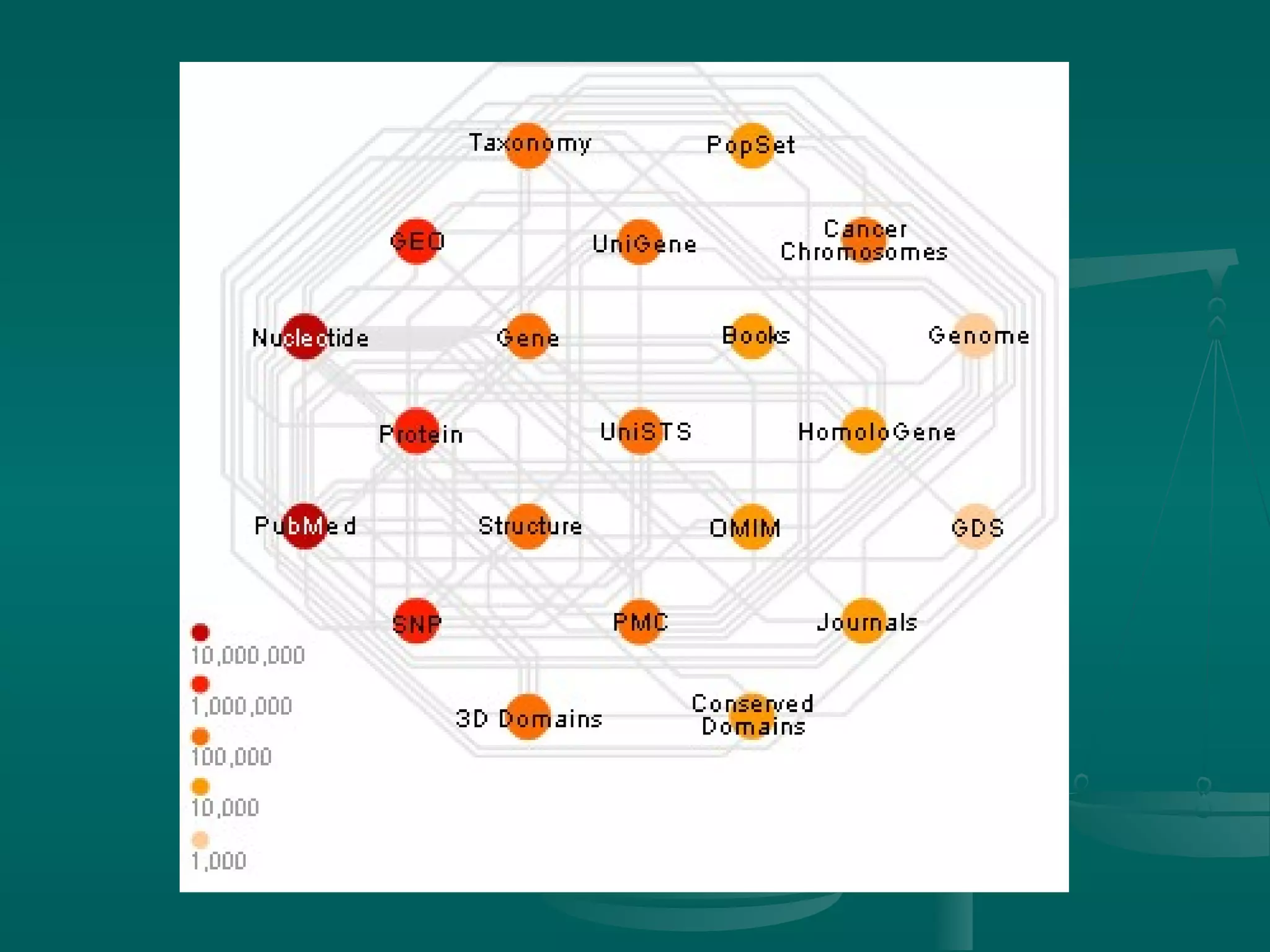
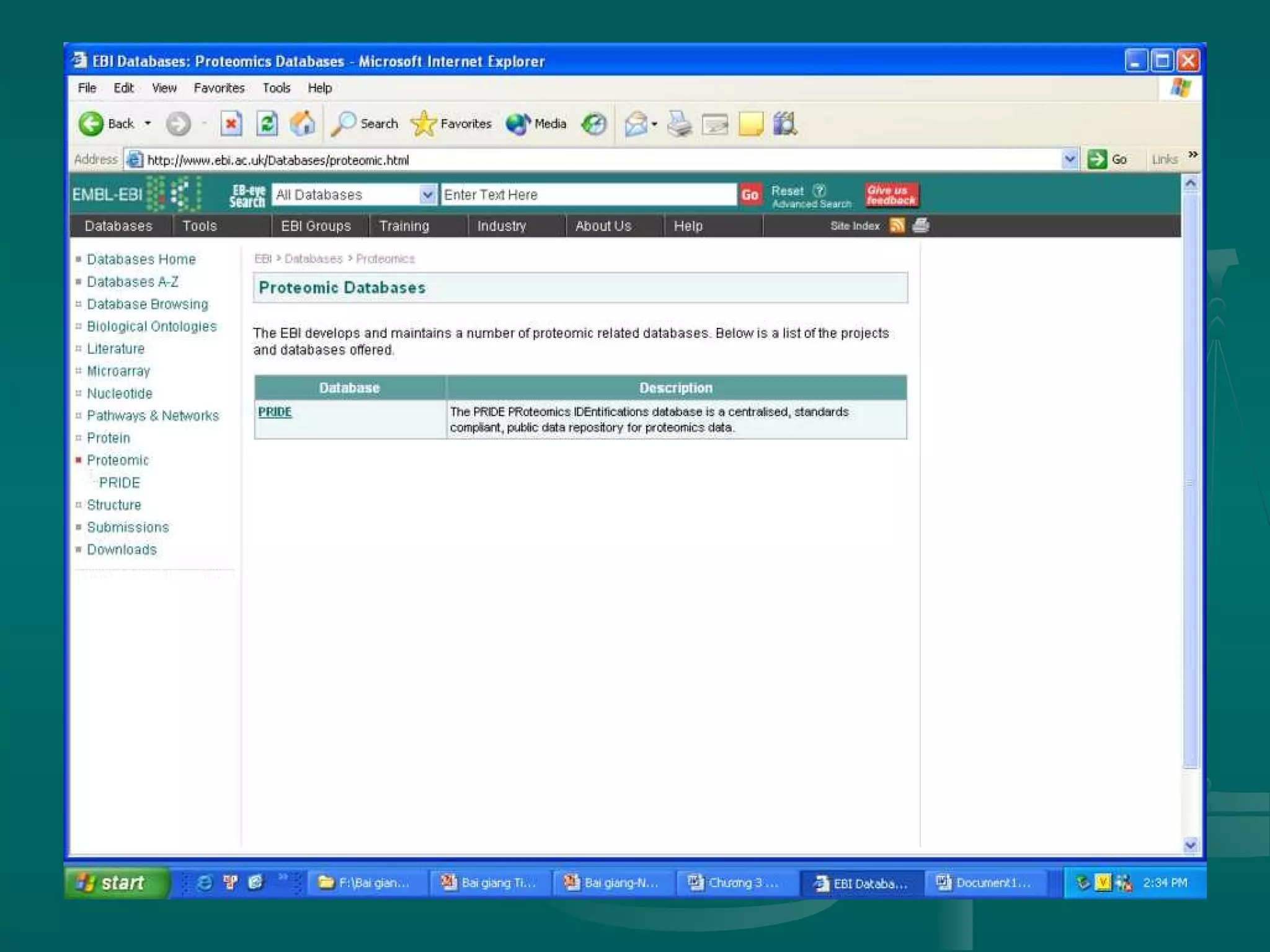
Tài liệu trình bày về sự ra đời của internet, sự hình thành mạng ở Việt Nam và các cấu trúc mạng internet, bao gồm mạng cục bộ, mạng vùng trung tâm và mạng diện rộng. Nó cũng giải thích về hoạt động của WWW và trình duyệt web, cùng các dịch vụ internet và đạo đức khi sử dụng mạng. Cuối cùng, tài liệu nghiêng về tin sinh học, nhấn mạnh sự phát triển của nó, các ngành nghề liên quan, cùng vai trò trong việc quản lý và phân tích dữ liệu sinh học.