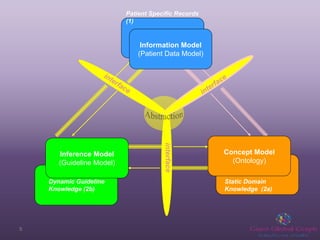
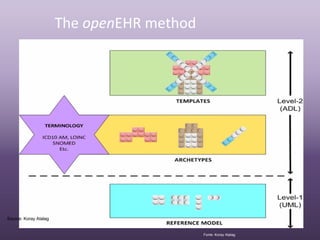
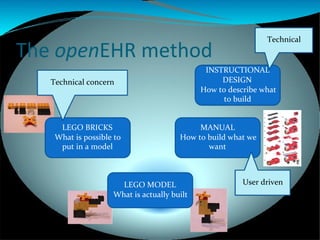
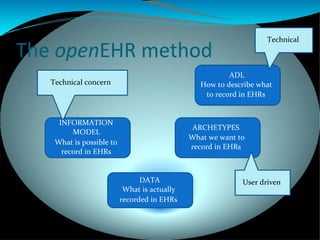
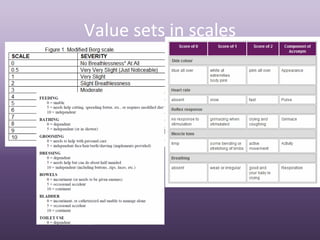
Terminologies play an important role in openEHR by enabling semantic interoperability between electronic health records. OpenEHR uses archetypes to define structured data sets and templates to specify use cases. Terminology bindings associate coded concepts and value sets from reference terminologies to openEHR data points. Different kinds of bindings are used including direct bindings and using local value sets. Future work includes developing context-dependent and compositional bindings.