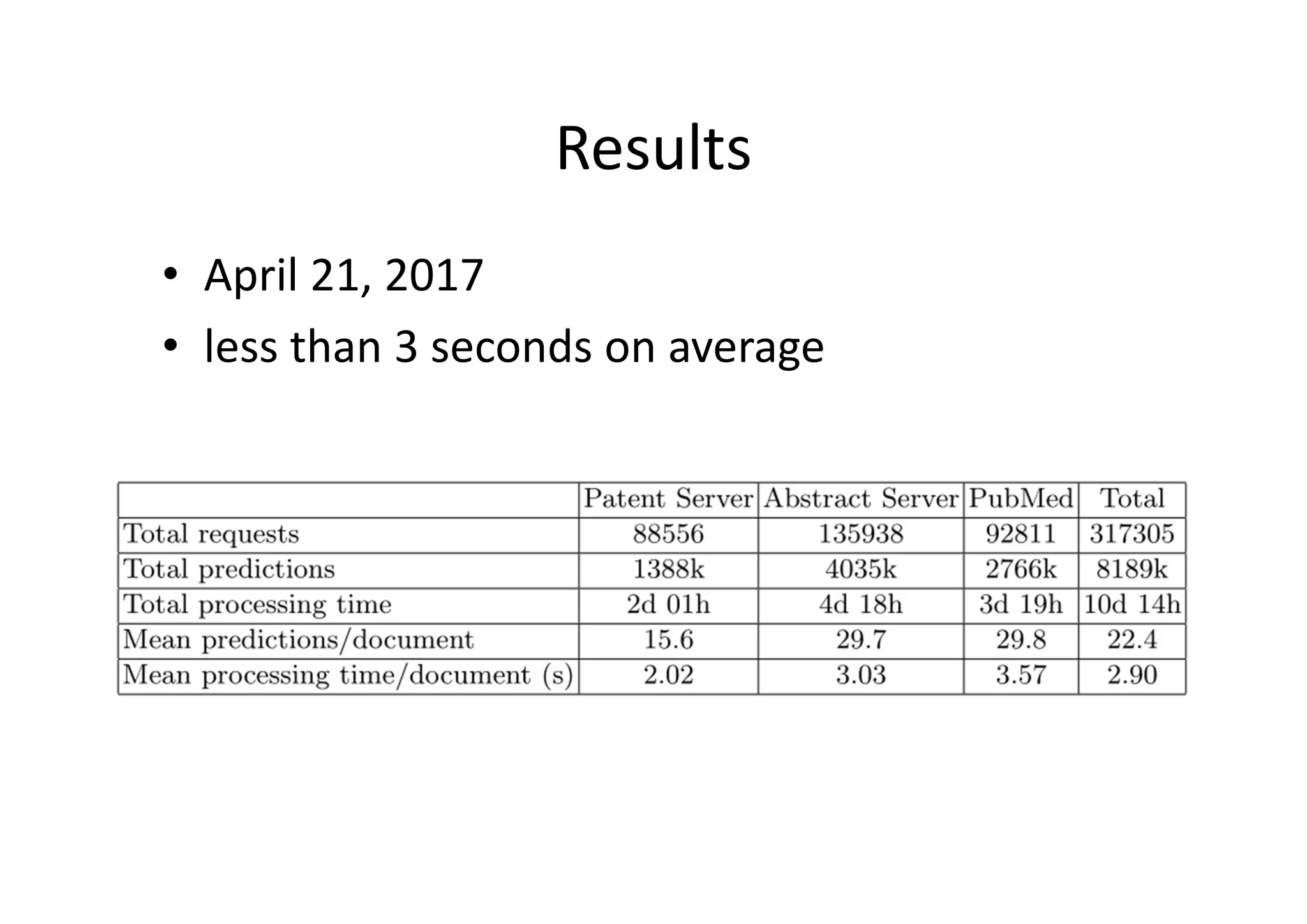
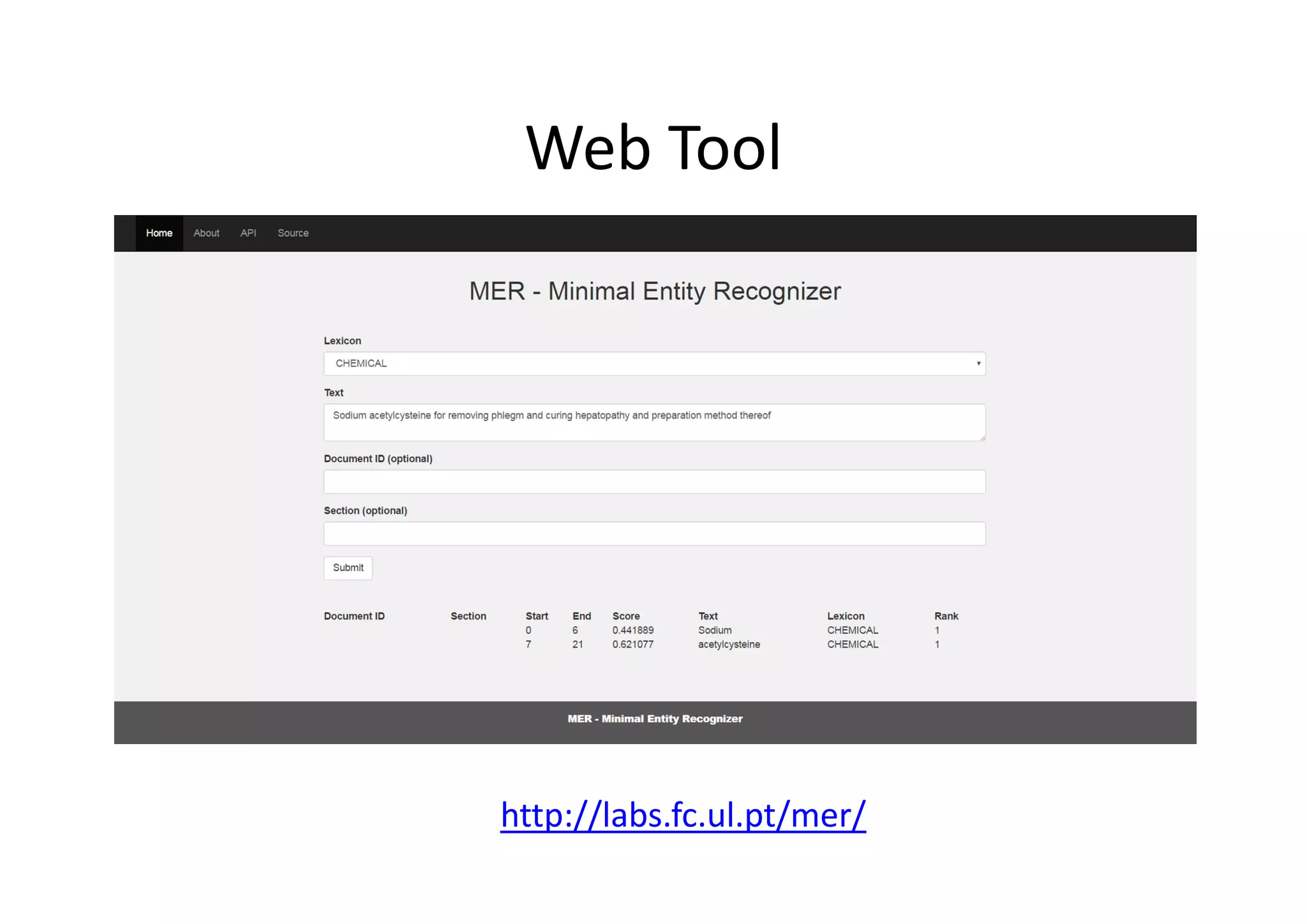
MER is a minimal named-entity recognition tool and annotation server developed by researchers at the University of Lisbon. It uses simple text files as input lexicons and requires only two components - lexicon processing and annotation generation - implemented as a GNU Bash shell script, allowing it to run on any Unix system. The annotation server uses MER to rapidly recognize entities in text, returning results in under 3 seconds on average by matching text to an inverted lexicon of over 1 million terms. The open-source system requires minimal computational resources and software dependencies.