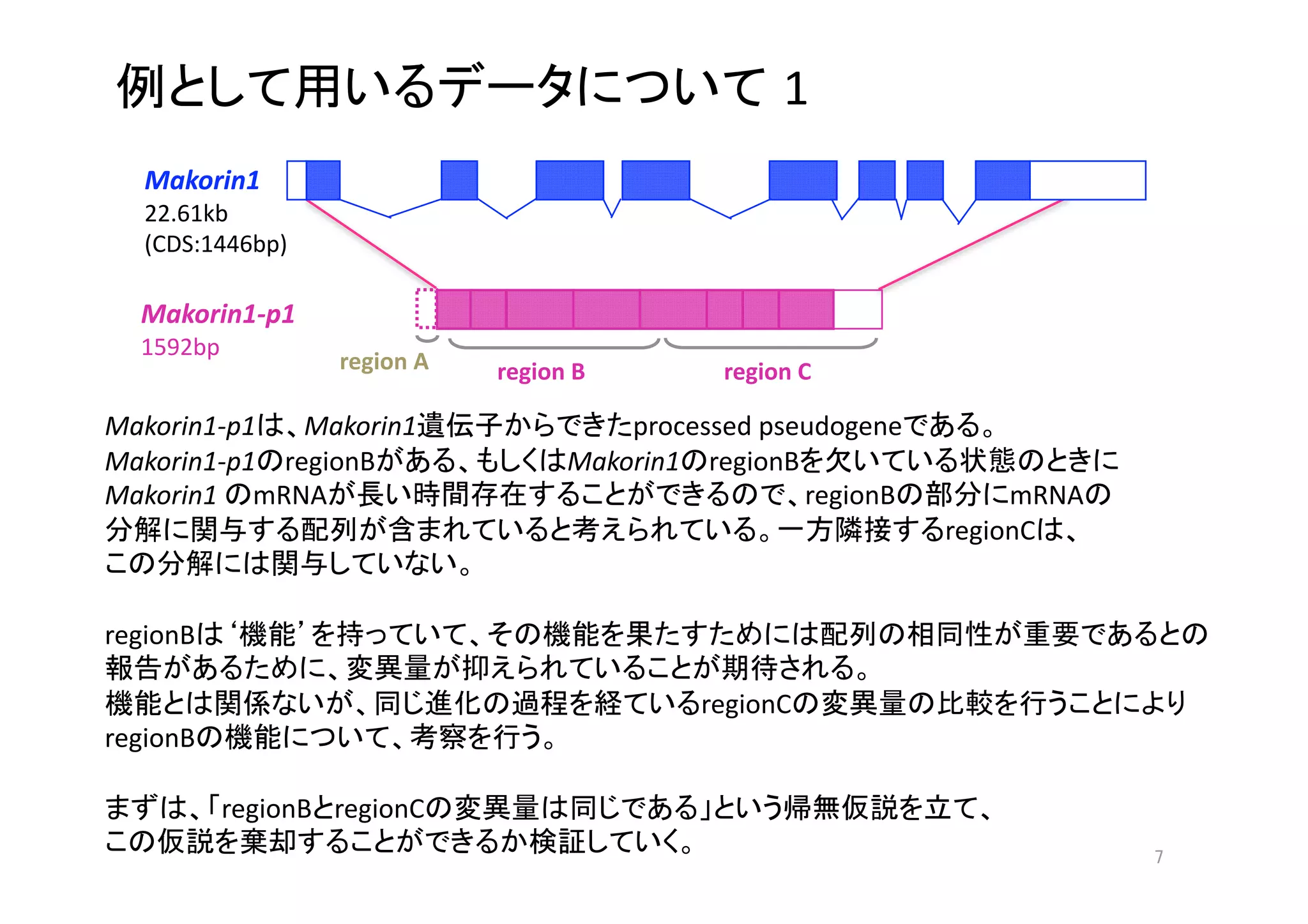
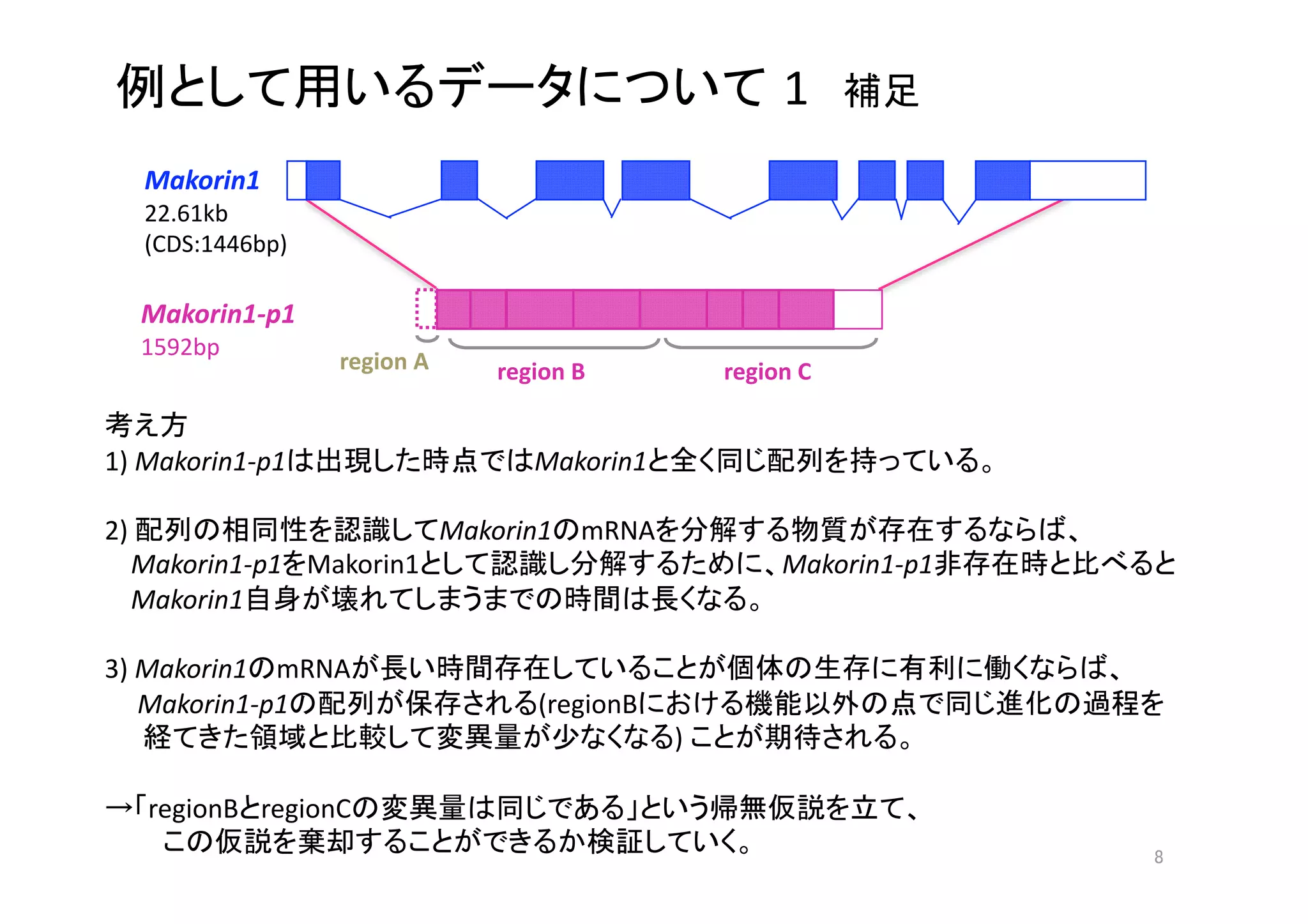
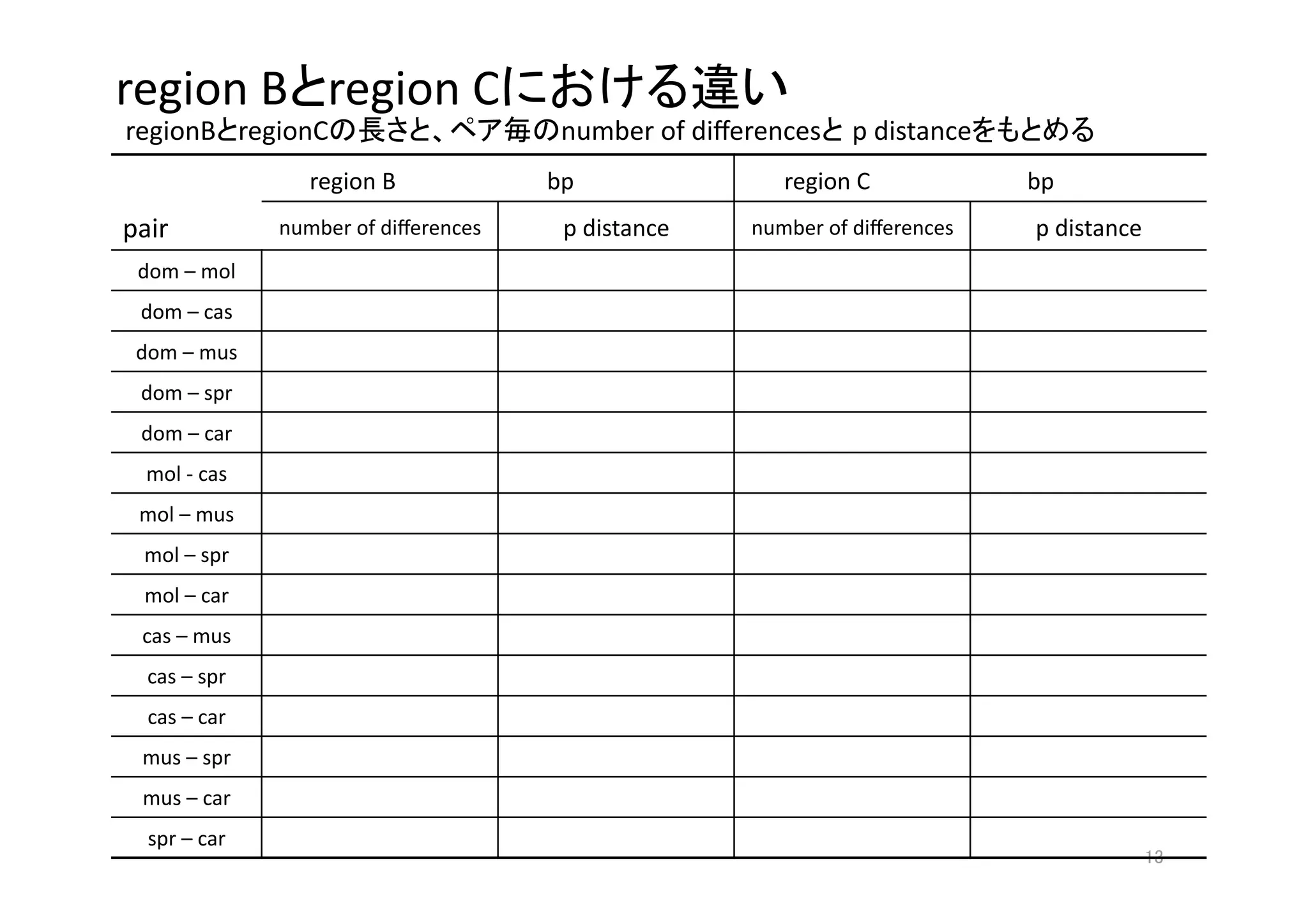
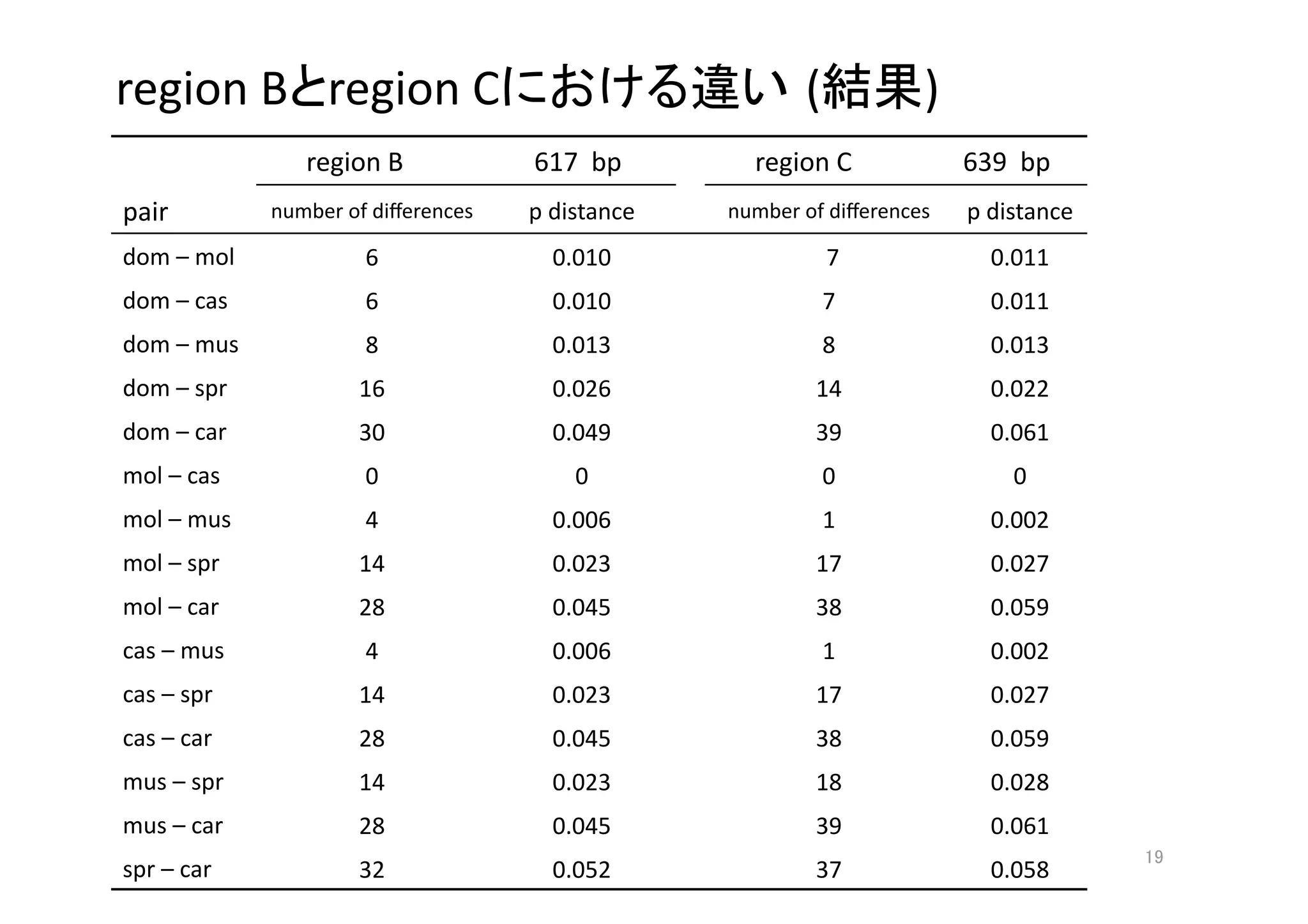
1) The document discusses using the Biostrings package in R to analyze DNA sequences of the Makorin1-p1 pseudogene from different Mus musculus subspecies, calculating the p-distance between sequences in regions B and C.
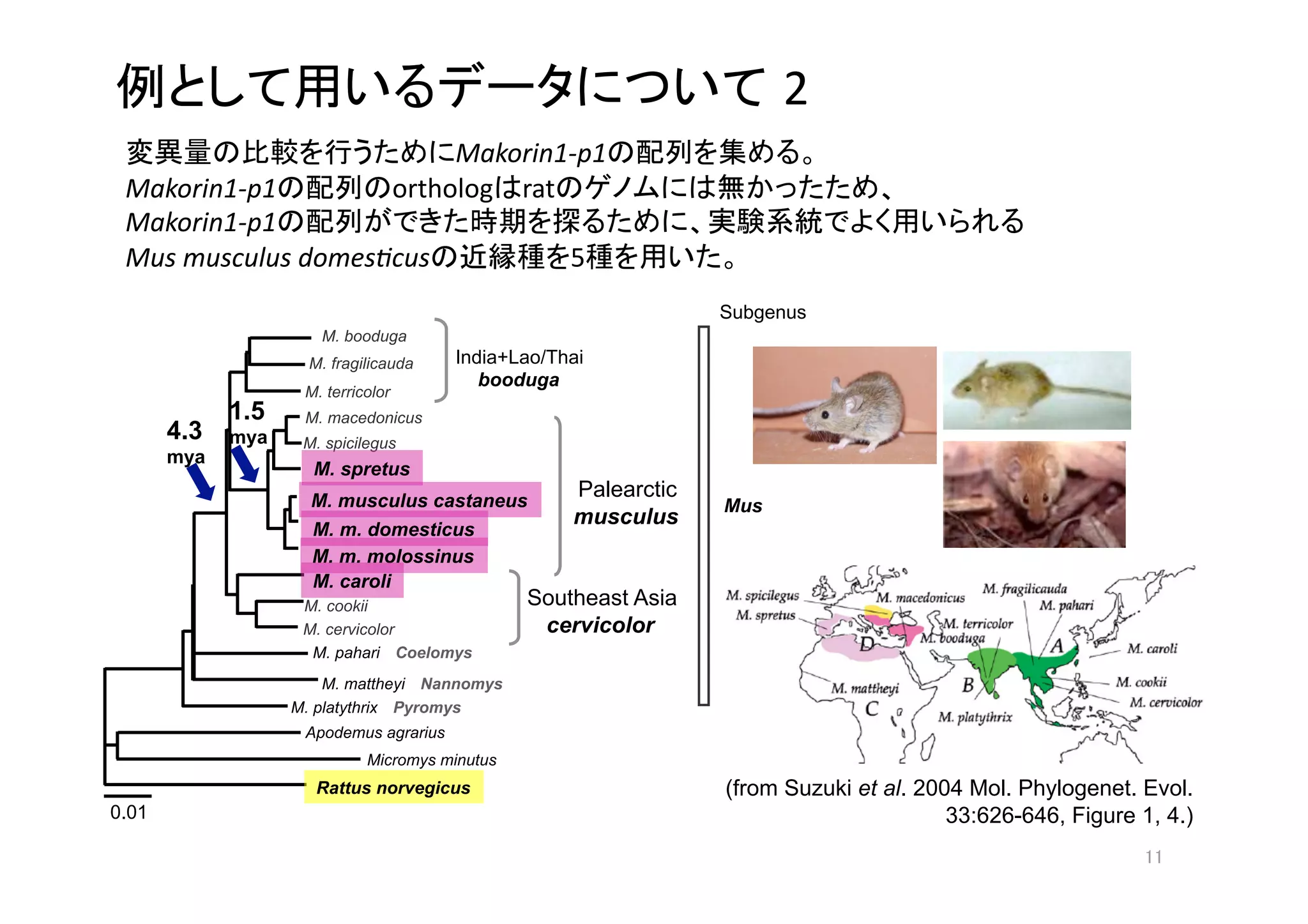
2) A phylogenetic tree shows the evolutionary relationships between different M. musculus subspecies and related species. Makorin1-p1 is found to have an ortholog in rats.
3) The methodology describes plans to use the Biostrings package to extract region B and region C sequences from a Makorin1-p1.fasta file for different M. musculus subspecies, and calculate p



![Bioconductor/Biostrings 1
> ls(“package:Biostrings”) #Biostrings
> x <‐ "CGTACGTAGTAGCTAGCTAGCTAGCTAGCTGATCGATGCTAGCTGATCGATGCT"
> DNAString(x) #DNA
54‐le>er "DNAString" instance
seq: CGTACGTAGTAGCTAGCTAGCTAGCTAGCTGATCGATGCTAGCTGATCGATGCT
> s <‐ DNAString(x) # s DNA (x)
> length(s)
[1] 54 # s DNA 54
> length(x)
[1] 1 # x 1](https://image.slidesharecdn.com/100624-100624060410-phpapp01/75/100624_statistics2-4-2048.jpg)
![Bioconductor/Biostrings 2
54‐le>er "DNAString" instance
seq: CGTACGTAGTAGCTAGCTAGCTAGCTAGCTGATCGATGCTAGCTGATCGATGCT
> alphabetFrequency(s, baseOnly=TRUE) #
A C G T other
[1,] 12 12 15 15 0
> reverseComplement(s) #
54‐le>er "DNAString" instance
seq: AGCATCGATCAGCTAGCATCGATCAGCTAGCTAGCTAGCTAGCTACTACGTACG
> dna2rna(s) #RNA (T ‐>U)
54‐le>er "RNAString" instance
seq: CGUACGUAGUAGCUAGCUAGCUAGCUAGCUGAUCGAUGCUAGCUGAUCGAUGCU](https://image.slidesharecdn.com/100624-100624060410-phpapp01/75/100624_statistics2-5-2048.jpg)
![Bioconductor/Biostrings 3
54‐le>er "DNAString" instance
seq: CGTACGTAGTAGCTAGCTAGCTAGCTAGCTGATCGATGCTAGCTGATCGATGCT
> m1 <‐ matchPa>ern(“GCTA”, s) #
> m1
Views on a 54‐le>er DNAString subject
subject: CGTACGTAGTAGCTAGCTAGCTAGCTAGCTGATCGATGCTAGCTGATCGATGCT
views:
start end width
[1] 12 15 4 [GCTA]
[2] 16 19 4 [GCTA]
[3] 20 23 4 [GCTA]
[4] 24 27 4 [GCTA]
[5] 38 41 4 [GCTA]](https://image.slidesharecdn.com/100624-100624060410-phpapp01/75/100624_statistics2-6-2048.jpg)


![3
Makorin1‐p1.fasta
Mus musculus domes3cus
dom
[Macintosh HD/ /tg03/bin]
Mus musculus molossinus
mol
Mus musculus castaneus
cas
Makorin1‐p1.fasta
Mus musculus musculus
mus
regionB 1‐617, regionC 618‐1256
Mus spretus
spr
Mus caroli
car
p distance
2 (number of differences)/
alignment
dom CCTGCCCCAA ATGTCAGATC ACATCTCACT TTGTCATTCC AAGTAATCAC TGGGTGGAGT
spr1 .......... ...C...... .......... .......... ......GT.. ..........
car1 .......... ...C.GA... ......A... ..T....... ....G.GT.. .........G
dom‐spr1: 3/60 = 0.05
dom‐car1: 9/60 = 0.15
spr1‐car1: 6/60 = 0.10](https://image.slidesharecdn.com/100624-100624060410-phpapp01/75/100624_statistics2-12-2048.jpg)

![Biostrings p distance 1
library(Biostrings) #R
#Makorin1‐p1.fasta domesgcus ””
# DNA dom
> dom <‐ DNAString("")
#dom 1 617 domB
> domB <‐ substring(dom,1,617)
#domB lengthB (p distance )
> lengthB <‐ length(domB)
#dom 618 1256 domC
> domC <‐ substring(dom, 618,1256)
#domC lengthC (p distance )
> lengthC <‐ length(domC)
#
# lengthB
> lengthB
[1] 617](https://image.slidesharecdn.com/100624-100624060410-phpapp01/75/100624_statistics2-15-2048.jpg)

![Biostrings p distance 3
> x <‐ domB
> y <‐ molB
# x y( domB molB)
> comp<‐ c(compareStrings(x,y))
# ? ?
> subt <‐ gsub("(['?'])", "", comp)
#subt DNA ide
> ide <‐ DNAString(subt)
#ide len
> len <‐ length(ide)
#x y dif
> dif <‐ (lengthB – len )
> dif #x y
#regionB p distance
> pdis <‐ dif/lengthB
> pdis #p distance 4](https://image.slidesharecdn.com/100624-100624060410-phpapp01/75/100624_statistics2-17-2048.jpg)
![Biostrings p distance 4
CotEditor pdistanceB.R
[Macintosh HD/ /tg03/bin]
comp <‐ c(compareStrings(x,y))
2 lengthB lengthC
subt <‐ gsub("(['?'])", "", comp)
pdistanceC.R bin
ide <‐ DNAString(subt)
len <‐ length(ide)
dif <‐ (lengthB ‐ len)
pdis <‐ dif/lengthB
x y
> x <‐
> y <‐
> source("/Users/tg03/bin/pdistanceB.R")
bin
> source("pdistanceB.R")
2
> dif
> pdis](https://image.slidesharecdn.com/100624-100624060410-phpapp01/75/100624_statistics2-18-2048.jpg)

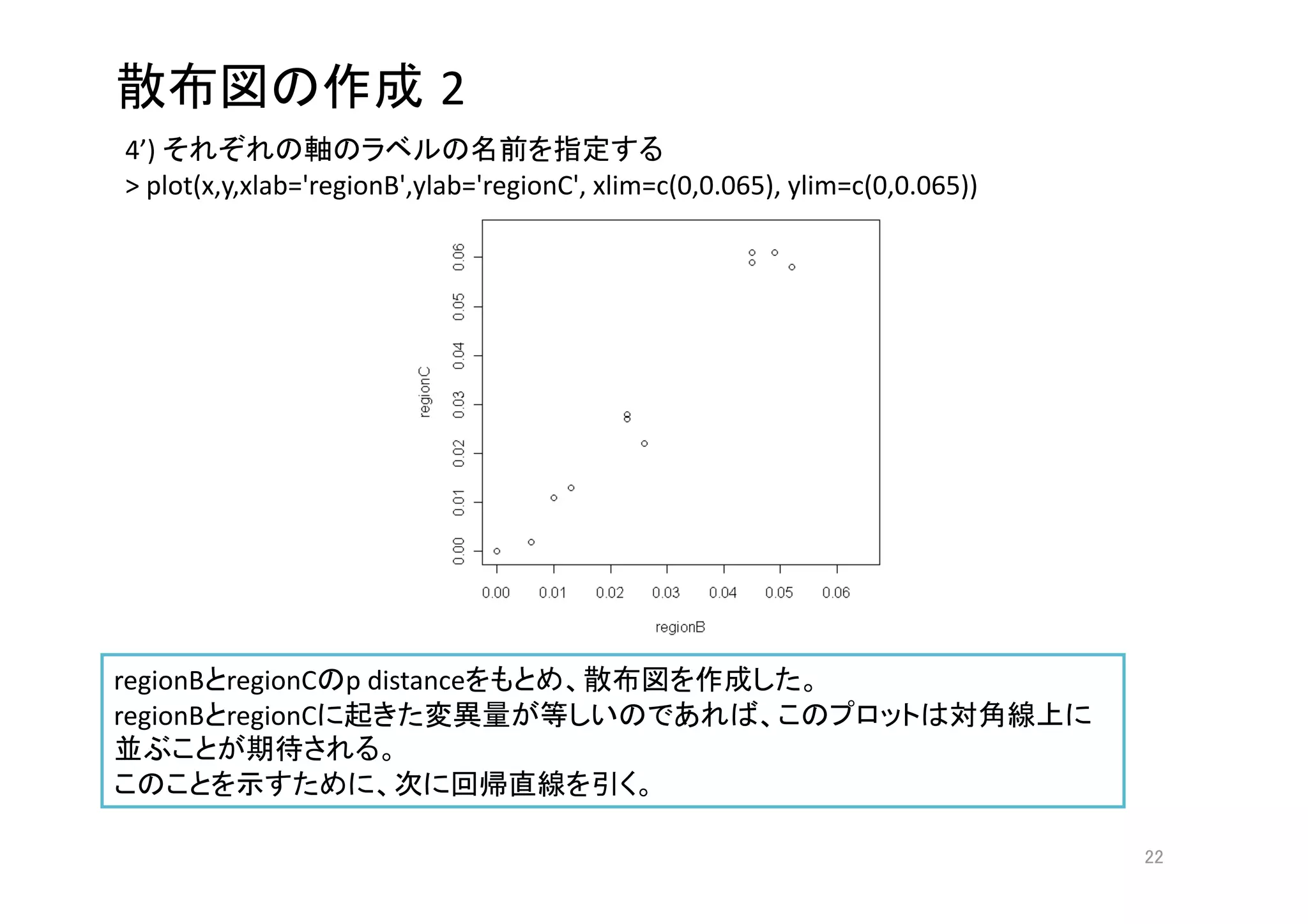
![1
1) regionB p distance x x
> x <‐ c(0.010, 0.010, 0.013, 0.026, 0.049, 0, 0.006 ,0.023, 0.045, 0.006 , 0.023 , 0.045,
0.023, 0.045, 0.052 )
2) regionC p distance y y
> y <‐ c(0.011, 0.011, 0.013, 0.022, 0.061, 0, 0.002, 0.027, 0.059, 0.002, 0.027, 0.059,
0.028, 0.061, 0.058 )
3) max() x y
> max(x)
[1] 0.052
> max(y)
[1] 0.061
4) plot()
> plot(x,y,xlim=c(0,0.065), ylim=c(0,0.065))](https://image.slidesharecdn.com/100624-100624060410-phpapp01/75/100624_statistics2-21-2048.jpg)
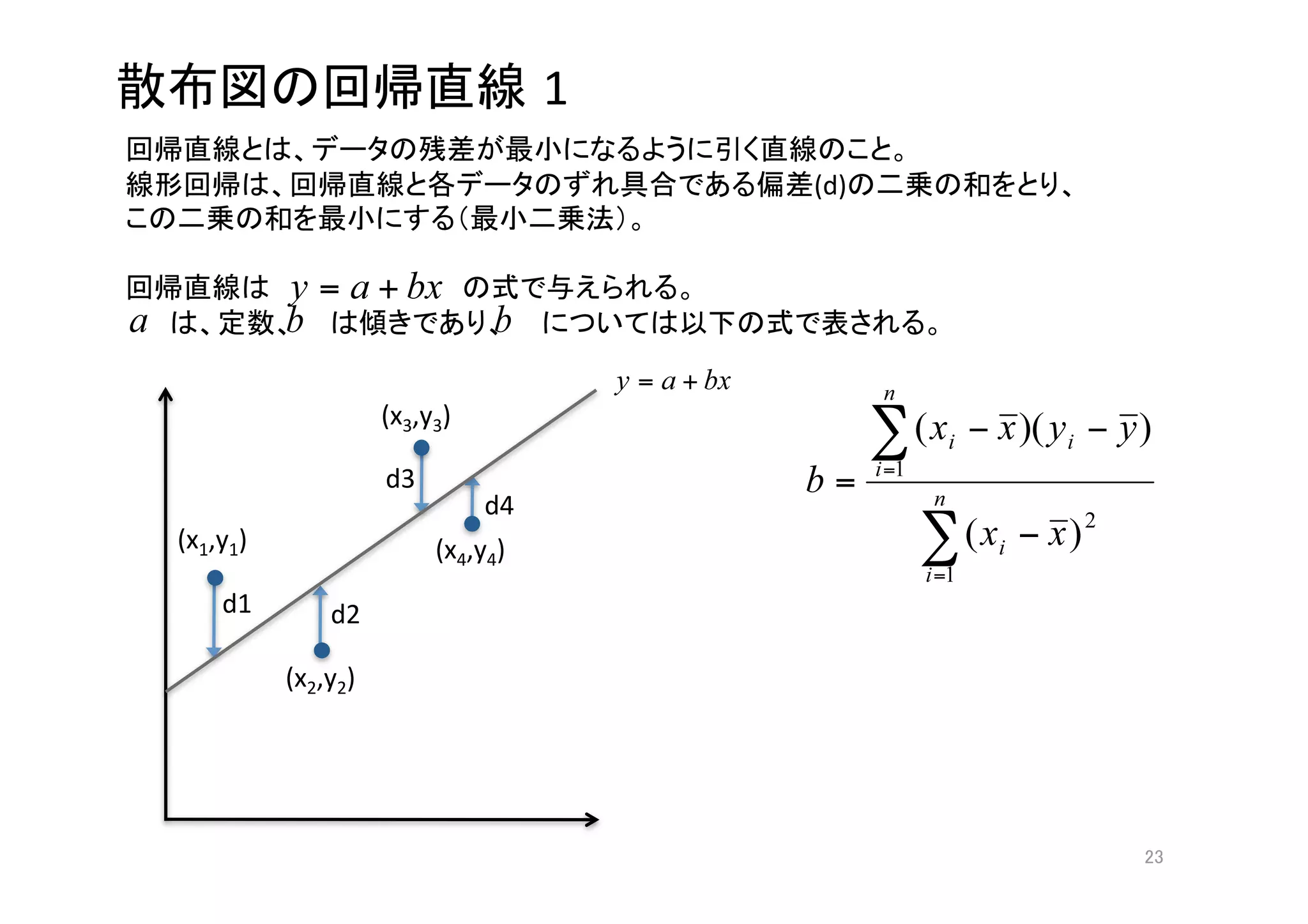
![2
> xdev <‐ (x‐mean(x)) # x
> ydev <‐ (y‐mean(y)) # y
> bmul<‐ xdev*ydev # x y
> bnum <‐ sum(bmul) # x y ( )
> bsqu <‐ xdev^2 # x 2
> bden <‐ sum(bsqu) # x 2 ( )
> b <‐ bnum/bden # ( )
> b
[1] 1.317939](https://image.slidesharecdn.com/100624-100624060410-phpapp01/75/100624_statistics2-24-2048.jpg)
![3
> a1 <‐ sum(y)/length(y)
> a2 <‐ b*(sum(x)/length(x))
> a <‐ a1‐a2
[1] ‐0.003636326
> abline (a,b)
#a b y = a + bx](https://image.slidesharecdn.com/100624-100624060410-phpapp01/75/100624_statistics2-25-2048.jpg)
![p distance 1
‐pdis_line.R‐
library("Biostrings");
x <‐""
dom <‐ "[domesgcus ]";
mol <‐ "[molossinus ]";
cas <‐ "[castaneus ]";
mus <‐ "[musculus ]";
spr <‐ "[spretus ]";
car <‐ "[caroli ]";
seqs <‐ c(dom,mol,cas,mus,spr,car);
seqnames <‐ c("dom","mol","cas","mus","spr","car");
nseqs <‐ length(seqs);
npoints <‐ length(x);
x = vector(length=npoints);
y = vector(length=npoints);
k = 0;](https://image.slidesharecdn.com/100624-100624060410-phpapp01/75/100624_statistics2-27-2048.jpg)
![p distance 2
for (i1 in 1:(nseqs‐1)){
for (i2 in (i1+1):nseqs ){
k = k + 1;
# cat(sprint("%d %dn",i1,i2));
seq1 = DNAString(seqs[i1]);
seq2 = DNAString(seqs[i2]);
seq_b1 = substring( seq1, 1, 617 );
seq_c1 = substring( seq1, 618, 1256 );
seq_b2 = substring( seq2, 1, 617 );
seq_c2 = substring( seq2, 618, 1256 );
len_b = length( seq_b1 );
cmp_b = c(compareStrings(seq_b1,seq_b2));
sub_b = gsub("(['?'])","",cmp_b);
subt_b = DNAString(sub_b);
dif_b = length(subt_b);
n_b = len_b ‐ dif_b;
pdis_b = n_b / len_b;](https://image.slidesharecdn.com/100624-100624060410-phpapp01/75/100624_statistics2-28-2048.jpg)
![p distance 3
‐pdis_line.R‐
x[k] = pdis_b;
len_c = length( seq_c1 );
cmp_c = c(compareStrings(seq_c1,seq_c2));
sub_c = gsub("(['?'])","",cmp_c);
subt_c = DNAString(sub_c);
dif_c = length(subt_c);
n_c = len_c ‐ dif_c;
pdis_c = n_c / len_c;
y[k] = pdis_c;
cat(sprint('%s %s %d %g %gn',seqnames[i1],seqnames[i2],k,pdis_b,pdis_c));
}
}
xdev <‐ x‐mean(x);
ydev <‐ y‐mean(y);
b <‐ sum(xdev*ydev)/sum(xdev*xdev);
a <‐ mean(y) ‐ b*mean(x);
cat(sprint('a=%g, b=%gn',a,b));](https://image.slidesharecdn.com/100624-100624060410-phpapp01/75/100624_statistics2-29-2048.jpg)