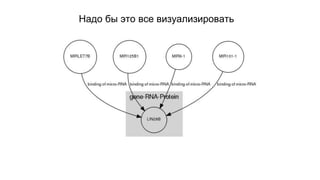
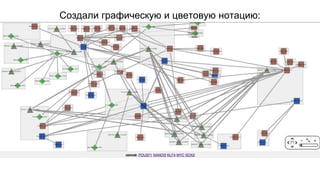
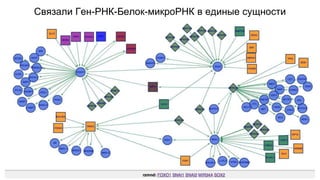
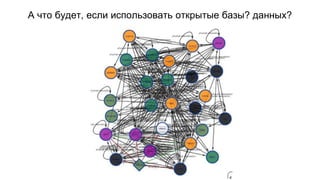
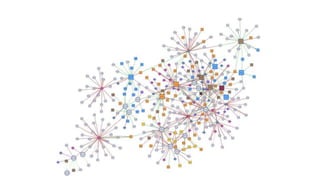
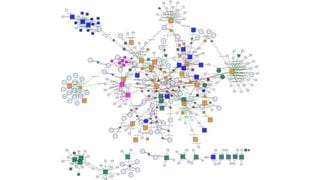
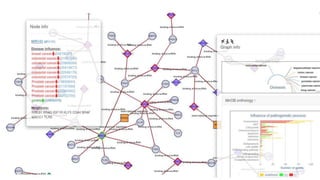
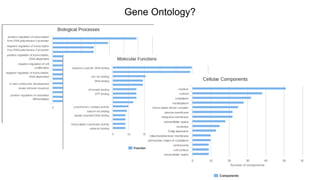
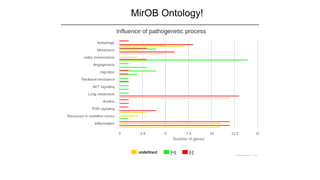
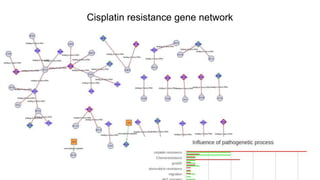
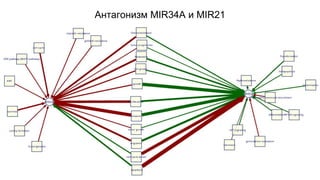
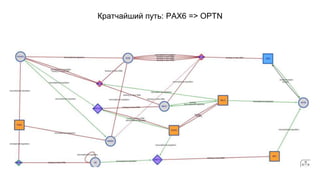
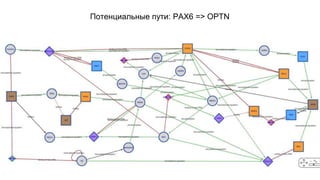
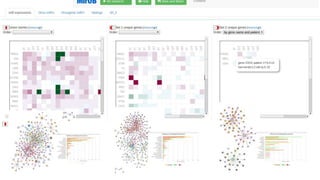
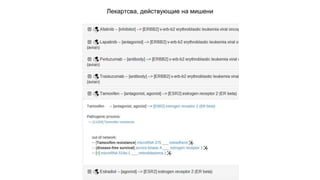
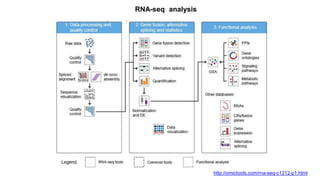
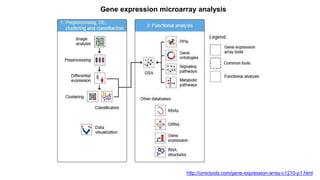
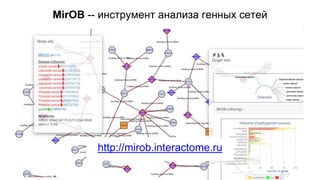
Документ посвящен анализу и визуализации генных сетей, подчеркивая важность координированного взаимодействия генов и их продуктов для выполнения функций организма. В нем обсуждаются необходимые инструменты и базы данных для работы с генными сетями, а также предлагаются решения для упрощения обработки и представления информации. Также описываются планы по динамической визуализации и анализу данных о генных сетях, включая гайд-лайны по лечению онкозаболеваний.