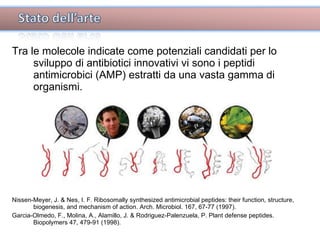
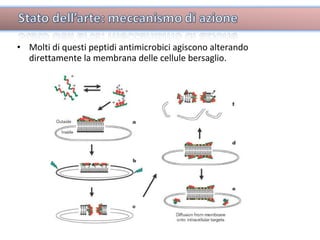
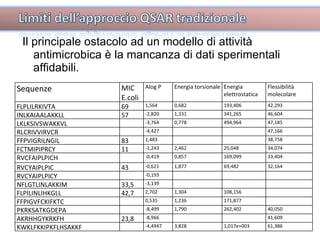
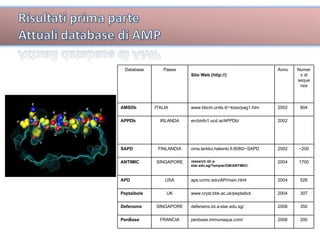
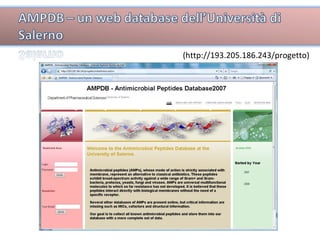
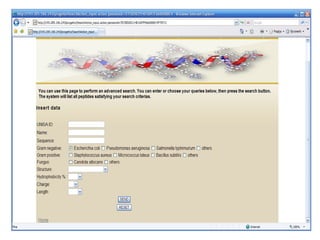
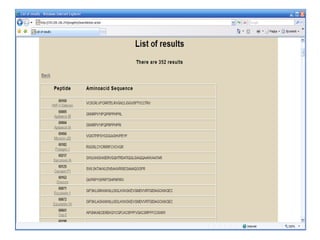
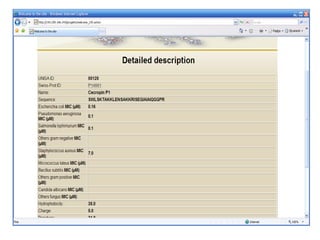
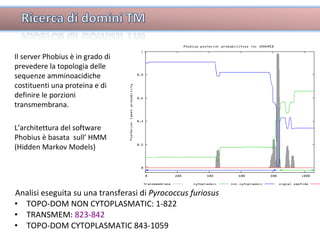
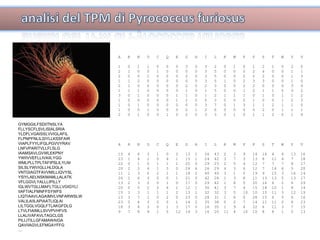
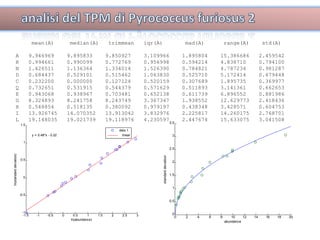
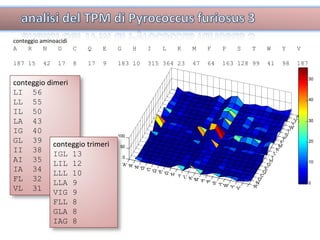
Il documento tratta lo sviluppo di nuovi antibiotici attraverso peptidi antimicrobici (AMP) e l'uso di tecniche QSAR per analizzarne l'attività. Viene enfatizzata l'importanza di raccogliere dati affidabili e sviluppare un knowledge base su AMP per progettare nuove molecole. L'analisi delle sequenze transmembrana e la correlazione con la tossicità sono cruciali per definire modelli predittivi di attività antimicrobica.