Report
Share
Download to read offline
Recommended
More Related Content
More from Satoshi Kume
More from Satoshi Kume (20)
CRANパッケージの作成・投稿とコードレビュー対応 〜 GoogleImage2Array パッケージでの事例紹介 〜 @ BioPackathon

CRANパッケージの作成・投稿とコードレビュー対応 〜 GoogleImage2Array パッケージでの事例紹介 〜 @ BioPackathon
210609 Biopackthon: BioImageDbs for ExperimentalHub (修正版)

210609 Biopackthon: BioImageDbs for ExperimentalHub (修正版)
Recently uploaded
Recently uploaded (7)
ゲーム理論 BASIC 演習106 -価格の交渉ゲーム-#ゲーム理論 #gametheory #数学

ゲーム理論 BASIC 演習106 -価格の交渉ゲーム-#ゲーム理論 #gametheory #数学
生成AIの回答内容の修正を課題としたレポートについて:お茶の水女子大学「授業・研究における生成系AIの活用事例」での講演資料

生成AIの回答内容の修正を課題としたレポートについて:お茶の水女子大学「授業・研究における生成系AIの活用事例」での講演資料
UniProject Workshop Make a Discord Bot with JavaScript

UniProject Workshop Make a Discord Bot with JavaScript
ゲーム理論 BASIC 演習105 -n人囚人のジレンマモデル- #ゲーム理論 #gametheory #数学

ゲーム理論 BASIC 演習105 -n人囚人のジレンマモデル- #ゲーム理論 #gametheory #数学
qPCR_primer_construction_ver_1.0
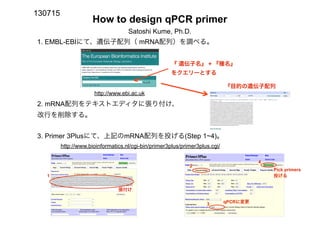
- 1. 1. EMBL-EBIにて、遺伝子配列( mRNA配列)を調べる。 2. mRNA配列をテキストエディタに張り付け、 改行を削除する。 3. Primer 3Plusにて、上記のmRNA配列を投げる(Step 1~4)。 How to design qPCR primer 130715 http://www.ebi.ac.uk 『 遺伝子名』 + 『種名』 をクエリーとする http://www.bioinformatics.nl/cgi-bin/primer3plus/primer3plus.cgi/ 張付け qPCRに変更 投げる Satoshi Kume, Ph.D. Pick primers 1 2 3 4 『目的の遺伝子配列
- 2. 4. Primer-BLAST in NCBI (※)右のような結果が得られる。 ココ! Primer配列の張付け! 種の選択(例: rat) 実行! Primer配列 5. 増幅断片数の確認 増幅候補は1つであることが必須! qPCRのProduct lengthは、150 ~ 300がBetter。 長さOK! 候補OK! http://www.ncbi.nlm.nih.gov/tools/primer-blast/index.cgi?LINK_LOC=BlastHome NCBI → BLAST → Specialized BLAST → Primer-BLAST 1 2 3 4
- 3. → 違う候補を探す。 ● Primerの3’末端から5 bpのうち、G/Cが4 bp以上だと、良くない! ● 3’末端最後の塩基は、GかCにする。 E1 E2 E3 Gene mRNA E1 E2 E3 Forward Reverse Transcription 理想型Primer ● 理想的には、下図のようにエキソンとエキソンの間でPrimerを設計する。 5. qPCR primerの最終チェック → 違う場合は、Primerを伸ばすか削るかして対処する。 ● Tm(60 ℃くらい)は、Primer間でできる限り える。 EMBL-EBIにて調べられる。 エキソンが1個だけなら、 考えなくてよい。
