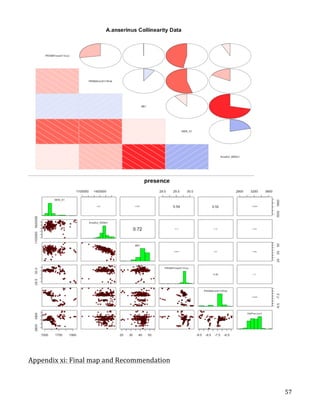
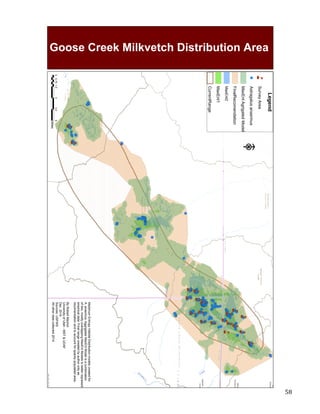
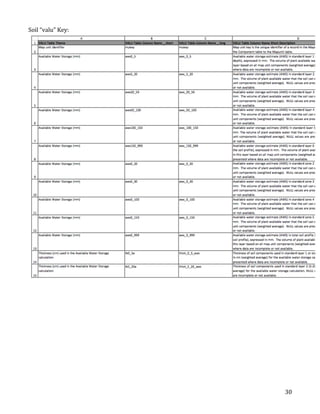
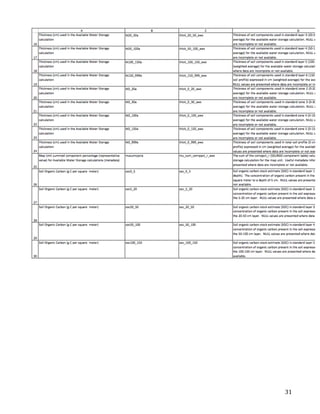
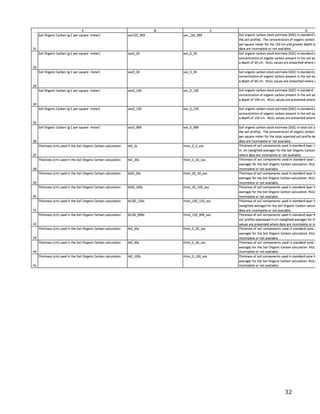
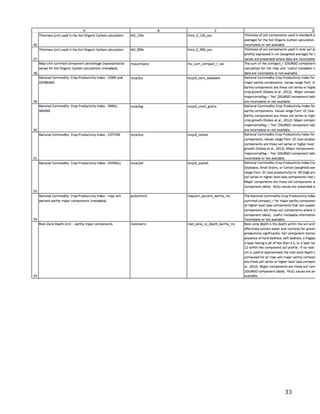
This document describes the creation of habitat distribution models for the endemic plant species Astragalis anserinus. The author collected environmental data for the species' range and used MaxEnt modeling in the R statistical programming language to create two habitat distribution models, MaxEnt1 and MaxEnt2. Both models were evaluated using metrics like AUC. A combination of the two models was used to make a final recommended habitat map. However, the author notes that some presence data points may be inaccurate and require field verification. An improved model incorporating verified data is recommended. The modeling provides a cost-effective way to predict habitat compared to solely using fieldwork.























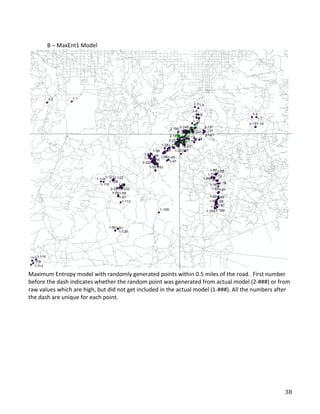








![51
Appendix
ix:
Sample
R
code
####
MaxEnt
Code
Example
####
Robert
Machol
####
UofU
-‐
MST
&
UDAF
####
Ded
2014
##Set
working
directory
setwd("~/Documents/MST/Summer
2014/Final
Project")
##Load
dismo
library
library("dismo",
lib.loc="/Library/Frameworks/R.framework/Versions/3.1/Resources/library")
##Load
maptools
library("maptools",
lib.loc="/Library/Frameworks/R.framework/Versions/3.1/Resources/library")
##rgdal
library("rgdal",
lib.loc="/Library/Frameworks/R.framework/Versions/3.1/Resources/library")
##
proj4
spatial
plotting
library("proj4",
lib.loc="/Library/Frameworks/R.framework/Versions/3.1/Resources/library")
##scales-‐
for
transparnt
plotting
library("scales",
lib.loc="/Library/Frameworks/R.framework/Versions/3.1/Resources/library")
##Corgram
to
view
colinearity
library("corrgram",
lib.loc="/Library/Frameworks/R.framework/Versions/3.1/Resources/library")
##MASS
to
write
Matrix
library("MASS",
lib.loc="/Library/Frameworks/R.framework/Versions/3.1/Resources/library")
#####Load
rasters
#####
##
cut
from
171
rasters
to
6
according
to
'DataFileList.xlsx'
##
@
30m
resolution
dem
=
raster("RasterOutput/DEM_S1.tif")
SolEnergy=raster("RasterOutput/AreaSol_DEMs1.tif")
IMI=raster("RasterOutput/IMI1.tif")
TmaxJul8110=raster("RasterOutput/PRISMTmax8110Jul.tif")
tmin8110Feb
=raster("RasterOutput/PRISMtmin8110Feb.tif")
HisPrecJun
=
raster("RasterOutput/HisPrecJun1.tif")
#
Envirofiles
Stack
envirofiles
=
(c(dem,SolEnergy,IMI,TmaxJul8110,tmin8110Feb,HisPrecJun))
enviropredictors=(stack(envirofiles))
summary(enviropredictors)
#####Load
Points######
##
Presence
points:
Points
=
read.csv("PresentFinal.csv")
Points
##
I
only
want
the
coordinates...
Pointsxy
=
Points[,1:2]
Pointsxy
#787
points
##Absence
Points:
Absence
=
read.csv("NegsFinal.csv")
Absencexy
=
Absence[,1:2]
Absencexy
#362
points
#tail(Absencexy)
####
Points
&
Coordinate
Metadata:
####](https://image.slidesharecdn.com/4f0642fe-ff34-400e-9d28-12ccf8090a4b-150407130702-conversion-gate01/85/MacholInternship5-51-320.jpg)
![52
##cooridinates
for
study
area:
42.47,41.24
&
-‐114.88,-‐113.42
##Geographic
Coordinate
System:
GCS_WGS_1984
##Datum:
D_WGS_1984
##Prime
Meridian:
Greenwich
##Angular
Unit:
Degree
####
Plot
Points:
####
par=(mfrow=c(1,1))
PointsCRS
=
CRS("+proj=longlat
+ellps=WGS84")
Points.sp
=
SpatialPoints(cbind(Pointsxy$XCoord,
Pointsxy$YCoord),
proj4string=PointsCRS)
summary(Points.sp)
bbox(Points.sp)
proj4string(Points.sp)
coordinates(Points.sp)
Points.spdf
=
SpatialPointsDataFrame(cbind(Pointsxy$XCoord,
Pointsxy$YCoord),
Pointsxy,
proj4string
=
PointsCRS)
summary(Points.spdf)
plot(Points.spdf)
###
General
visualization
of
point
data
###
##USA
Download
USA
=
getData('GADM',
country='USA',
level
=
1)
Absence.spdf
=
SpatialPointsDataFrame(cbind(Absencexy$XCoord,
Absencexy$YCoord),
Absence,
proj4string
=
PointsCRS)
plot(USA,
xlim=c(-‐114.45,
-‐113.9),
ylim=c(41.6,42.1),
axes=TRUE)#Point
Coordinates
points(Pointsxy$XCoord,
Pointsxy$YCoord,
col=alpha("green",
0.3),
cex=.5,
pch=3)#Include
accual
and
random
from
survey
plots
points(Absencexy$XCoord,
Absence$YCoord,
col=alpha("red",
0.3),
cex=.5,
pch=4)#Include
accual
and
random
from
survey
plots
legend("topleft",
legend
=
c("Presence
-‐
787
points",
"Absence
-‐
362
points"),
col
=
c("green",
"red"),
pch
=
c(3,4)
)
title(
main="A.anserinus
Point
Data")
##All
predictors
plot
(enviropredictors)
#######
Extract
Values
From
Rasters
Via
Points
###########
##Presence
Values:
presvals=extract(enviropredictors,Pointsxy)#(RnR)
##Absence
VAlues
absvals=extract(enviropredictors,Absencexy)#(RnR)
##for
presence
or
absence
points,
we
will
call
"pb"
(new
variable,
0
absence
points,
1
presence
points)
pb
=c(rep(1,nrow(presvals)),
rep(0,nrow(absvals)))
##combine
these
points
into
a
singel
data.frame
admdata=data.frame(cbind(pb,rbind(presvals,absvals)))
names(admdata)
summary(admdata)
##vissually
check
for
Collinearity:
##Plot
data
in
pairs
pairs(admdata[,2:6],cex=0.1,
fig=TRUE)
library("corrgram",
lib.loc="/Library/Frameworks/R.framework/Versions/3.1/Resources/library")
par(mfrow=c(1,1))](https://image.slidesharecdn.com/4f0642fe-ff34-400e-9d28-12ccf8090a4b-150407130702-conversion-gate01/85/MacholInternship5-52-320.jpg)
![53
corrgram(admdata[,2:6],
order=TRUE,
lower.panel=panel.shade,
upper.panel=panel.pie,
text.panel=panel.txt,
main="A.anserinus
Collinearity
Data")
corrgram(admdata[,2:6],
order=TRUE,
lower.panel=panel.ellipse,
upper.panel=panel.pts,
text.panel=panel.txt,
diag.panel=panel.minmax,
main="A.anserinus
Collinearity
Data")
####Model
Fitting:
Generalized
Linear
Model(glm)####
gc()
#clear
garbage
from
memmory
M2
=
glm(formula=pb~.,data=admdata)
summary(M2)
##Bioclim
only
takes
presence
values
bc=
bioclim(presvals)
pairs(bc)
##Predicting
GLM
pM2
=
predict(enviropredictors,
M2,
na.rm=TRUE)
plot(pM2)
title(main
=
"Generalized
Linear
Model
Raw
Value")
summary(M2)
####
Train/Test
data
####
##get
training/testing
sets
of
data
from
admdata
head(admdata)
samp
=
sample(nrow(admdata),
round(0.75*nrow(admdata)))
traindata=admdata[samp,]
traindata=traindata[traindata[,1]
==
1,
2:7]
testdata=admdata[-‐samp,]
pres
=
admdata[admdata[,1]
==
1,
2:7]
absen
=
admdata[admdata[,1]
==
0,
2:7]
##AUC
could
be
biased
due
to
high
extent
=
high
AUC
values
(Lobo
et
al.
2008,
Jimenez-‐Valverde
2011)
##So,
get
rid
of
"spatial
sorting
bias"
via
"point-‐wise
distance
sampling"
nr=nrow(Pointsxy)
s=sample(nr,0.25*nr)
pres_train=Pointsxy[-‐s,]
pres_test=Pointsxy[s,]
nr=nrow(Absencexy)
s=sample(nr,0.25*nr)
back_train=Absencexy[-‐s,]
back_test=Absencexy[s,]
sb=ssb(pres_test,back_test,pres_train)
sb[,1]/sb[,2]
##Spacial
Sorting
Bias
(SSB)
=
0.326
(If
no
bias
SSB=1)
##Create
subsample
where
SSB
is
removed
i
=
pwdSample(pres_test,back_test,pres_train,
n=1,tr=0.1)
pres_test_pwd=pres_test[!is.na(i[,1]),]
back_test_pwd=back_test[na.omit(as.vector(i)),]](https://image.slidesharecdn.com/4f0642fe-ff34-400e-9d28-12ccf8090a4b-150407130702-conversion-gate01/85/MacholInternship5-53-320.jpg)
![54
sb2=ssb(pres_test_pwd,back_test_pwd,pres_train)
sb2[1]/sb2[2]
##SSB
=
0.999
-‐-‐
Much
Reduced!
###Visualize
train/test
points
r=raster(enviropredictors,1)
plot(!is.na(r),col=c('white','light
grey'),legend=FALSE)
points(back_train,
pch='-‐',
cex=0.5,
col=
'yellow')
points(back_test,
pch='-‐',
cex=0.5,
col=
'black')
points(pres_train,
pch=
'+'
,
col=
'green'
)
points(pres_test,
pch=
'+'
,
col=
'blue'
)
##########
MaxEnt
##############
##Note!
Most
widely
used
SDM
algorythm
(according
to
Hijmans
and
Elith)
##limit
extent:
ext
=
extent(-‐144.6,
-‐113.7,
41.5,
42.3)
##Uses
"jar"
package
-‐
http://www.cs.princeton.edu/~schapire/maxent/
jar
<-‐
paste(system.file(package="dismo"),
"/java/maxent.jar",
sep='')
jar
if
(file.exists(jar)){
xm=maxent(enviropredictors,pres_train,
ext
=
ext)
plot(xm)
}
else
{
cat('cannot
run
this
example
because
maent
is
not
available')
plot(1)
}
response(xm)
e
=
evaluate(pres_test,back_test,xm,enviropredictors)
e
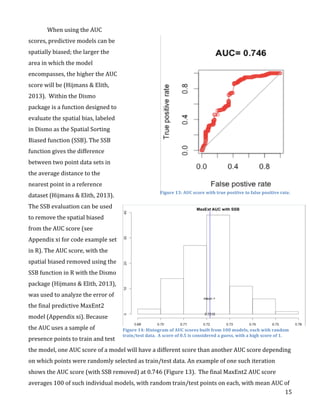
#AUC=0.7712
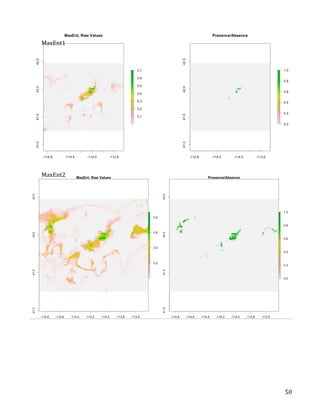
px=predict(enviropredictors,xm,progress='')
par(mfrow=c(1,2))
plot(px,main='MaxEnt,
Raw
Values')
trpx=threshold(e,'spec_sens')
plot(px>trpx,
main='Presence/Absence')
points(pres_train,
pch='+')
##Write
raw
data
as
raster
writeRaster(px,file="RawMaxExt.tif",overwrite=TRUE)
##Write
pres/abs
model
as
raster
MaxExtPresAbs=(px>trpx)
writeRaster(MaxExtPresAbs,file="MaxExtPresAbs.tif",overwrite=TRUE)
####
Max
Ext
Evaluation
####
e=evaluate(testdata[testdata==1,],
testdata[testdata==0,],
xm)
e
plot(e,'ROC')
##AUC
value
=
.827
(guess
@
.5)
##k-‐fold
-‐
do
it
agin
k
times
pres
=
admdata[admdata[,1]
==
1,
2:7]
absen
=
admdata[admdata[,1]
==
0,
2:7]
k
=
100
##I
am
going
to
run
this
10
times
group
=
kfold(pres,
k)
##presence
only,
which
is
good
becuase
the
absence
falls
very
close
##absence
is
supposed
to
be
used
to
test
model](https://image.slidesharecdn.com/4f0642fe-ff34-400e-9d28-12ccf8090a4b-150407130702-conversion-gate01/85/MacholInternship5-54-320.jpg)
![55
group[1:100]
unique(group)
e=list()
for
(i
in
1:k)
{
train=pres[group!=i,]
test=pres[group==i,]
xm=maxent(enviropredictors,pres_train)
e[[i]]=evaluate(p=test,a=absen,xm)
}
e
##AUC
values
for
sensitivity(true
positive
rate)
##
and
specificity
(true
negaitive
rate)
##With
SSB:
evaluate(xm,
p=pres_test,a=back_test,x=enviropredictors)
##AUC
with
=0.719
(0.5
is
guess)
##SSB
removed
evaluate(xm,
p=pres_test_pwd,
a=back_test_pwd,
x=enviropredictors)
##AUC
SSB
reduced
=
0.621
##These
numbers
changed
each
time
you
run
new
model,
so...
##Hist
for
AUC
with
SSB
removed
k
=
100
##I
am
going
to
run
this
100
times
group
=
kfold(pres,
k)
##presence
only,
which
is
good
becuase
the
absence
falls
very
close
##absence
is
supposed
to
be
used
to
test
model
group[1:100]
unique(group)
e=list()
for
(i
in
1:k)
{
j
=
pwdSample(pres_test,back_test,pres_train,
n=1,tr=0.1)
pres_test_pwd=pres_test[!is.na(j[,1]),]
back_test_pwd=back_test[na.omit(as.vector(j)),]
sb2=ssb(pres_test_pwd,back_test_pwd,pres_train)
xm=maxent(enviropredictors,pres_train)
e[[i]]=evaluate(xm,
p=pres_test,a=back_test,x=enviropredictors)
}
e
##AUC
values
for
sensitivity(true
positive
rate)
##
and
specificity
(true
negaitive
rate)
auc
=
sapply(e,function(x){slot(x,'auc')}
)
auc
par(mar=c(1,1,1,0))
par(oma=c(1,1,0.5,0))
par(mfrow=c(1,1))
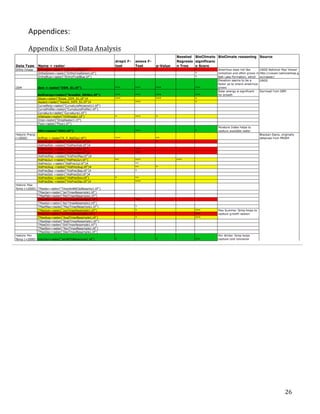
hist(auc,
main
=
'MaxExt
AUC
with
SSB',
sub
=
'mean
=
0.796')
meanauc
=
mean(auc)
#0.745
abline(v
=
meanauc,
col
=
"blue",
lwd
=
2)
text(meanauc,
0
,
round(meanauc,
4))
text(meanauc,
6
,
"mean
=
",
cex
=
1)
TprTnr
=
sapply(e,function(x){
x@t[which.max(x@TPR
+
x@TNR)]
}
)
TprTnr
par(mar=c(1,1,1,0))](https://image.slidesharecdn.com/4f0642fe-ff34-400e-9d28-12ccf8090a4b-150407130702-conversion-gate01/85/MacholInternship5-55-320.jpg)